JAGS: распространение априора внутри дистрибутива
Это наша первая модель:
# Data:
x1 = as.factor(c(0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1))
x2 = as.factor(c(0, 0, 0, 0, 1, 1, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1))
x3 = as.factor(c(0, 1, 1, 0, 0, 0, 1, 1, 0, 1, 1, 0, 0, 0, 1, 1))
x4 = as.factor(c(0, 0, 1, 1, 0, 1, 0, 1, 0, 0, 1, 1, 0, 1, 0, 1))
n = rep(1055, 16)
y = c(239, 31, 15, 11, 7, 5, 18, 100, 262, 32, 38, 32, 8, 7, 16, 234)
# Model:
mymodel = function(){
for (i in 1:16){
y[i] ~ dbin(theta[i], n[i] )
eta[i] <- gamma*x1[i]+beta1*x2[i]+beta2*x3[i]+beta3*x4[i]
theta[i] <- 1/(1+exp(-eta[i]))
}
# Prior
gamma ~ dnorm(0,0.00001)
beta1 ~ dnorm(0,0.00001)
beta2 ~ dnorm(0,0.00001)
beta3 ~ dnorm(0,0.00001)
}
Теперь нас просят добавить альфа как нормальное с известным средним и неизвестной дисперсией. Но дисперсия имеет одинаковый априор, как показано на рисунке:
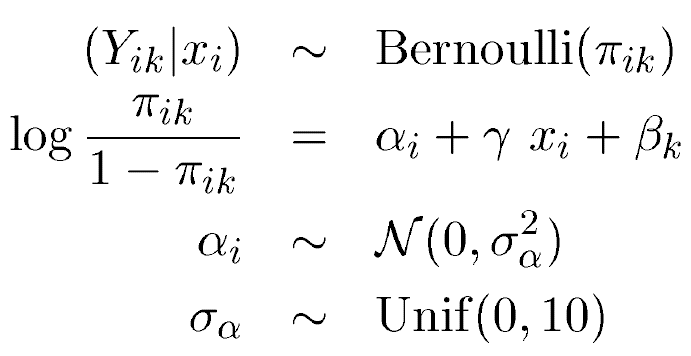
Я не знаю, как добавить альфа в модель, а затем указать новый параметр в приоры...
1 ответ
Вы бы просто добавили alpha в ваш линейный предиктор и дать ему распределение, как и любой другой параметр. Тем не мение, JAGS параметризовывает нормальное распределение как точность вместо дисперсии (точность является обратной величиной дисперсии). Модель будет выглядеть примерно так. Также вы можете просто использовать logit(eta) вместо применения обратного логита.
mymodel = function(){
for (i in 1:16){
y[i] ~ dbin(eta[i], n[i] )
logit(eta[i]) <- alpha + gamma*x1[i]+beta1*x2[i]+beta2*x3[i]+beta3*x4[i]
}
# Prior
alpha ~ dnorm(0, tau_alpha)
tau_alpha <- 1 / var_alpha
var_alpha ~ dunif(0, 10)
gamma ~ dnorm(0,0.00001)
beta1 ~ dnorm(0,0.00001)
beta2 ~ dnorm(0,0.00001)
beta3 ~ dnorm(0,0.00001)
}