Метка и цвет листьев дендрограммы (филогения) в R с использованием пакета обезьяны
После предыдущего поста ( Дендрограмма ярлыка и цветного листа в r) у меня есть дополнительный вопрос.
Мои вопросы похожи на упомянутый пост, но мне интересно, можно ли это сделать с помощью ape (например, plot(as.phylo(fit), type="fan", labelCol) как это имеет больше типа филогении.
Упомянутые вопросы почты были:
Как я могу показать групповые коды в ярлыке листа (вместо номера образца)?
Я хочу назначить цвет для каждой группы кодов и раскрасить листовую метку в соответствии с ней (может случиться так, что они не будут находиться в одном и том же кладе и тем самым я смогу найти больше информации)?
И пример кода:
sample = data.frame(matrix(floor(abs(rnorm(20000)*100)),ncol=200))
groupCodes <- c(rep("A",25), rep("B",25), rep("C",25), rep("D",25))
## make unique rownames (equal rownames are not allowed)
rownames(sample) <- make.unique(groupCodes)
colorCodes <- c(A="red", B="green", C="blue", D="yellow")
## perform clustering
distSamples <- dist(sample)
hc <- hclust(distSamples)
## function to set label color
labelCol <- function(x) {
if (is.leaf(x)) {
## fetch label
label <- attr(x, "label")
code <- substr(label, 1, 1)
## use the following line to reset the label to one letter code
# attr(x, "label") <- code
attr(x, "nodePar") <- list(lab.col=colorCodes[code])
}
return(x)
}
## apply labelCol on all nodes of the dendrogram
d <- dendrapply(as.dendrogram(hc), labelCol)
plot(d)
2 ответа
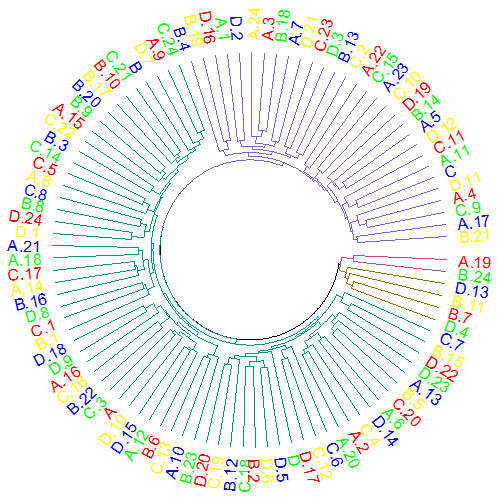
Посмотри на ?"plot.phylo":
library("ape")
plot(as.phylo(hc), tip.color=colorCodes[substr(rownames(sample), 1, 1)], type="fan")
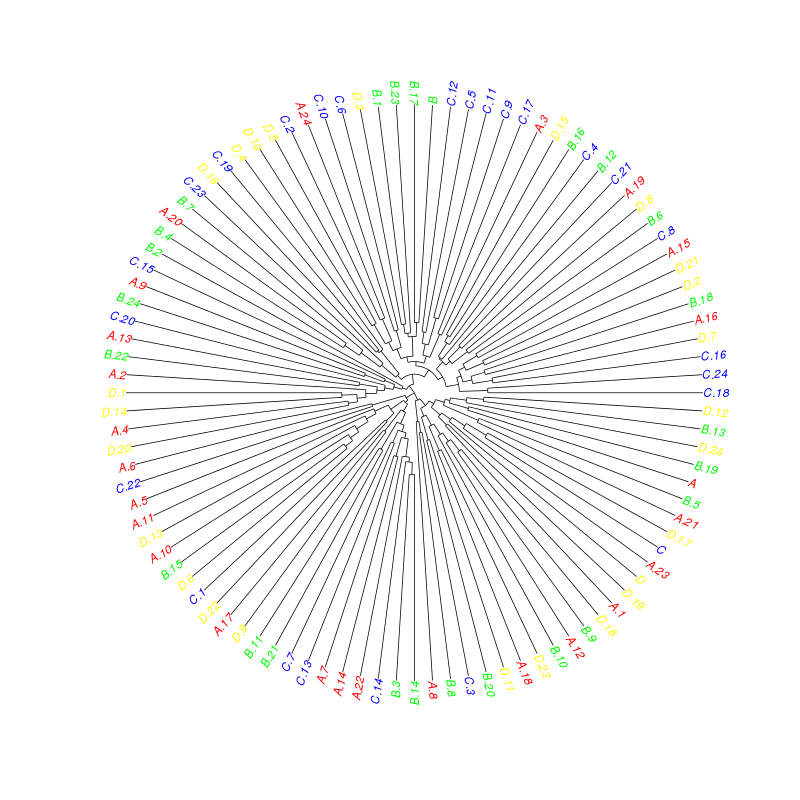
Еще одно решение вопроса заключается в использовании нового circlize_dendrogram функция, которая объединила два пакета: circlize и dendextend. Сначала вам нужно будет установить их:
install.packages("circlize")
devtools::install_github('talgalili/dendextend')
Вот код для запуска:
# YOUR CODE
sample = data.frame(matrix(floor(abs(rnorm(20000)*100)),ncol=200))
groupCodes <- c(rep("A",25), rep("B",25), rep("C",25), rep("D",25))
## make unique rownames (equal rownames are not allowed)
rownames(sample) <- make.unique(groupCodes)
colorCodes <- c(A="red", B="green", C="blue", D="yellow")
## perform clustering
distSamples <- dist(sample)
hc <- hclust(distSamples)
#--------------
# NEW CODE
dend <- as.dendrogram(hc )
library(dendextend)
labels_colors(dend) <- colorCodes
# plot(dend)
dend <- color_branches(dend, k=4)
# plot the radial plot
par(mar = rep(0,4))
# circlize_dendrogram(dend, dend_track_height = 0.8)
circlize_dendrogram(dend)
И вот результирующий сюжет: