Визуализируйте файл BAM с несколькими геномами
Я хочу визуализировать BAM-файл с несколькими геномами. Я отсортировал его с помощью samtools и тоже получил индексный файл.
Сначала я попробовал Qualimap, но когда было произведено покрытие по эталону, это не получилось, так как значения перекрывались к концу осей.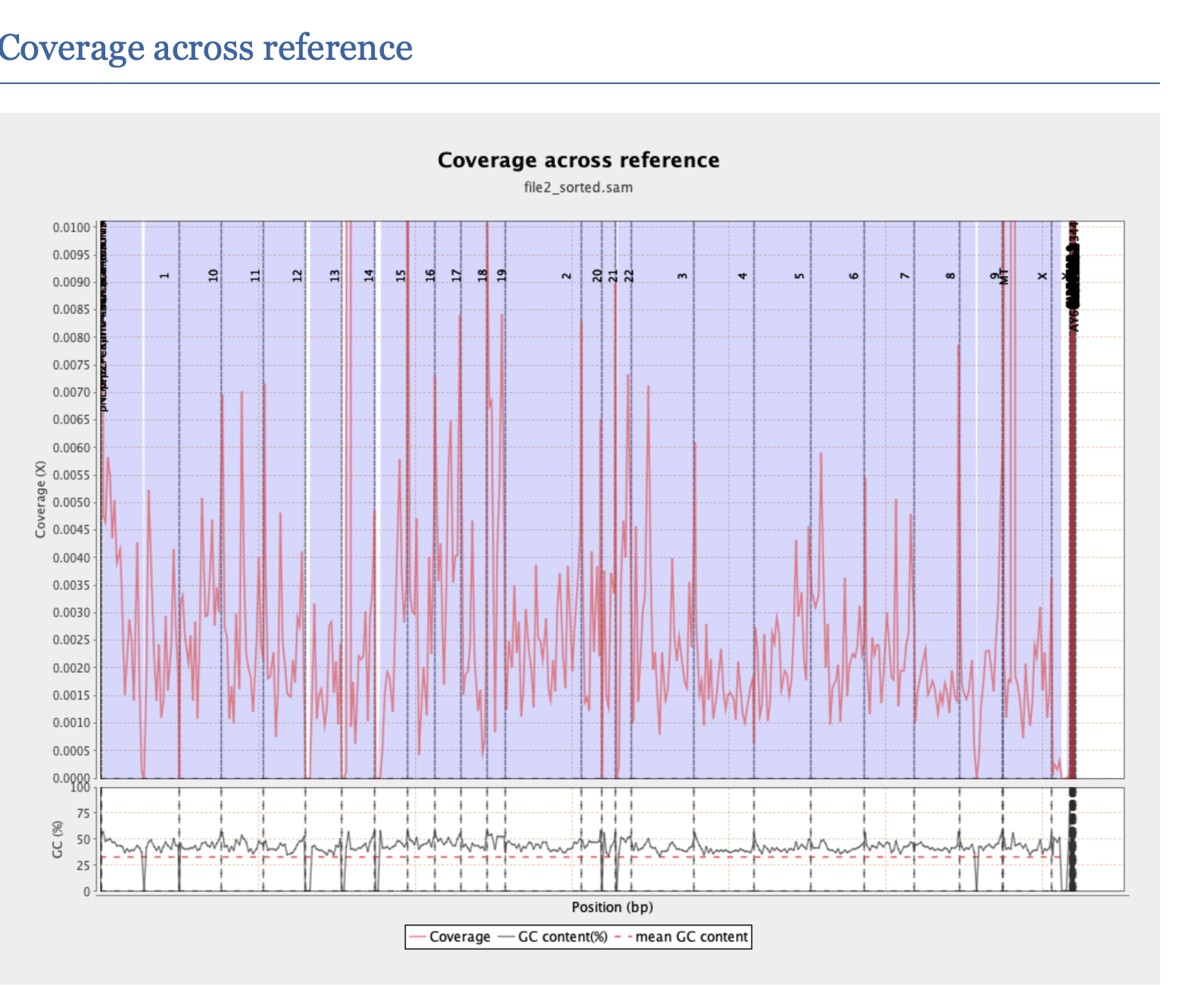
Мне нужно улучшить визуализацию. Я использовал различные инструменты, IGV, GenomeWorkbench и даже ngscat, но многие из этих инструментов не принимают мой файл BAM, поскольку он имеет несколько / несколько типов генома (вирусный и человеческий). Мне нужен инструмент, который примет это и даст мне более четкий график.
Спасибо!