Как раскрасить метки дендрограммы в соответствии с определенными группами? (в R)
У меня есть числовая матрица в R с 24 строками и 10000 столбцов. Имена строк этой матрицы в основном являются именами файлов, из которых я прочитал данные, соответствующие каждой из 24 строк. Помимо этого у меня есть отдельный список факторов с 24-ю началами, определяющий группу, к которой принадлежат 24 файла. Есть 3 группы - Спирты, Углеводород и Эстер. Имена и соответствующая группа, к которой они принадлежат, выглядят так:
> MS.mz
[1] "int-354.19" "int-361.35" "int-368.35" "int-396.38" "int-408.41" "int-410.43" "int-422.43"
[8] "int-424.42" "int-436.44" "int-438.46" "int-452.00" "int-480.48" "int-648.64" "int-312.14"
[15] "int-676.68" "int-690.62" "int-704.75" "int-312.29" "int-326.09" "int-326.18" "int-326.31"
[22] "int-340.21" "int-340.32" "int-352.35"
> MS.groups
[1] Alcohol Alcohol Alcohol Alcohol Hydrocarbon Alcohol Hydrocarbon Alcohol
[9] Hydrocarbon Alcohol Alcohol Alcohol Ester Alcohol Ester Ester
[17] Ester Alcohol Alcohol Alcohol Alcohol Alcohol Alcohol Hydrocarbon
Levels: Alcohol Ester Hydrocarbon
Я хотел создать дендрограмму, чтобы посмотреть, как данные в матрице могут быть кластеризованы. Итак, я использовал следующие команды:
require(vegan)
dist.mat<-vegdist(MS.data.scaled.transposed,method="euclidean")
clust.res<-hclust(dist.mat)
plot(clust.res)
и я получил дендограмму. Теперь я хочу раскрасить имена файлов в дендрограмме в соответствии с группой, к которой они принадлежат, т.е. Алкоголь, Углеводород или Эстер. Я посмотрел на различные примеры, размещенные на форуме, как
Метка и цвет листьев дендрограммы в г
Метка и цвет листьев дендрограммы в R, используя пакет обезьяны
Кластеризация с начальной загрузкой
, но не смог реализовать это для моих данных. Я не уверен, как соотносить row.names с MS.groups, чтобы получить цветные имена в дендрограмме.
При создании дерева с использованием dendextend (как описано в https://nycdatascience.com/wp-content/uploads/2013/09/dendextend-tutorial.pdf), я получаю следующее дерево
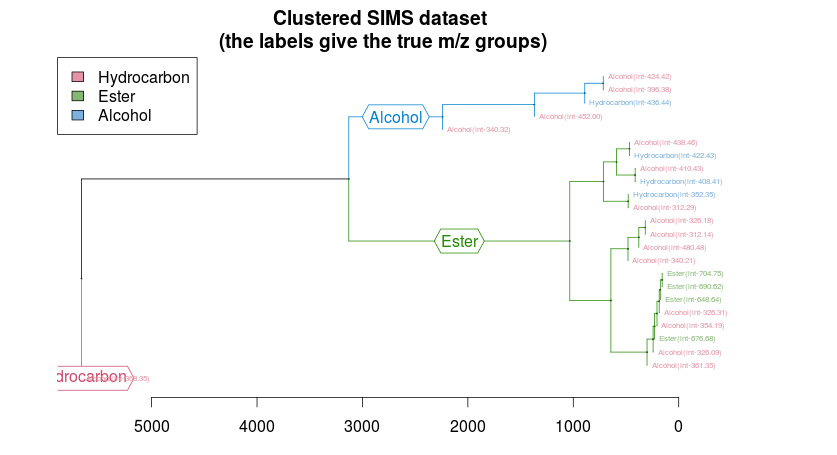
Вот код, используемый для его генерации:
require(colorspace)
d_SIMS <- dist(firstpointsample5[,-1])
hc_SIMS <- hclust(d_SIMS)
labels(hc_SIMS)
dend_SIMS <- as.dendrogram(hc_SIMS)
SIMS_groups <- rev(levels(firstpointsample5[, 1]))
dend_SIMS <- color_branches(dend_SIMS, k = 3, groupLabels = SIMS_groups)
is.character(labels(dend_SIMS))
plot(dend_SIMS)
labels_colors(dend_SIMS) <- rainbow_hcl(3)[sort_levels_values(as.numeric(firstpointsample5[,1])[order.dendrogram(dend_SIMS)])]
labels(dend_SIMS) <- paste(as.character(firstpointsample5[, 1])[order.dendrogram(dend_SIMS)],"(", labels(dend_SIMS), ")", sep = "")
dend_SIMS <- hang.dendrogram(dend_SIMS, hang_height = 0.1)
dend_SIMS <- assign_values_to_leaves_nodePar(dend_SIMS, 0.5,"lab.cex")
par(mar = c(3, 3, 3, 7))
plot(dend_SIMS, main = "Clustered SIMS dataset\n (the labels give the true m/z groups)",horiz = TRUE, nodePar = list(cex = 0.007))
legend("topleft", legend = SIMS_groups, fill = rainbow_hcl(3))
3 ответа
Я подозреваю, что функция, которую вы ищете, это либо color_labels или же get_leaves_branches_col, Первый цвет ваших ярлыков основан на cutree (лайк color_branches do), а второй позволяет получить цвета веток каждого листа, а затем использовать его для окрашивания меток дерева (если вы используете необычные методы для окрашивания ветвей (как это происходит при использовании branches_attr_by_labels). Например:
# define dendrogram object to play with:
hc <- hclust(dist(USArrests[1:5,]), "ave")
dend <- as.dendrogram(hc)
library(dendextend)
par(mfrow = c(1,2), mar = c(5,2,1,0))
dend <- dend %>%
color_branches(k = 3) %>%
set("branches_lwd", c(2,1,2)) %>%
set("branches_lty", c(1,2,1))
plot(dend)
dend <- color_labels(dend, k = 3)
# The same as:
# labels_colors(dend) <- get_leaves_branches_col(dend)
plot(dend)
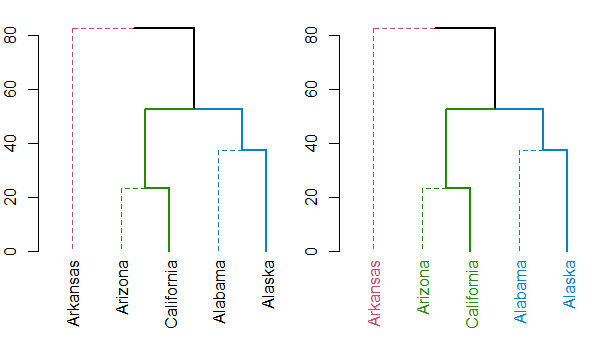
В любом случае, вы всегда должны смотреть на set функция, для идей о том, что можно сделать с вашей дендрограммой (это избавит вас от необходимости запоминать названия различных функций).
Вы можете взглянуть на этот учебник, который отображает несколько решений для визуализации дендограмм в R по группам
https://rstudio-pubs-static.s3.amazonaws.com/1876_df0bf890dd54461f98719b461d987c3d.html
Тем не менее, я думаю, что лучшее решение, подходящее для ваших данных, предлагается пакетом dendextend. Смотрите руководство (пример, касающийся набора данных 'iris', который похож на вашу проблему): https://nycdatascience.com/wp-content/uploads/2013/09/dendextend-tutorial.pdf
Смотрите также виньетка: http://cran.r-project.org/web/packages/dendextend/vignettes/Cluster_Analysis.html
Вы можете попробовать это решение, измените "labs" только с вашими "MS.groups" и "var" с вашими "MS.groups", преобразованными в числовые (возможно, с as.numeric). Это происходит из того, как окрасить метки дендрограммы дополнительной факторной переменной в R
## The data
df <- structure(list(labs = c("a1", "a2", "a3", "a4", "a5", "a6", "a7",
"a8", "b1", "b2", "b3", "b4", "b5", "b6", "b7"), var = c(1L, 1L, 2L,
1L,2L, 2L, 1L, 2L, 1L, 2L, 1L, 1L, 2L, 2L, 2L), td = c(13.1, 14.5, 16.7,
12.9, 14.9, 15.6, 13.4, 15.3, 12.8, 14.5, 14.7, 13.1, 14.9, 15.6, 14.6),
fd = c(2L, 3L, 3L, 1L, 2L, 3L, 2L, 3L, 2L, 4L, 2L, 1L, 4L, 3L, 3L)),
.Names = c("labs", "var", "td", "fd"), class = "data.frame", row.names =
c(NA, -15L))
## Subset for clustering
df.nw = df[,3:4]
# Assign the labs column to a vector
labs = df$labs
d = dist(as.matrix(df.nw)) # find distance matrix
hc = hclust(d, method="complete") # apply hierarchical clustering
## plot the dendrogram
plot(hc, hang=-0.01, cex=0.6, labels=labs, xlab="")
## convert hclust to dendrogram
hcd = as.dendrogram(hc)
## plot using dendrogram object
plot(hcd, cex=0.6)
Var = df$var # factor variable for colours
varCol = gsub("1","red",Var) # convert numbers to colours
varCol = gsub("2","blue",varCol)
# colour-code dendrogram branches by a factor
# ... your code
colLab <- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
attr(n, "label") <- labs[a$label]
attr(n, "nodePar") <- c(a$nodePar, lab.col = varCol[a$label])
}
n
}
## Coloured plot
plot(dendrapply(hcd, colLab))