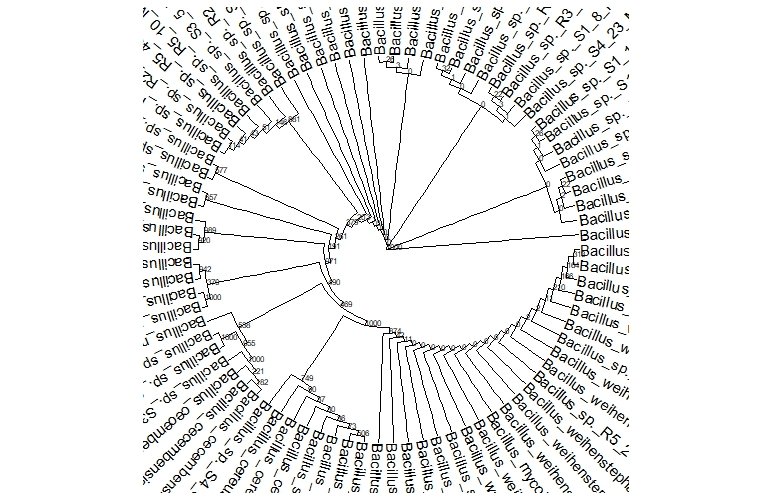
Недостаточно большая площадь графика
Я пытаюсь построить круговое филогенетическое дерево с узлами, помеченными начальной загрузкой, и определенными пользователем / цветными метками кончиков. Я заставил результаты начальной загрузки и метки работать должным образом, но почему-то я просто не мог полностью нарисовать график в своем графическом устройстве. Всегда были метки (которые исходили от центра круглого филологического дерева), отсутствующие части их самих. Единственный способ состоял в том, чтобы установить размер этикетки наконечника очень очень маленьким.
Файл DNAbin, на котором основаны матрица расстояний и бутстрап, состоит из 82 штаммов микробных данных (812aa). Ярлык подсказки был окрашен на основе отдельного файла атрибутов.
dna_b<-read.dna("filename.txt", format="fasta")
dist.b<-dist.dna(dna_b)
tree_b<-nj(dist.b)
bp_b<-boot.phylo(tree_b, dna_b, B=1000, function(xx) nj(dist.dna<xx)))
tree_b2<-apeBoot(tree_b, bp_b)
phylo_b<-ggtree(tree_b2, layout='circular', size=0.02, branch.length = "none")+geom_text2(aes(label=bootstrap), size = 2)+geom_tiplab(aes(angle=angle))
#I changed the tiplab to original so using user defined attribute data was not required (the next line).
#phylo_b<-phylo_b %<+% isolate_attr +geom_tiplab(aes(color=tr_level,angle=angle),size=1)
phylo_b+theme(legend.position = "right", legend.text = element_text(size=3))+guides(colour = guide_legend(override.aes = list(size=3)))
Я попытался установить новые графические устройства с большей шириной и высотой, но безрезультатно. Регулировка маржи тоже не сработала. Установив размер наконечника меньше 0,05, я мог бы получить полный график, но это действительно не оптимально. Мог бы действительно ценить некоторую помощь здесь.
Информация о сессии:
R version 3.3.3 (2017-03-06)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows >= 8 x64 (build 9200)
locale:
[1] LC_COLLATE=Chinese (Simplified)_China.936
[2] LC_CTYPE=Chinese (Simplified)_China.936
[3] LC_MONETARY=Chinese (Simplified)_China.936
[4] LC_NUMERIC=C
[5] LC_TIME=Chinese (Simplified)_China.936
attached base packages:
[1] parallel stats4 stats graphics grDevices utils datasets
[8] methods base
other attached packages:
[1] ggtree_1.6.11 ggplot2_2.2.1 S4Vectors_0.12.2
[4] BiocGenerics_0.20.0
loaded via a namespace (and not attached):
[1] Rcpp_0.12.11 lattice_0.20-35 ape_4.1 tidyr_0.6.3
[5] digest_0.6.12 grid_3.3.3 plyr_1.8.4 jsonlite_1.5
[9] nlme_3.1-131 gtable_0.2.0 magrittr_1.5 scales_0.4.1
[13] rlang_0.1.1 lazyeval_0.2.0 labeling_0.3 tools_3.3.3
[17] munsell_0.4.3 colorspace_1.3-2 tibble_1.3.3
Rstudio версия 1.0.136
Пересмотрено: Как указывалось ранее, данные должны были быть предоставлены для воспроизведения проблемы. Я прилагаю часть данных здесь, полные данные слишком велики. Я попытался использовать команду stringi для случайной генерации последовательностей днк, но случайные последовательности часто приводят к появлению NaN в файле dist. Следующие данные были непосредственно скопированы из txt-файла, указанного в первой строке кода, с отредактированной частью имени ~
Данные Фаста (б):
>Bacillus_somenameherethatsverylone_R2_1_anothernumber
AACTACGCTCGCGTATCGGATTATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACCTTTATGGAGCCAGCCGCCTAAGTGGGACAGATGATTGGGGTGAAGTCGTACAAGGTACCCAAA
>Bacillus_sp._R2_8_anothernumber
TTACTATGATAGCGTGTCGGATTATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCTTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCACACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGCAGGCTGCAACTCGCCTGCATGAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGTGAATACGTTCCCGGGCCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAGCACCCGAAGTCGTGACGTAACCTTCATGGAGCCAGTCAGCCTATGTGGGACAGATGATTGGGGTGAGTCGTACCAAAGTAAACACA
>Bacillus_sp._R2_9_anothernumber
GTTACCCCTGACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCTTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGCAGGCTGCAACTCGCCTGCATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGTCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTCATGTAGTCAGCCGCCTATGTGGGACAGATGATTGGGGTGAGTCGTACCAAGGAAAGCCAGC
>Bacillus_someotherlongname_R2_21_anothernumber
TTACTTTTCTACGTATCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACCTTTATGGAGCCAGCCGCCTATGTGGGACAGATGATTGGGGTGAAGTCGTACCAAGGTAGCCCATA
>Bacillus_sp._R2_42_anothernumber
ATTCTTTCTCGCGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCTTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAAGTGGGACAGATGATTGGGGTGAAGTCGTACCACAGGTTAACCA
>Bacillus_sp._R2_75_anothernumber
TCTTCAACCGCGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCGAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTTTTGCCCTCCCTAGAGATAGGGACTTCCCTTCGGGGACAAAAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTTAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGAGCTGCAAACCCGCGAGGGTAAGCGAATCTCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGTGGATCAGCATGCCACGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCGCAAGGAGCCAGCCGCCTAAAGTGGGACAGATGATTGGGGTGAGTCGACACAGGGGAAACCCA
>Bacillus_sp._R2_106_anothernumber
GCGGATATGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTTAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGGATGGTACAAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCCGGGCCTTGTACACACCCGCCCCGTCACACCACGAGAAGTTTGTAACACCCGAAAGTCGGGTGAGGTAACCCTTTTATGG
>Bacillus_somelongnameagain_R2_115_anothernumber
GTCGAATTATGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGTCTTGTACAT
>Bacillus_sp._R2_116_anothernumber
AAAACAGTGTTTGCTTTTTATTCAGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACCCTAGAGATAGGGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACTTTATGGAGCCAGCCGCCTAAGTGGGACAGATGATTGGGGTGAAGTCGTACCAAGGTAAGCCAT
>Bacillus_sp._R3_1_anothernumber
GTAGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAAACTCGCCTACATGAAAGCCGGAAATCGCTAGTTATTCGCGGATCAGCATGCCGCGGTTGAATACGTTCCCCGGGCCTTGTAACACACCCGCCCCGTCACACCACGAGAGTTTTGTAACACCCGAAAGTCGGTGGAGGGTAACCCTTTTTATTG
>Bacillus_cereus_R3_2_anothernumber
GTAAGCATTTGGATGCATCTGGAATCGGTATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACCCTAGAGATAGGGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACCTTTTATGGAGCCAGCCGCCTAAAGTGGGACAGATGATTGGGGGTGAAGTCGTAACAAGGGTAGCC
>Bacillus_sp._R3_19_anothernumber
AACGGTTCAGCCACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGTGGGACAGATGATTGGGGTGAAGTCGTACAACAGGAGGTAACA
>Bacillus_sp._R3_20_anothernumber
GGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAAATAACGTTCCCGGGGCCTTGTACACACCGCCCCGTCACACCACGAGAGTTTGTAAACAACCCGAAAGTCGGGTGAGGTAACCCTTTATGGGAGCCAGCCGCCTTAAAGGTGGAACAGATGAATTGGGGTGAAAGTCGTAACAAGGGTAAGCCAA
>Bacillus_sp._R3_22_anothernumber
TCGGTACACTCACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGGGGGGACAGATGATTGGGGTGAAGTCGACAACAAAGGGTATAACA
>Bacillus_sp._R3_74_anothernumber
AGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGACCTTGTACACACCGCTCCGTCACACCACGAGAGTTTTGTTACTACCCGAAAGTCGGTGGGGGTAACCTTTATGGGAGTCAGGCCGCCTTAAGGTGGGACAGAATGATTGGGGGGTGAAAGTTCGTTAACCAAGGGTTAGCCAAATT
>Bacillus_sp._R3_76_anothernumber
AACACGTGACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGTGGACAGATGATGGGGTGAGCTAC
>Bacillus_sp._R3_89_anothernumber
TCGTCTACCGCACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGGTGGGACAGATGATTGGGGTGAAGTCGACACA
>Bacillus_sp._R4_18_anothernumber
GGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTTCGGATTGTGGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGAGACCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTACACCCGAAGTCGGTGGAGTAACCCTTTATGGAGTCCAGCGCCTAAGTGGGACAGATGATTGGGGGTGAAGTCGTACCAGTACCCATCT
>Bacillus_someotherlongname_R4_23_anothernumber
AGGTACAGTAGACGTATCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACTTTATGGAGCAGCCGCCTAAGGTGGGACAGATGATTGGGGTGAAGTCGTAACAAGGTAGCCA
>Bacillus_sp._R4_38_anothernumber
AAACTCTTTGTTTACTTATACCACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCACGTGTAGCGGTGAAATGCGTAGAGATGTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCGAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTTTTGCCCTCCCTAGAGATAGGGACTTCCCTTCGGGGACAAAAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTTAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGAGCTGCGAACCCGCGAGGGTAAGCGAATCTCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCGCAAGGAGTCAGCCGCCTAAGTGGGACAGATGATTGGGGTGAAGTCGTACACAAGTAAAACT
>Bacillus_somelongname_R5_1_anothernumber
CGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACCCTAGAGATAGGGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGGAACGCTAGTAATCGCGGATCACATGCCGCGGGGAATACGTTCCCGGGCCTTGTACACACCCGCCCGTCACACCACGAGAGTTTTGTTACACCCGAAATTCGGTGGGGTAACCTTCATGGGAGCCAGCCCGCCTAAGGGGGGACAGAATGATTGGGTGAAGTC
>Bacillus_sp._R5_6_anothernumber
TGAATAACAAACGACGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCTCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGGTGGGACAGATGATTGGGGTGAAGTCGTACAC
>Bacillus_sp._R5_8_anothernumber
TTAGGATATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCGTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAGCCATTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGA
>Bacillus_sp._R5_10_anothernumber
TTACTTATGTGACGTGTCGGATTATTGGGCGTAAGCGCGCGCAGGTGGTTCCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGGAACTTGAGTGCAGAAGAGGAAAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGACAACCCTAGAGATAGGGCTTTCCCCTTCGGGGGACAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCAGCATTCAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGATGGTACAAAGGGCTGCAAACCTGCGAAGGTAAGCGAATCCCATAAGCCATTCTCAGTTCGGATTGCAGGCTGCAACTCGCCTGCATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGAGGTAACCTTTATGGAGCCAGCCGCCTAAGGGGGGACAGATGATTGGGGTGAAGTCGTACACAAGGTAAACTT
>Bacillus_somelongnamehere_R5_11_anothernumber
ATACTAACTTCTCACGTACGGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAGACTTGAGTGCAGAAGAGGAAAGTGGAATTCCATGTGTAGCGGTGAAATGCGTAGAGATATGGAGGAACACCAGTGGCGAAGGCGACTTTCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTAGAGGGTTTCCGCCCTTTAGTGCTGAAGTTAACGCATTAAGCACTCCGCCTGGGGAGTACGGCCGCAAGGCTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCTCTGAAAACTCTAGAGATAGAGCTTCTCCTTCGGGAGCAGAGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTGATCTTAGTTGCCATCATTAAGTTGGGCACTCTAAGGTGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAAGAGCTGCAAGACCGCGAGGTGGAGCTAATCTCATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCTGGAATCGCTAGTAATCGCGGATCATCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACCTTTATGGAGCCAGCCGCCTAAGTGGGACAGATGATTGGGGTGAAGTCGTACAAGGGTAGCCATAAC
>Bacillus_longnamehere_R5_12_anothernumber
TACTTTCCCGCGTGTCGGATATTGGGCGTAAGCGCGCGCAGGTGGTTTCTTAAGTCTGATGTGAAAGCCCACGGCTCAACCGTGGAGGGTCATTGGAAACTGGGAAACTTGAGTGCAGAAGAGGATAGTGGAATTCCAAGTGTAGCGGTGAAATGCGTAGAGATTTGGAGGAACACCAGTGGCGAAGGCGACTATCTGGTCTGTAACTGACACTGAGGCGCGAAAGCGTGGGGAGCAAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGATGAGTGCTAAGTGTTGGGGGGTTTCCGCCCCTCAGTGCTGCAGCTAACGCATTAAGCACTCCGCCTGGGGAGTACGGTCGCAAGACTGAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGTGGAGCATGTGGTTTAATTCGAAGCAACGCGAAGAACCTTACCAGGTCTTGACATCCCATTGACCACTGTAGAGATACAGTTTTCCCTTCGGGGACAACGGTGACAGGTGGTGCATGGTTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTATTCTTAGTTGCCATCATTTAGTTGGGCACTCTAAGGAGACTGCCGGTGACAAACCGGAGGAAGGTGGGGATGACGTCAAATCATCATGCCCCTTATGACCTGGGCTACACACGTGCTACAATGGACGGTACAAACGGTTGCCAACCCGCGAGGGGGAGCTAATCCGATAAAACCGTTCTCAGTTCGGATTGTAGGCTGCAACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACACCACGAGAGTTTGTAACACCCGAAGTCGGTGGGGTAACCTTTATGGAGCCAGCCGCCGAAGTGGGATAGATGATTGGGGTGAAGTCGTACCAAGGTACTAA
4 ответа
Как отметил @Axeman в своем комментарии, ggsave Вы можете настроить размеры. Вы можете комбинировать это с корректировкой размера этикетки.
library(ggtree)
library(ape)
# Generate a random tree
set.seed(2018)
my_labels <- paste(paste(LETTERS, collapse = ""), 1:20) # generate long labels
tree <- ape::rcoal(n = 20, tip.label = my_labels)
# plot the tree
ggtree(tr = tree, layout = 'circular') +
geom_tiplab(size = 2, aes(angle = angle))
# Save as PDF and force a 'huge' size plot
ggsave("assets/test.pdf", width = 50, height = 50, units = "cm", limitsize = FALSE)
Вы пытались вручную настроить с помощью xlim(xmin, xmax)?
Пример:
tree <- rtree(10)
ggtree(tree) + xlim(-10, 10)
У меня тоже была эта проблема, и я использовал как ggsave() ответ предоставили Валентин и xlim() ответ предоставлен RaphiS. ggsave() будет работать, но мы можем не захотеть делать такой огромный сюжет, и в этом случае xlim() было бы предпочтительнее.
Я хотел бы отметить, что людям нужно поиграть с xlim()значения, а не просто попробуйте (-10,10) или (0,0.06) (с https://support.bioconductor.org/p/72398/) и откажитесь, если они получат ошибку. Вxlim()необходимые значения также зависят от вашего дерева и его структуры. Для справки: для моего дерева с 261 узлом для обычного вертикального дерева макета с длинами ветвей я использовалxlim(0,1), и для того же дерева с layout = "circular" а также branch.length = "none" я использовал xlim(0,36). Вы также можете играть сplot.margin() В паре с xlim().
Отрегулируйте размер графика вручную с помощью
+ xlim(NA, 24)и отрегулируйте размер по своему усмотрению
или же
+ theme(plot.margin = unit(c(14,8,14,8), "mm"))
Отрегулируйте шрифт в маркировке наконечника с помощью
+ geom_tiplab(size =3)и отрегулируйте размер, как вы хотите.
например
tree2 <- tree1+ xlim(NA, 24)
#or
tree2 <- tree1 + theme(plot.margin = unit(c(14,8,14,8), "mm"))
#In this case 14mm, 8mm, 14mm and 8mm can be your starting settings. By either increasing or reducing individual settings of margins as you like till you get an optimised plot.
tree2 <- tree2 + theme(plot.margin = unit(c(10,8,10,8), "mm"))
tree2 <- tree2 + theme(plot.margin = unit(c(20,10,20,10), "mm"))
tree2 <- tree2 + geom_tiplab(size =3)#to choose your preferred font size of the tip labels
С наилучшими пожеланиями,
Эванс