Сезонное разложение по неоднородным разнесенным временным рядам, какой-нибудь устоявшийся алгоритм в R или Python?
Пакет stats в R имеет stl(), но для него требуется равномерно распределенный временной ряд, созданный функцией ts(). Это не может иметь дело с объектами зоопарка.
Как ни странно, он также не может справиться с пропущенными значениями, хотя метод STL утверждает, что может заполнить пропущенное значение с помощью LOESS. (см. этот вопрос в резюме.)
Так, например, если у вас есть данные рабочего дня, вы не можете просто сделать это календарным днем, поставив NA на выходные и вызвав stl().
Я также вижу автора Python statsmodel, пытающегося перенести stl () для работы с Pandas TimeSeries, но, похоже, его пока нет.
Спасибо
Изменить: просто чтобы добавить, что я знаю, что я могу просто сделать очень простую модель, например, подгонку гармоник, но я хочу, чтобы хорошо зарекомендовавшая себя модель, по крайней мере, обеспечивала эталонный тест. У меня есть субмесячные данные, поэтому X12 не применяется.
1 ответ
Согласно @Julius в этом посте можно ли использовать stl с na.approx, от zoo пакет, используя stl(x, na.action = na.approx, ...), Это делает какую-то интерполяцию.
к несчастью stl предпочитает регулярные временные ряды.
Поэтому я использую подход @Julius, чтобы проверить, насколько хорошо loess выполняет в заполнении NAs предполагая нерегулярный временной ряд. Видимо ошибка очень маленькая.
Позволяет моделировать
Следующая функция берет данные и моделирует сценарии с 3^(0:p) НС с naapprox=TRUE для использования na.approx а также optspan=TRUE оптимизировать параметр диапазона. Функция потерь - КАРТА.
#The approach is based in @Julius stl with NAs
library(zoo)
library(plyr)
library(reshape)
library(ggplot2)
mape <- function(f, x) colMeans(abs(1 - f / x) * 100,na.rm=T)
loessCheck <- function(data,p=2, naapprox=TRUE, optspan=TRUE){
set.seed(20130201)
pos <- lapply(3^(0:p), function(x) sample(1:length(data), x))
datasetsNA <- lapply(pos, function(x) {data[x] <- NA; data})
original <- data.frame(y.predict=as.numeric(data))
original$id <- "Original"
datasetsNA <- lapply(datasetsNA, function(y){
posna=which(is.na(y))
tvo=1:length(y)
yo=y;tv=tvo
if (any(posna%in%c(1,length(y)))) tv=tvo[-posna[which(posna%in%c(1,length(y)))]]
if(naapprox) y=na.approx(y) #instead of inside loess
if(optspan){ fseq=function(x, y)
mape(matrix(predict(loess(y ~ dt, span=x, data.frame(dt=tv, y=y))),ncol=1),as.vector(y[!is.na(y)]))
ospan <- optimize(fseq, c(0.1,1), maximum=FALSE,y=y)
spanmim <- ospan$minimum
} else spanmim <- 0.75
y.loess <- loess(y ~ dt, span=spanmim, data.frame(dt=tv, y=y))
y.predict <- predict(y.loess, data.frame(dt=tvo))
y.predict[-posna] <- yo[-posna]
data.frame(y.predict,
id = paste(length(posna), "NAs"),
stringsAsFactors = FALSE)
})
loessAll <- rbind.fill(c(list(original), datasetsNA))
loessAll$Date <- time(data)
results <- data.frame(y.predict = sapply(lapply(datasetsNA, '[', i = "y.predict"), mape, original[, "y.predict"]))
results$id <- unique(loessAll$id[-(1:nrow(original))])
results <- melt(results, id.var = "id")
results$x <- min(loessAll$Date) + diff(range(loessAll$Date)) / 4
results$y <- min(original[, "y.predict"],na.rm=T) + diff(range(original[, "y.predict"],na.rm=T)) / (4 * p) * (0:p)
results$value <- round(results$value, 2)
ggplot(loessAll, aes(x = Date, y = y.predict, colour = id, group = id)) + geom_line() +
theme_bw() +
theme(legend.title = element_blank(), strip.background = element_rect(fill = "white")) +
labs(x = NULL, y = NULL) + scale_colour_brewer(palette = "Set1") +
lapply(unique(results$id), function(z)
geom_text(data = results, colour = "black", size = 3,
aes(x = x, y = y, label = paste0("MAPE (", id, "): ", value, "%"))))
}
args(loessCheck) # first boolean for using na.approx, the second for optimize span parameter in sample
loessCheck(nottem,p=3) #T T
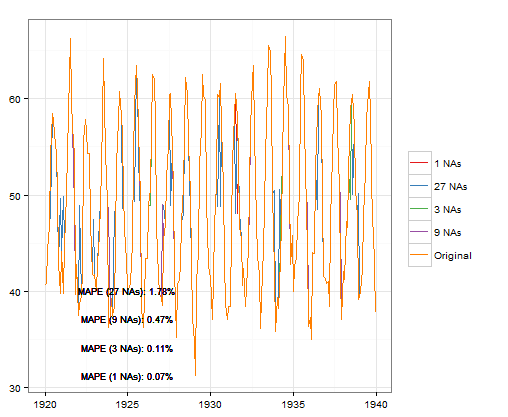
loessCheck(nottem,p=3,FALSE) #FT
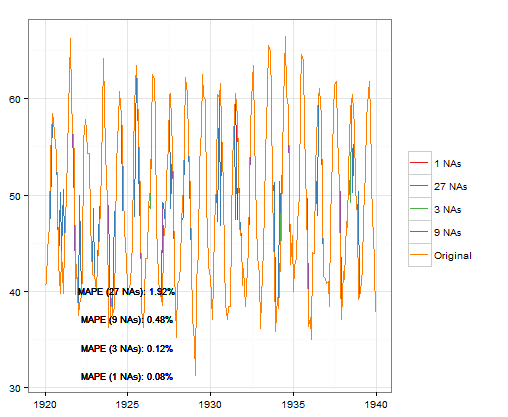
loessCheck(nottem,p=3,FALSE,FALSE)
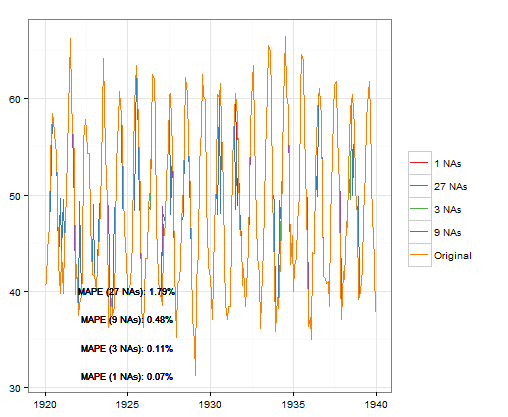 loessCheck (nottem, р =3, значение TRUE,FALSE)
loessCheck (nottem, р =3, значение TRUE,FALSE)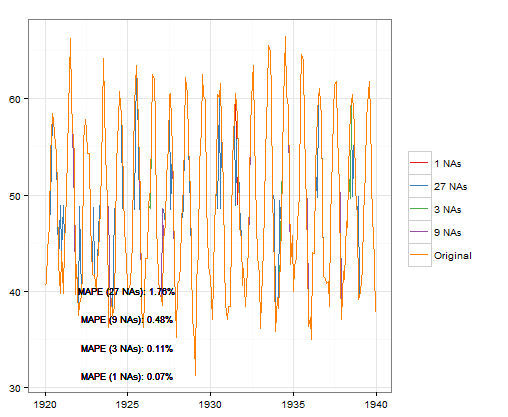
Меньше КАРТЫ при использовании na.approx из пакета зоопарка и рекомендуется span= 0.75, После заполнения НС могут быть рассмотрены другие варианты моделирования.