Пометьте и раскрасьте каждое название образца другим цветом с помощью Deprogram в R
Может кто-то помочь мне, пожалуйста?
Я пытаюсь нарисовать дендрограмму, используя матрицу расстояний ДНК, используя следующий код. Кажется, все в порядке, но мне кажется, что у каждого экземпляра нет другого цвета; Файл моей матрицы расстояний находится по следующей ссылке:
https://gist.github.com/plxsas/f02cd17e804f9fe1fe4a
T6 <- fasta2DNAbin(file="T6.fas", quiet=FALSE, snpOnly=FALSE)
dis <- dist.dna(T6, as.matrix = TRUE)
dis2 <- matrix(dis, nr=70,nc=70)
groupCodes <- c(rep("1A_53",6), rep("1A_56",5), rep("1A_57",6), rep("1A_59",6), rep("1A_63",5),
rep("1A_64",6), rep("1A_69",6), rep("1A_70",6), rep("1A_71",6),rep("1A_72",5),
rep("5A_15",6), rep("5A_32",7))
rownames(dis2) <- make.unique(groupCodes)
colorCodes <- c(1A_53="red", 1A_56="green", 1A_57="blue", 1A_59="yellow", 1A_63="darkgoldenrod1",
1A_64="burlywood3",1A_69="darkgray",1A_70="darkolivegreen",1A_71="darkorchid4",1A_72="darkkhaki",
5A_15="gray2",5A_32="darkseagreen2")
But I do get this error after this code:
Error: unexpected symbol in "colorCodes <- c(1A_53"
## perform clustering
hc <- hclust(as.dist(dis2))
## function to set label color
labelCol <- function(x) {
if (is.leaf(x)) {
## fetch label
label <- attr(x, "label")
code <- substr(label, 1, 1)
## use the following line to reset the label to one letter code
# attr(x, "label") <- code
attr(x, "nodePar") <- list(lab.col=colorCodes[code])
}
return(x)
}
## apply labelCol on all nodes of the dendrogram
d <- dendrapply(as.dendrogram(hc), labelCol)
plot(d)
plot(as.phylo(hc), cex = 0.9, label.offset = 1)
1 ответ
Причиной вашей ошибки является то, что вы пытаетесь использовать имена переменных в "colorCodes", который начинается с цифры (то, что R не принимает). Вы можете обойти это, используя обратную галочку, чтобы обернуть необычное имя, например:
> c(`1A_53`="red")
1A_53
"red"
Но в любом случае, я думаю, что для окрашивания ваших этикеток было бы проще всего использовать labels_colors функция из пакета dendextend. Например:
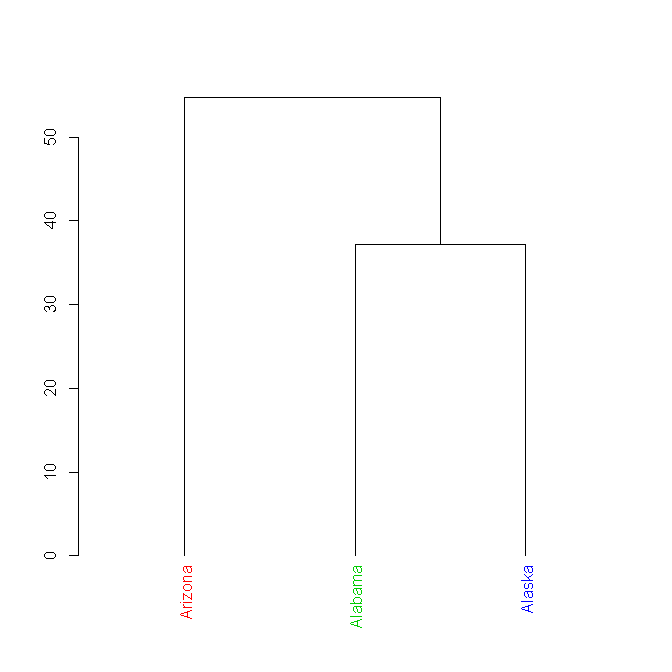
hc <- hclust(dist(USArrests[1:3,]), "ave")
dend <- as.dendrogram(hc)
# Defaults:
labels_colors(dend)
# NULL
plot(dend)
# let's add some color:
labels_colors(dend) <- 2:4
labels_colors(dend)
# Arizona Alabama Alaska
# 2 3 4
plot(dend)
Для более подробной информации о пакете, вы можете взглянуть на его виньетка.