Сделать матрицу символов 0/1 из случайного филогенетического дерева в R?
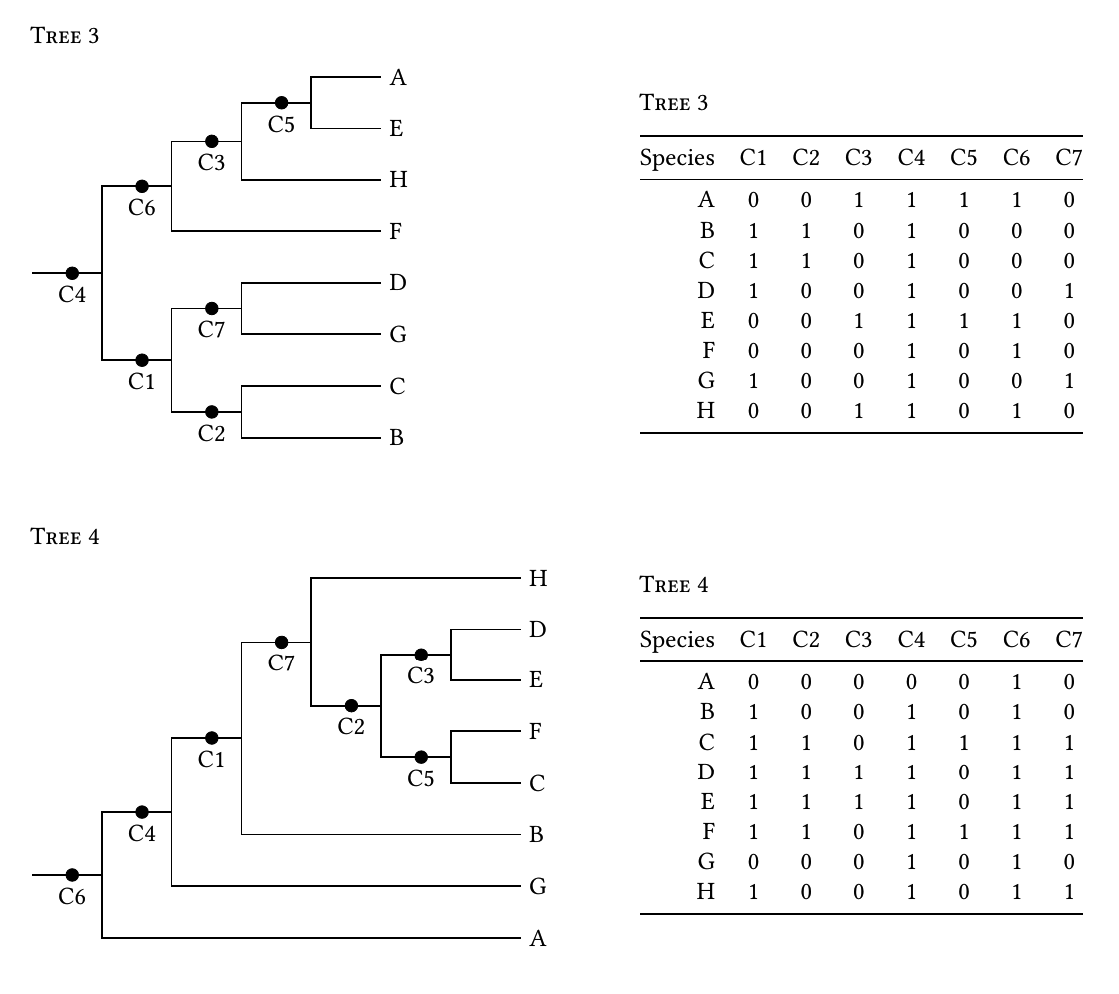
Можно ли сгенерировать 0/1 матрицы символов, подобные тем, которые показаны ниже справа от раздвоенных филогенетических деревьев, таких как слева. 1 в матрице указывается наличие общего символа, объединяющего клады.
Этот код генерирует красивые случайные деревья, но я понятия не имею, с чего начать превращать результаты в символьную матрицу.
library(ape) # Other package solutions are acceptable
forest <- rmtree(N = 2, n = 10, br = NULL)
plot(forest)
Чтобы было ясно, я могу использовать следующий код для генерации случайных матриц, а затем построить деревья.
library(ape)
library(phangorn)
ntaxa <- 10
nchar <- ntaxa - 1
char_mat <- array(0, dim = c(ntaxa, ntaxa - 1))
for (i in 1:nchar) {
char_mat[,i] <- replace(char_mat[,i], seq(1, (ntaxa+1)-i), 1)
}
char_mat <- char_mat[sample.int(nrow(char_mat)), # Shuffle rows
sample.int(ncol(char_mat))] # and cols
# Ensure all branch lengths > 0
dist_mat <- dist.gene(char_mat) + 0.5
upgma_tree <- upgma(dist_mat)
plot.phylo(upgma_tree, "phylo")
Я хочу создать случайные деревья, а затем сделать матрицы из этих деревьев. Это решение не делает правильный тип матрицы.
Отредактируйте для ясности: я генерирую двоичные матрицы символов, которые студенты могут использовать для рисования филогенетических деревьев, используя простую экономию. 1 символ представляет гомологии, которые объединяют таксоны в клады. Таким образом, все строки должны иметь один символ (1 по всем строкам в одном столбце) и некоторые символы должны быть общими только для двух таксонов. (Я обесцениваю аутапоморфии.)
Примеры:
2 ответа
Вы можете взглянуть на rTraitDisc функция в ape это довольно просто:
library(ape)
## You'll need to simulate branch length!
forest <- rmtree(N = 2, n = 10)
## Generate on equal rate model character
(one_character <- rTraitDisc(forest[[1]], type = "ER", states = c(0,1)))
# t10 t7 t5 t9 t1 t4 t2 t8 t3 t6
# 0 0 0 1 0 0 0 0 0 0
# Levels: 0 1
## Generate a matrix of ten characters
(replicate(10, rTraitDisc(forest[[1]], type = "ER", states = c(0,1))))
# [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
# t10 "0" "0" "0" "0" "1" "0" "0" "0" "0" "0"
# t7 "0" "0" "0" "0" "1" "0" "0" "0" "0" "0"
# t5 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t9 "0" "0" "1" "0" "0" "0" "0" "0" "0" "0"
# t1 "0" "0" "1" "0" "0" "0" "0" "0" "0" "0"
# t4 "0" "0" "1" "0" "0" "0" "0" "0" "0" "0"
# t2 "0" "0" "1" "0" "0" "0" "0" "0" "0" "0"
# t8 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t3 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t6 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
Чтобы применить его к нескольким деревьям, лучше всего создать функцию lapply следующим образом:
## Lapply wrapper function
generate.characters <- function(tree) {
return(replicate(10, rTraitDisc(tree, type = "ER", states = c(0,1))))
}
## Generate 10 character matrices for each tree
lapply(forest, generate.characters)
# [[1]]
# [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
# t10 "0" "0" "0" "1" "0" "0" "0" "0" "0" "0"
# t7 "0" "0" "0" "1" "0" "0" "0" "0" "0" "0"
# t5 "0" "0" "0" "1" "0" "0" "0" "0" "0" "0"
# t9 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t1 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t4 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t2 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t8 "0" "0" "0" "1" "0" "1" "0" "0" "0" "1"
# t3 "0" "0" "0" "0" "0" "1" "0" "0" "0" "0"
# t6 "0" "0" "0" "0" "0" "1" "0" "0" "0" "0"
# [[2]]
# [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
# t7 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t9 "1" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t5 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t2 "0" "0" "0" "0" "0" "0" "0" "0" "0" "0"
# t4 "0" "1" "0" "0" "1" "0" "0" "0" "0" "0"
# t6 "0" "1" "0" "0" "1" "0" "0" "0" "0" "0"
# t10 "0" "1" "1" "0" "1" "1" "0" "0" "0" "1"
# t8 "0" "1" "1" "0" "1" "0" "0" "0" "0" "0"
# t3 "0" "1" "0" "0" "0" "0" "0" "0" "0" "0"
# t1 "0" "1" "0" "0" "0" "0" "0" "0" "0" "0"
Другой вариант заключается в использовании sim.morpho от dispRity пакет. Эта функция повторно использует rTraitDisc функция, но имеет немного больше реализованных моделей и позволяет предоставлять скорости как распределения для выборки. Это также позволяет персонажам выглядеть более "реалистично" без большого количества инвариантных данных и обеспечения того, чтобы сгенерированный символ "выглядел" как настоящий морфологический персонаж (например, с правильным количеством гомопласий и т. Д.).
library(dispRity)
## You're first tree
tree <- forest[[1]]
## Setting up the parameters
my_rates = c(rgamma, rate = 10, shape = 5)
my_substitutions = c(runif, 2, 2)
## HKY binary (15*50)
matrixHKY <- sim.morpho(tree, characters = 50, model = "HKY",
rates = my_rates, substitution = my_substitutions)
## Mk matrix (15*50) (for Mkv models)
matrixMk <- sim.morpho(tree, characters = 50, model = "ER", rates = my_rates)
## Mk invariant matrix (15*50) (for Mk models)
matrixMk <- sim.morpho(tree, characters = 50, model = "ER", rates = my_rates,
invariant = FALSE)
## MIXED model invariant matrix (15*50)
matrixMixed <- sim.morpho(tree, characters = 50, model = "MIXED",
rates = my_rates, substitution = my_substitutions, invariant = FALSE,
verbose = TRUE)
Я предлагаю вам прочитать на sim.morpho функция для правильных ссылок о том, как работает модель, или в соответствующем разделе руководства по пакету dispRity.
Я разобрался как сделать матрицу используя Descendants из пакета phangorn. Мне все еще нужно настроить подходящие метки узлов, чтобы они соответствовали образцу матрицы в исходном вопросе, но рамки есть.
library(ape)
library(phangorn)
ntaxa <- 8
nchar <- ntaxa - 1
tree <- rtree(ntaxa, br = NULL)
# Gets descendants, but removes the first ntaxa elements,
# which are the individual tips
desc <- phangorn::Descendants(tree)[-seq(1, ntaxa)]
char_mat <- array(0, dim = c(ntaxa, nchar))
for (i in 1:nchar) {
char_mat[,i] <- replace(char_mat[,i], y <- desc[[i]], 1)
}
rownames(char_mat) <- tree$tip.label
char_mat
#> [,1] [,2] [,3] [,4] [,5] [,6] [,7]
#> t6 1 1 0 0 0 0 0
#> t3 1 1 1 0 0 0 0
#> t7 1 1 1 1 0 0 0
#> t2 1 1 1 1 1 0 0
#> t5 1 1 1 1 1 0 0
#> t1 1 0 0 0 0 1 1
#> t8 1 0 0 0 0 1 1
#> t4 1 0 0 0 0 1 0
plot(tree)

Создано в 2019-01-28 пакетом представлением (v0.2.1)