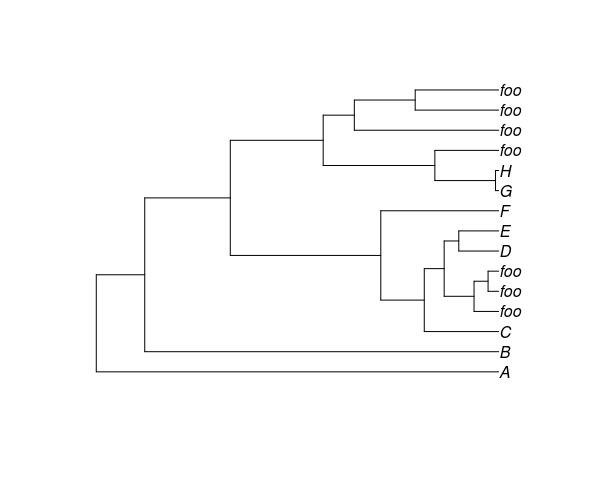
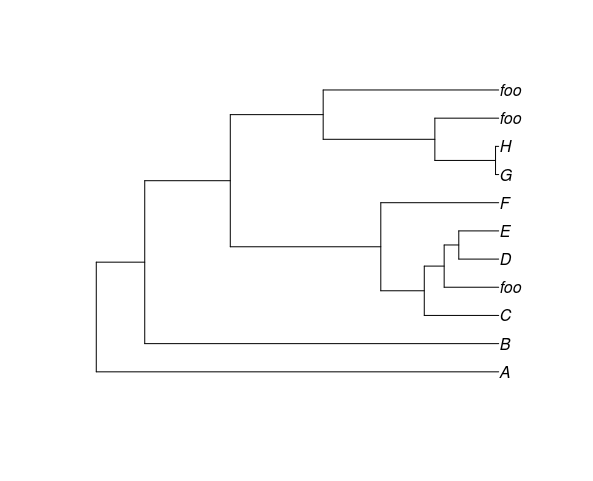
Свернуть кладу кончиками надписей, сохраняя филогенетическое положение
Возможно, я мог бы лучше сформулировать заголовок, но я хочу свернуть любую кладу в филогенетическом дереве (даже если у клады есть один член), у которой есть метка кончика "foo", а затем подсчитать количество кончиков, которые были отброшены из этот конкретный клад и создать ветку с меткой наконечника, отображающей 35 фу.
Подсчитать часть легко; однако, когда я использую
drop.tip(rooted.tree,tip=which(rooted.tree$tip.label=='foo'),subtree=TRUE)
упавшие кончики не сохраняют свое положение в дереве. Скорее, они все сгруппированы в конце (однако, подсчитаны правильно). Есть ли в любом случае свернуть кладу по меткам наконечника и сохранить свое положение на дереве?
1 ответ
Сложная часть определяет, сколько у вас есть отдельных монофилетических клад. Эта функция должна сделать свое дело.
collapse_identical_tips <- function(phy,tip_label){
matching_tips <- which(phy$tip.label==tip_label)
nt <- length(phy$tip.label) # number of tips in tree
nm <- length(matching_tips) # Number of tips matching the label
keep <- numeric(nm)
cur_tip <- 1
while(cur_tip<=nm){
if(cur_tip == nm){
keep[cur_tip] <- 1
break
}
next_tip <- cur_tip + 1
mrca_ <- getMRCA(phy,c(matching_tips[cur_tip],matching_tips[next_tip]))
descendants <- getDescendants(phy, mrca_)
descendant_tips <- descendants[descendants<=nt]
if(all(descendant_tips %in% matching_tips)){
keep[cur_tip] <- 1
cur_tip <- cur_tip + length(descendant_tips)
}else{
keep[cur_tip] <- 1
cur_tip <- cur_tip + 1
}
}
to_drop <- matching_tips[!keep]
new_phy <- drop.tip(phy,to_drop)
return(new_phy)
}
Тестирование это:
tc <- "(A:3.135206161,(B:2.757615245,(((C:0.5796267872,((foo:0.1917981792,(foo:0.08246947568,foo:0.08246947568):0.1093287035):0.2328473818,(D:0.3107268924,E:0.3107268924):0.1139186686):0.1549812262):0.3387382152,F:0.9183650024):1.172666972,(((G:0.02437174382,H:0.02437174382):0.4727952475,foo:0.4971669913):0.8701228492,(foo:1.124632261,(foo:0.6503599778,foo:0.6503599778):0.4742722831):0.2426575797):0.7237421344):0.6665832698):0.3775909166);"
phy <- read.tree(textConnection(tc))
plot(phy)
plot(collapse_identical_tips(phy,"foo"))