Разные кластеры одним и тем же методом
Я застрял в проблеме с иерархической кластеризацией. Я хочу сделать дендрограмму и тепловую карту с дистанционным методом корреляции (d_mydata=dist(1-cor(t(mydata)))) и ward.D2 в качестве метода кластеризации.
В качестве гаджета в pheatmap пакета вы можете построить дендрограмму на левой стороне, чтобы визуализировать кластеры.
Конвейер моего анализа будет следующим:
- создать дендрограмму
- проверить, сколько кластеров будет оптимальным (k)
- извлечь предметы в каждом кластере
- создать тепловую карту
Мое удивление возникает, когда дендрограмма, изображенная на тепловой карте, не совпадает с той, что была нанесена ранее, даже если методы совпадают.
Поэтому я решил создать раскраску pheatmap по кластерам, классифицированным ранее по cutree, и проверить, соответствуют ли цвета кластерам в дендрограмме.
Это мой код:
# Create test matrix
test = matrix(rnorm(200), 20, 10)
test[1:10, seq(1, 10, 2)] = test[1:10, seq(1, 10, 2)] + 3
test[11:20, seq(2, 10, 2)] = test[11:20, seq(2, 10, 2)] + 2
test[15:20, seq(2, 10, 2)] = test[15:20, seq(2, 10, 2)] + 4
colnames(test) = paste("Test", 1:10, sep = "")
rownames(test) = paste("Gene", 1:20, sep = "")
test<-as.data.frame(test)
# Create a dendrogram with this test matrix
dist_test<-dist(test)
hc=hclust(dist_test, method="ward.D2")
plot(hc)
dend<-as.dendrogram(hc, check=F, nodePar=list(cex = .000007),leaflab="none", cex.main=3, axes=F, adjust=F)
clus2 <- as.factor(cutree(hc, k=2)) # cut tree into 2 clusters
groups<-data.frame(clus2)
groups$id<-rownames(groups)
#-----------DATAFRAME WITH mydata AND THE CLASSIFICATION OF CLUSTERS AS FACTORS---------------------
test$id<-rownames(test)
clusters<-merge(groups, test, by.x="id")
rownames(clusters)<-clusters$id
clusters$clus2<-as.character(clusters$clus2)
clusters$clus2[clusters$clus2== "1"]= "cluster1"
clusters$clus2[clusters$clus2=="2"]<-"cluster2"
plot(dend,
main = "test",
horiz = TRUE, leaflab = "none")
d_clusters<-dist(1-cor(t(clusters[,7:10])))
hc_cl=hclust(d_clusters, method="ward.D2")
annotation_col = data.frame(
Path = factor(colnames(clusters[3:12]))
)
rownames(annotation_col) = colnames(clusters[3:12])
annotation_row = data.frame(
Group = factor(clusters$clus2)
)
rownames(annotation_row) = rownames(clusters)
# Specify colors
ann_colors = list(
Path= c(Test1="darkseagreen", Test2="lavenderblush2", Test3="lightcyan3", Test4="mediumpurple", Test5="red", Test6="blue", Test7="brown", Test8="pink", Test9="black", Test10="grey"),
Group = c(cluster1="yellow", cluster2="blue")
)
require(RColorBrewer)
library(RColorBrewer)
cols <- colorRampPalette(brewer.pal(10, "RdYlBu"))(20)
library(pheatmap)
pheatmap(clusters[ ,3:12], color = rev(cols),
scale = "column",
kmeans_k = NA,
show_rownames = F, show_colnames = T,
main = "Heatmap CK14, CK5/6, GATA3 and FOXA1 n=492 SCALE",
clustering_method = "ward.D2",
cluster_rows = TRUE, cluster_cols = TRUE,
clustering_distance_rows = "correlation",
clustering_distance_cols = "correlation",
annotation_row = annotation_row,
annotation_col = annotation_col,
annotation_colors=ann_colors
)
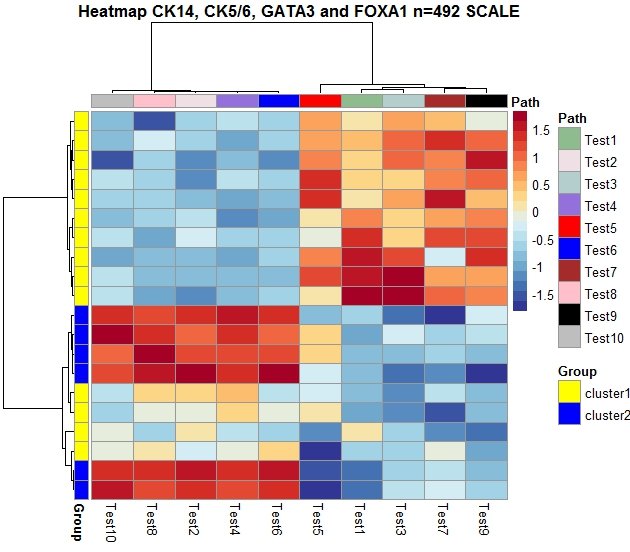
кто-нибудь с такой же проблемой? Я делаю глупую ошибку?
заранее спасибо