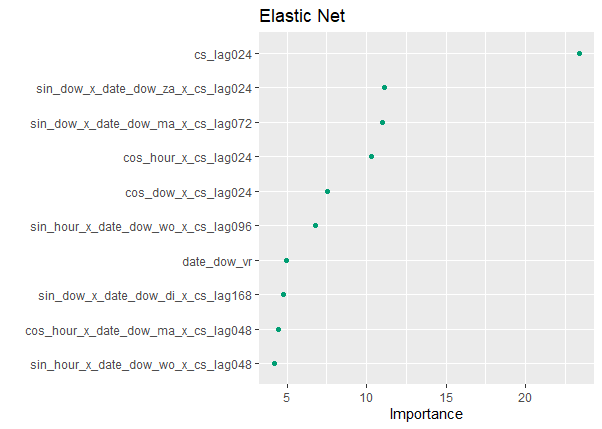
Интеграция графиков переменной важности в аккуратную структуру моделирования
Может ли кто-нибудь показать мне, как генерировать имплоты переменных на основе перестановок в аккуратной структуре моделирования? В настоящее время у меня есть это:
library(tidymodels)
# variable importance
final_fit_train %>%
pull_workflow_fit() %>%
vip(geom = "point",
aesthetics = list(color = cbPalette[4],
fill = cbPalette[4])) +
THEME +
ggtitle("Elastic Net")
который генерирует это:
Однако хотелось бы иметь что-то вроде этого
Мне непонятно, как довольно новый фреймворк аккуратного моделирования интегрируется с текущим пакетом VIP. Кто может помочь. Спасибо!
https://koalaverse.github.io/vip/articles/vip.html (API VIP-пакета).
1 ответ
Чтобы вычислить важность переменной с помощью перестановки, вам нужно собрать всего несколько частей, по сравнению с использованием важности переменной, зависящей от модели.
Давайте посмотрим на пример модели SVM, у которой нет оценки важности переменной, зависящей от модели.
library(tidymodels)
#> ── Attaching packages ──────────────────────── tidymodels 0.1.1 ──
#> ✓ broom 0.7.0 ✓ recipes 0.1.13
#> ✓ dials 0.0.8 ✓ rsample 0.0.7
#> ✓ dplyr 1.0.0 ✓ tibble 3.0.3
#> ✓ ggplot2 3.3.2 ✓ tidyr 1.1.0
#> ✓ infer 0.5.3 ✓ tune 0.1.1
#> ✓ modeldata 0.0.2 ✓ workflows 0.1.2
#> ✓ parsnip 0.1.2 ✓ yardstick 0.0.7
#> ✓ purrr 0.3.4
#> ── Conflicts ─────────────────────────── tidymodels_conflicts() ──
#> x purrr::discard() masks scales::discard()
#> x dplyr::filter() masks stats::filter()
#> x dplyr::lag() masks stats::lag()
#> x recipes::step() masks stats::step()
data("hpc_data")
svm_spec <- svm_poly(degree = 1, cost = 1/4) %>%
set_engine("kernlab") %>%
set_mode("regression")
svm_fit <- workflow() %>%
add_model(svm_spec) %>%
add_formula(compounds ~ .) %>%
fit(hpc_data)
svm_fit
#> ══ Workflow [trained] ════════════════════════════════════════════
#> Preprocessor: Formula
#> Model: svm_poly()
#>
#> ── Preprocessor ──────────────────────────────────────────────────
#> compounds ~ .
#>
#> ── Model ─────────────────────────────────────────────────────────
#> Support Vector Machine object of class "ksvm"
#>
#> SV type: eps-svr (regression)
#> parameter : epsilon = 0.1 cost C = 0.25
#>
#> Polynomial kernel function.
#> Hyperparameters : degree = 1 scale = 1 offset = 1
#>
#> Number of Support Vectors : 2827
#>
#> Objective Function Value : -284.7255
#> Training error : 0.835421
Наша модель теперь обучена и готова к вычислению важности переменных. Обратите внимание на пару шагов:
- Вы
pull()подобранный объект модели вне рабочего процесса. - Вы должны указать переменную цели / результата,
compounds. - В этом случае нам нужно передать как исходные данные обучения (используйте здесь данные обучения, а не данные тестирования), так и правильную базовую функцию для прогнозирования (в некоторых случаях это может быть сложно выяснить, но для большинства пакетов будет просто
predict()).
library(vip)
#>
#> Attaching package: 'vip'
#> The following object is masked from 'package:utils':
#>
#> vi
svm_fit %>%
pull_workflow_fit() %>%
vip(method = "permute",
target = "compounds", metric = "rsquared",
pred_wrapper = kernlab::predict, train = hpc_data)

Создано 17.07.2020 пакетом REPEX (v0.3.0)
Вы можете увеличить nsim вот делать это не раз.