Выбор количества листовых узлов дендрограммы в heatmap.2 в R
В Matlab вы можете указать количество узлов в дендрограмме, которые вы хотите построить как часть dendrogram функция: dendrogram(tree,P) генерирует график дендрограммы, содержащий не более P листовых узлов.
Мои попытки сделать то же самое с heatmap2 в R с треском провалились. Посты в stackru и biostars предложили использовать cutree но heatmap2 застревает с предложениями сообщений на Rowv вариант. Здесь "TAD" - это матрица данных 8 столбцов по 831 строк.
# cluster it
hr <- hclust(dist(TAD, method="manhattan"), method="average")
# draw the heat map
heatmap.2(TAD, main="Hierarchical Cluster",
Rowv=as.dendrogram(cutree(hr, k=5)),
Colv=NA, dendrogram="row", col=my_palette, density.info="none", trace="none")
возвращает сообщение:
Error in UseMethod("as.dendrogram") :
no applicable method for 'as.dendrogram' applied to an object of class "c('integer', 'numeric')"
Использует cutree правильный путь для построения ограниченной дендрограммы? Есть ли более простой способ сделать это сродни Matlab?
2 ответа
Вопрос в том, что вы имеете в виду, когда пишете "выбор количества конечных узлов".
Параметру Rowv в heatmap.2 требуется дендрограмма или значение ИСТИНА / ЛОЖЬ. Из файла справки:
Rowv = определяет, следует ли и как изменить порядок дендрограмм строк. По умолчанию это TRUE, что означает, что дендрограмма вычисляется и переупорядочивается на основе средних значений строк. Если NULL или FALSE, то дендрограмма не вычисляется и переупорядочение не выполняется. Если дендрограмма, то она используется "как есть", то есть без переупорядочения. Если вектор целых чисел, то дендрограмма вычисляется и переупорядочивается на основе порядка вектора.
Итак, при использовании cutree(hr, k=5), вы получите вектор целого числа (сообщающий, к какому кластеру относится каждый элемент, в разрезе, который производит 5 кластеров). С помощью as.dendrogram на нем не получится дендрограмма, следовательно: Rowv=as.dendrogram(cutree(hr, k=5)), выдает ошибку.
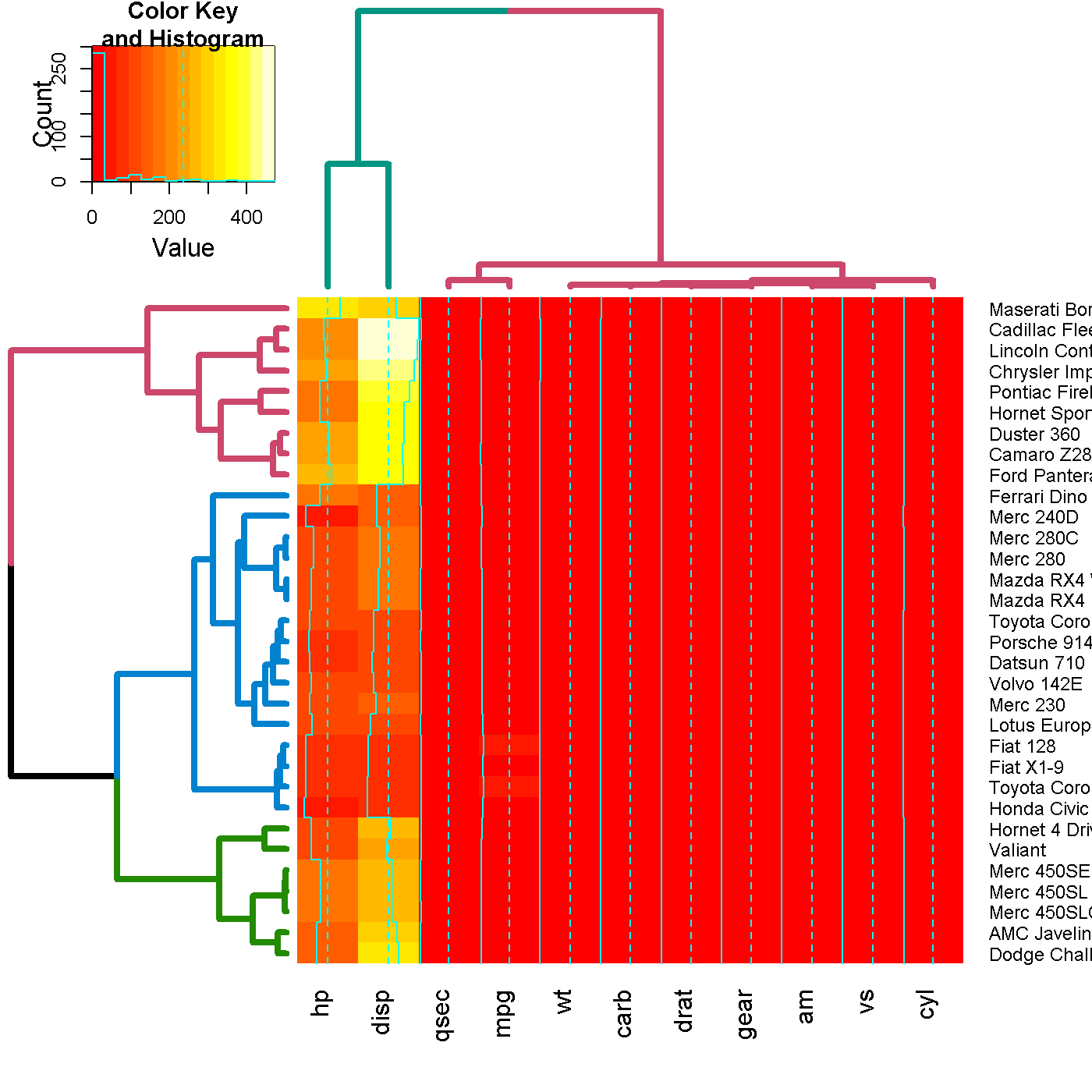
Если вы хотите выделить некоторые ветви в вашем дереве, для этого я предлагаю вам заглянуть в пакет dendextend, чтобы узнать, какое решение подходит вам лучше всего. Вот пример, который может быть тем, о чем вы просите:
library(gplots)
data(mtcars)
x <- as.matrix(mtcars)
# now let's spice up the dendrograms a bit:
Rowv <- x %>% dist %>% hclust %>% as.dendrogram %>%
set("branches_k_color", k = 3) %>% set("branches_lwd", 4) %>%
rotate_DendSer(ser_weight = dist(x))
Colv <- x %>% t %>% dist %>% hclust %>% as.dendrogram %>%
set("branches_k_color", k = 2) %>% set("branches_lwd", 4) %>%
rotate_DendSer(ser_weight = dist(t(x)))
heatmap.2(x, Rowv = Rowv, Colv = Colv)
Со следующим выводом:
Также обратите внимание на недавно опубликованное руководство dendextend, вы можете поработать с branches_attr_by_labels функция (в руководстве она находится в разделе: "Настройка ветвей на основе меток"), с возможностью манипулировать дендрограммами для создания таких графиков:
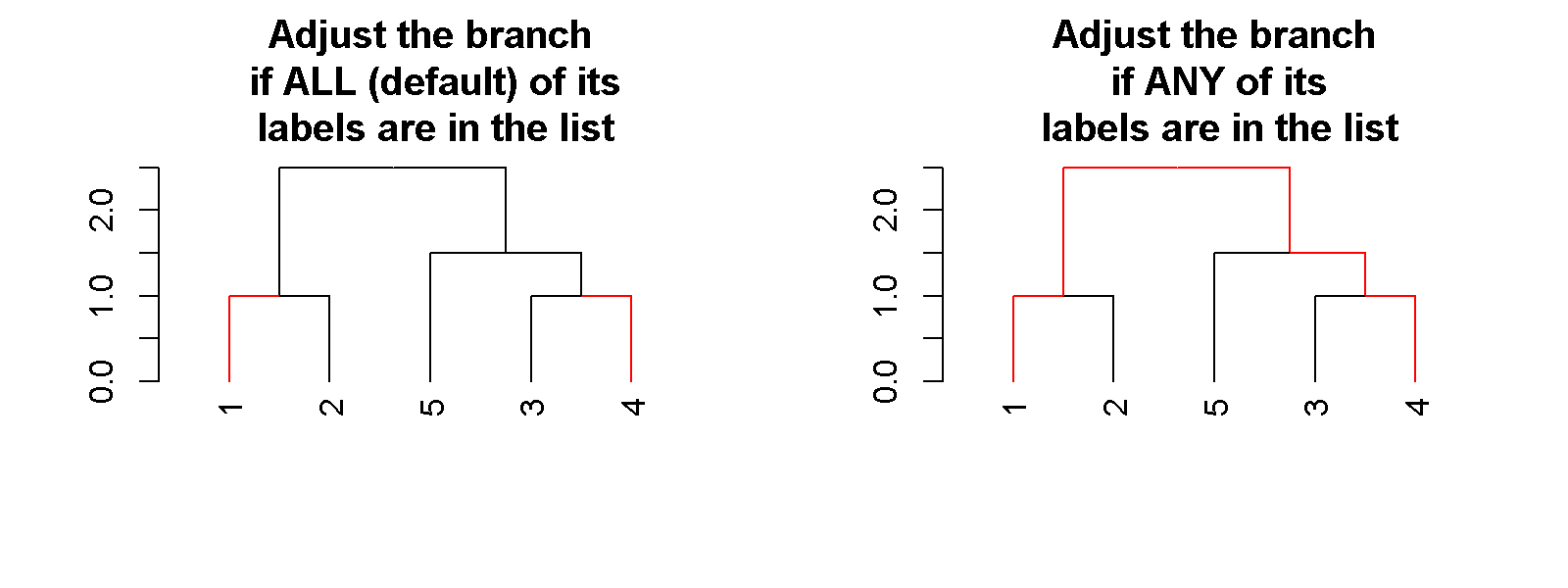
Если вам нужно удалить узлы и оставить на графике только несколько из них, вам, вероятно, следует просто создать тепловую карту для подмножества данных. Вы также можете посмотреть на prune функция в dendextend (для общего просмотра небольших дендрограмм), но если вы хотите использовать ее для тепловой карты, лучше просто работать с соответствующим подмножеством ваших данных.
Просто чтобы уточнить и предоставить некоторые данные... Я не хочу отбрасывать какие-либо строки; вместо того, чтобы строить / интерпретировать 831 ветвь, я хотел бы интерпретировать 3 ветви, и поэтому хотел бы, чтобы дендрограмма строк была ограничена 3 ветвями (на высоте 150), а соответствующая тепловая карта всех 831 строк была сгруппирована в 3 верхние ветви оригинальной дендрограммы.
#Here is a random n=10 subset of my data; which for 10 observed fish has the %of time each spent within
#a depth bin (Bin1-Bin8)
zz <- "ID Bin1 Bin2 Bin3 Bin4 Bin5 Bin6 Bin7 Bin8
1 0 0 0 0 0 0.0 0.0 100.0
2 0 0 0 0 0 0.0 0.0 100.0
3 0 0 0 0 0 0.0 0.0 100.0
4 0 0 0 0 0 70.8 29.2 0.0
5 0 0 0 100 0 0.0 0.0 0.0
6 0 0 0 0 0 0.0 93.3 6.7
7 0 0 0 0 0 27.5 72.5 0.0
8 0 0 0 0 0 53.5 46.5 0.0
9 0 0 0 0 0 0.0 100.0 0.0
10 0 0 0 0 0 0.0 72.1 27.9 "
TAD <- read.table(text=zz, header = TRUE)
IDnames <- TAD[,1]
x<-data.matrix(TAD[,2:ncol(TAD)])
rownames(x) <- IDnames
Не беспокоясь о тепловой карте, матрица расстояний и кластеризация выполняются на числовой матрице.
TAD.dist <- dist(x, method="manhattan", diag=FALSE, upper=FALSE)
TAD.cluster <- hclust(TAD.dist, method="average", members=NULL)
сюжет этой результирующей дендрограммы раскрывает все десять ветвей,
plot(TAD.cluster)
но высота среза 150 ограничит только 3 ветви
hcd = as.dendrogram(TAD.cluster)
rowDend<- cut(hcd, h = 150)$upper
plot(rowDend)
дендрограмма, построенная с помощью plot(rowDend) - это то, что я хотел бы видеть на дендрограмме строк для следующей тепловой карты
heatmap.2 (x,
distfun = function(x) dist(x, method='manhattan', diag=FALSE, upper=FALSE),
hclustfun = function(x) hclust(x,method = 'average'),
dendrogram = "row",
#Rowv=rowDend, #this is where I thought I could restrain the row dendrogram
Colv="NA",
trace="none",
)
Но я не могу найти способ ограничить строку дендрограммы в тепловой карте для нужного количества интерпретируемых ветвей. Построить все 831 ответвление крайне грязно.