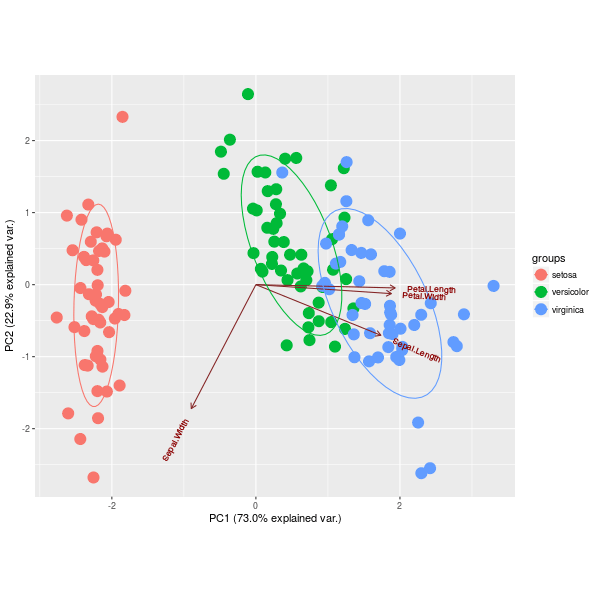
R: Как использовать ggbiplot с объектом pcaRes? построить результаты PCA данных с пропущенными значениями
Я обычно выполняю анализ основных компонентов с prcomp функция и представить результаты причудливым способом с ggbiplot (или иначе просто с ggplot2 экстрагирование pca.obj$x).
Как это:
#install_github("vqv/ggbiplot")
library(ggbiplot)
data(iris)
pca.obj <- prcomp(iris[,1:4], center=TRUE, scale.=TRUE)
P <- ggbiplot(pca.obj,
obs.scale = 1,
var.scale=1,
ellipse=T,
circle=F,
varname.size=3,
var.axes=T,
groups=iris$Species, #no need for coloring, I'm making the points invisible
alpha=0) #invisible points, I add them below
P$layers <- c(geom_point(aes(color=iris$Species), cex=5), P$layers) #add geom_point in a layer underneath (only way I have to change the size of the points in ggbiplot)
png(filename="test.png", height=600, width=600)
print(#or ggsave()
P
)
dev.off()
ОДНАКО, теперь я сталкиваюсь с данными с некоторым количеством NA, и я использую pca функция-обертка из пакета pcaMethods, применяя nipals метод (итерационный метод, способный обрабатывать небольшое количество пропущенных значений).
pca возвращает объект класса pcaRes, а также ggbiplot возвращает следующую ошибку:
#introduce NAs
iris$Sepal.Length[sample(1:150, 5)] <- NA
iris$Sepal.Width[sample(1:150, 5)] <- NA
iris$Petal.Length[sample(1:150, 5)] <- NA
iris$Petal.Width[sample(1:150, 5)] <- NA
#pca.obj2 <- prcomp(iris[,1:4], center=TRUE, scale.=TRUE) #cannot use prcomp with NAs
#source("https://bioconductor.org/biocLite.R")
#biocLite("pcaMethods")
library(pcaMethods)
pca.obj2 <- pca(iris[,1:4], method="nipals", nPcs=3, center=TRUE, scale.=TRUE)
class(pca.obj2)
ggbiplot(pca.obj2)
Ошибка в ggbiplot(pca.obj2): ожидается объект класса prcomp, princomp, PCA или lda
МОИ ВОПРОСЫ:
Как я могу подать заявку ggbiplot к pcaRes объект?
Как я могу преобразовать этот объект в prcomp объект?
Могу ли я получить такой же график с другой функцией вместо ggbiplot который принимает pcaRes объект?
Должен ли я просто заменить значения NA на среднее значение переменной и применить prcomp функционировать как обычно?
Большое спасибо!
1 ответ
Прежде всего, хорошая работа по поиску пакета PCA, который будет обрабатывать NA.
поскольку ggbiplot не примет pcaRes объекты, мы можем использовать данные, полученные pcaRes и проникнуть в оригинал prcomp объект.
Очевидно, ваши реальные данные уже будут содержать NA значения, поэтому мы начнем с этого набора данных и заменим их на некоторые фиктивные значения, чтобы мы могли запустить первый prcomppca,
iris_na<-iris
iris_na$Sepal.Length[sample(1:150, 5)] <- NA
iris_na$Sepal.Width[sample(1:150, 5)] <- NA
iris_na$Petal.Length[sample(1:150, 5)] <- NA
iris_na$Petal.Width[sample(1:150, 5)] <- NA
iris_dummy<-iris_na
iris_dummy[is.na(iris_dummy)]<-7777 #swap out your NAs with a dummy number so prcomp will run
Затем мы запускаем первый pca как вы сделали:
pca.obj <- prcomp(iris_dummy[,1:4], center=TRUE, scale.=TRUE)
Есть 5 компонентов этого объекта, x (баллы), rotation (нагрузки), sdev (стандартное отклонение), center а также scale, Хотя я подозреваю, что только баллы и нагрузки используются ggbiplotмы поменяем их все, чтобы быть уверенными.
Глядя на компонент оценки pca.obj$x показывает нам, что четыре основных компонента были рассчитаны в prcomp функция.
head(pca.obj$x)
# PC1 PC2 PC3 PC4
#[1,] -2.656740 0.3176722 0.03763067 -0.04122948
#[2,] -2.688275 -0.1821744 0.19912795 0.07297624
#[3,] -2.862673 -0.1447518 -0.02134749 -0.02462359
#[4,] -2.718294 -0.3189371 -0.03318459 -0.11675762
#[5,] -2.700864 0.3274887 -0.07503096 -0.11347939
#[6,] -2.252918 0.7436711 -0.14611455 -0.08218007
Поэтому, когда мы запускаем следующий PCA с pcaResмы обязательно указываем, что 4 главных компонента рассчитываются с использованием nPcs аргумент. Здесь мы используем достоверные данные, которые содержат NAs,
pca.obj2 <- pca(iris_na[,1:4], method="nipals", nPcs=4, center=TRUE, scale.=TRUE)
Тогда это просто вопрос обмена pcaRes значения для prcomp значения и передавая это ggbiplot
pca.obj$x<-pca.obj2@scores
pca.obj$rotation<-pca.obj2@loadings
pca.obj$sdev<-pca.obj2@sDev
pca.obj$center<-pca.obj2@center
pca.obj$scale<-pca.obj2@scale
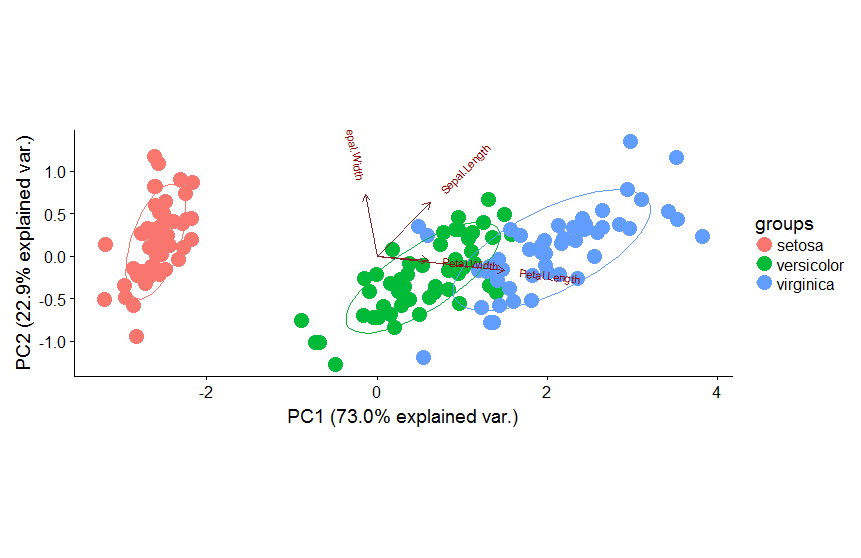
P2 <- ggbiplot(pca.obj,
obs.scale = 1,
var.scale=1,
ellipse=T,
circle=F,
varname.size=3,
var.axes=T,
groups=iris$Species,
alpha=0)
P2$layers <- c(geom_point(aes(color=iris$Species), cex=5), P2$layers)