Вывод из BlastP с использованием NCBIWWW не то, что я ожидал
Я пытаюсь получить результаты blastP для конкретного белка, используя NCBIWWW. Проблема в том, что то, что отправляется обратно, не является тем, что я распознаю как данные выравнивания, я получаю вот что (это содержимое "Blast_record" в исходном коде); Я использую код, полученный из "Учебника и поваренной книги по BioPython", я искал его и Интернет в поисках источника ошибки, но просто не могу его найти. Мой исходный код таков;
# biopython
from Bio import SeqIO
from Bio.Blast import NCBIWWW
from Bio.Blast import NCBIXML
# first get the sequence we want to parse from a FASTA file
# f_record = next(SeqIO.parse('m_cold.fasta', 'fasta'))
print('Doing the BLAST and retrieving the results...')
result_handle = NCBIWWW.qblast('blastp', 'tsa', '365176198')
# save the results for later, in case we want to look at it
save_file = open('m_cold_blast.out', 'w')
blast_results = result_handle.read()
save_file.write(blast_results)
save_file.close()
Полученный файл:
<?xml version="1.0"?>
<!DOCTYPE BlastOutput PUBLIC "-//NCBI//NCBI BlastOutput/EN" "http://www.ncbi.nlm.nih.gov/dtd/NCBI_BlastOutput.dtd">
<BlastOutput>
<BlastOutput_program>blastp</BlastOutput_program>
<BlastOutput_version>BLASTP 2.2.31+</BlastOutput_version>
<BlastOutput_reference>Stephen F. Altschul, Thomas L. Madden, Alejandro A. Sch&auml;ffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J. Lipman (1997), "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs", Nucleic Acids Res. 25:3389-3402.</BlastOutput_reference>
<BlastOutput_db>tsa</BlastOutput_db>
<BlastOutput_query-ID>gi|365176198|gb|AEW67975.1|</BlastOutput_query-ID>
<BlastOutput_query-def>polyprotein [Black queen cell virus] </BlastOutput_query-def>
<BlastOutput_query-len>171</BlastOutput_query-len>
<BlastOutput_param>
<Parameters>
<Parameters_matrix>BLOSUM62</Parameters_matrix>
<Parameters_expect>10</Parameters_expect>
<Parameters_gap-open>11</Parameters_gap-open>
<Parameters_gap-extend>1</Parameters_gap-extend>
<Parameters_filter>F</Parameters_filter>
</Parameters>
</BlastOutput_param>
<BlastOutput_iterations>
<Iteration>
<Iteration_iter-num>1</Iteration_iter-num>
<Iteration_query-ID>gi|365176198|gb|AEW67975.1|</Iteration_query-ID>
<Iteration_query-def>polyprotein [Black queen cell virus]</Iteration_query-def>
<Iteration_query-len>171</Iteration_query-len>
<Iteration_hits>
</Iteration_hits>
<Iteration_stat>
<Statistics>
<Statistics_db-num>0</Statistics_db-num>
<Statistics_db-len>0</Statistics_db-len>
<Statistics_hsp-len>0</Statistics_hsp-len>
<Statistics_eff-space>0</Statistics_eff-space>
<Statistics_kappa>-1</Statistics_kappa>
<Statistics_lambda>-1</Statistics_lambda>
<Statistics_entropy>-1</Statistics_entropy>
</Statistics>
</Iteration_stat>
</Iteration>
</BlastOutput_iterations>
</BlastOutput>
Теперь, если я выполняю поиск с использованием BlastN и нуклеотидной последовательности белка, использованного выше, я получаю все совпадающие последовательности, их значения E, баллы и т. Д. Так почему же это не так при использовании BlastP?
Я очень плохо знаком с Python и Biopython, и за всю свою жизнь я не могу понять, что я делаю неправильно.
2 ответа
QUERY 365176198 - это белок
Базы данных нуклеотики
Что такое база данных Transcriptome Shotgun Assembly (TSA)?
TSA - это архив вычислительно собранных последовательностей из первичных данных, таких как EST, трассировки и технологии секвенирования следующего поколения. Перекрывающиеся последовательности считываний из полного транскриптома собираются в транскрипты вычислительными методами вместо традиционного клонирования и секвенирования клонированных кДНК.
Доступны ли последовательности TSA поиском BLAST?
Доступна база данных BLAST Transcriptome Shotgun Assembly (TSA). Последовательности были первоначально включены в NT, но теперь были выделены в отдельную базу данных. База данных TSA доступна на домашней странице BLAST в разделе Basic BLAST по ссылкам нуклеотидов, tblastn и tblastx. Эти последовательности не доступны в NT.
BLAST FLAVORS
BLASTP:
сравнивает аминокислотную последовательность запроса с последовательностью белка
база данных
blastn: сравнивает последовательность запроса нуклеотидов с нуклеотидом
база данных последовательностей
blastx: сравнивает последовательность запроса нуклеотидов, переведенную во все
рамки считывания с базой данных последовательностей белков
tblastn: сравнивает последовательность запроса белка с нуклеотидом
база данных последовательностей динамически транслируется во все рамки чтения
tblastx: сравнивает шестикадровые переводы нуклеотидного запроса
последовательность против шестикадровых трансляций нуклеотида
база данных последовательностей. Обратите внимание, что программа tblastx не может быть
используется с базой данных nr на веб-странице BLAST.ВРУЧНУЮ ОТВЕТ
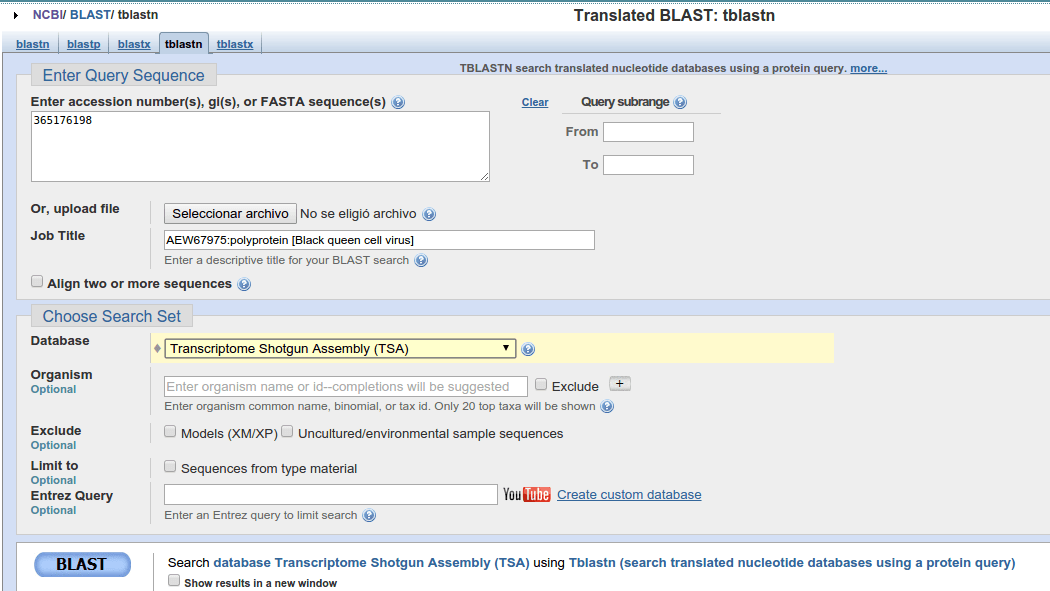
БИОПИТОН ОТВЕТ
вы должны использовать "tsa_nt" вместо "tsa" и "tblastn" вместо "blastp"
query = '365176198'
#note: this may take several minutes
result_handle = NCBIWWW.qblast('tblastn', 'tsa_nt', query, format_type="Text")
ты получаешь:
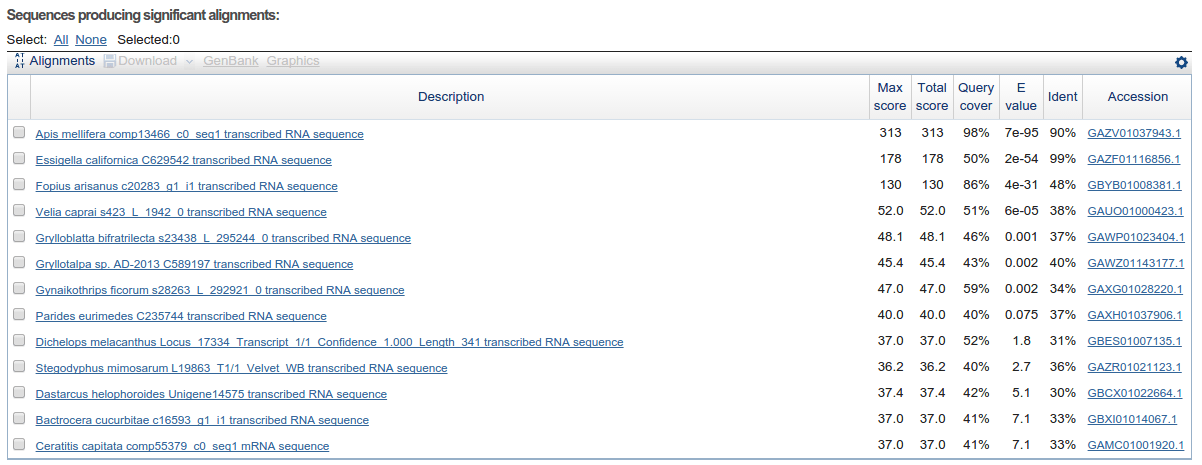
....... Оценка E Последовательности, приводящие к значительным выравниваниям: (биты) Значение gb|GAZV01037943.1| Apis mellifera comp13466_c0_seq1 транскрибируют... 342 4e-105
gb|GAZF01116856.1| Essigella californica C629542 расшифровывается... 179 1e-54
gb|GBYB01008381.1| Fopius arisanus c20283_g1_i1 транскрибировал R... 149 2e-37
gb|GAUO01000423.1| Velia caprai s423_L_1942_0 транскрибировал РНК... 58,9 3e-07
gb|GAXG01028220.1| Gynaikothrips ficorum s28263_L_292921_0 tr... 57,0 9e-07
gb|GAWP01023404.1| Grylloblatta bifratrilecta s23438_L_295244... 52,8 4e-05
gb|GAWZ01143177.1| Gryllotalpa sp. AD-2013 C589197 транскрибировать... 45,4 0,002 Гб |GAXW01013938.1| Euroleon nostras s13984_L_116369_0 transcr... 45.8 0.006
gb|GAXC01050700.1| Thrips palmi C235436 транскрибируется с последовательностью РНК 42,0 0,017 гб |GAXH01037906.1| Parides eurimedes C235744 транскрибировал РНК... 40,4 0,069 ГБ |GBES01007135.1| Dichelops melacanthus Locus_17334_Transcri... 39,7 0,18 Гб |GBXI01014067.1| Bactrocera cucurbitae c16593_g1_i1 transcr... 40,4 0,49 Гб |GAMC01001920.1| Capitata Ceratitis comp55379_c0_seq1 мРНК... 40,4 0,49 ГБ |GARL01030594.1| Spodoptera exigua SEUC25635_TC01 транскрибируют... 39,7 0,88 ГБ |GAZS01034153.1| Acanthoscurria geniculata L2169_T1/2_Turan... 38,5 2,1
gb|GAZS01034154.1| Acanthoscurria geniculata L2170_T2/2_Turan... 38,5 2,1 Гб |GAYD01030921.1| Blaberus atropos s30958_L_499964_0 transcr... 38,5 2,2 Гб |GAZR01021123.1| Stegodyphus mimosarum L19863_T1/1_Velvet_W... 36,2 3,0 Гб |GBCX01022664.1| Dastarcus helophoroides Unigene14575 trans... 37,7 3,8 Гб |GAYF01148415.1| Nilaparvata lugens C730037 транскрибируется РНК... 37,7 4,5 Гб |GAMK01054259.1| Phaseolus vulgaris Ref_259_comp8866_c0_seq... 37,0 8,5 Гб |EZ343106.1| Artemisia annua штамм Уганда Contig10322.Uhm ... 35,8 8,9 ВЫРАВНИВАНИЯ>gb|GAZV01037943.1| TSA: последовательность транскрибированной РНК Apis mellifera comp13466_c0_seq1 Длина =6998 баллов = 342 бита (878), ожидаемое = 4e-105, метод: корректировка композиционной матрицы. Тождества = 164/168 (98%), Положительных = 167/168 (99%), Пробелы = 0/168 (0%) Рама = +2 Запрос 4 YALYRGGVRVKVVTGRGVDFVRATVSPQQTYGSEVAPTTHISTPLAIEQIPIKGVAEFQI 63
YALYRGGVRVKVVT +GVDFVRATVSPQQTYGS+VAPTTHISTPLAIEQIPIKGVAEFQI
Sbjct 6317 YALYRGGVRVKVVTEKGVDFVRATVSPQQTYGSDVAPTTHISTPLAIEQIPIKGVAEFQI 6496 Запрос 64 PYYAPCLSSSFRANSETFYYSSGRNNLDIATSPPSINRYYAVGAGDDMDFSIFIGTPPCI 123
PYYAPCLSSSFRANSETFYYSSGRNNLDI+TSPPSINRYYAVGAGDDMDFSIFIGTPPCI
Sbjct 6497 PYYAPCLSSSFRANSETFYYSSGRNNLDISTSPPSINRYYAVGAGDDMDFSIFIGTPPCI 6676
.......
Я заметил, что ваш выходной файл заканчивается на.out. Попробуйте сохранить его в файл XML, и он отобразит их в красивые столбцы. В первой строке выходного файла вы увидите "? Xml. Функция qBLAST использует XML по умолчанию и также имеет необязательный аргумент для "текста", хотя форматирование - это кошмар.
Также верно, что blastp и tsa - это разные базы данных. Модуль qblast имеет некоторую встроенную справку, которая может помочь с различными аргументами, к которым можно получить доступ с помощью этого.
>>> from Bio.Blast import NCBIWWW
>>> help(NCBIWWW.qblast)
(Я бы запустил ваш код, но у qBLAST есть проблемы прямо во время написания этой статьи)