Масштаб радиуса изменения ggtree
Я пытаюсь создать филогенетическое дерево с помощью ggtree. У меня возникают проблемы с изменением размера центра дерева, чтобы символы на ребре, представляющие мои последовательности, не перекрывали друг друга и увеличивали радиус, чтобы длина ветвей не была такой длинной. Любые советы или советы о том, как это сделать, будут высоко оценены. Заранее спасибо!
Я использую следующий скрипт для создания дерева...
ggtree(PhyMLAATree, layout = "circular") +
geom_tippoint(aes(x=x+.01), shape=Shape, fill=Fill, size=3)
Эти два изображения показывают, что я создаю и что я пытаюсь создать. Дерево выглядит следующим образом: 
Хотелось бы, чтобы итоговое дерево выглядело так с гораздо большим центром: 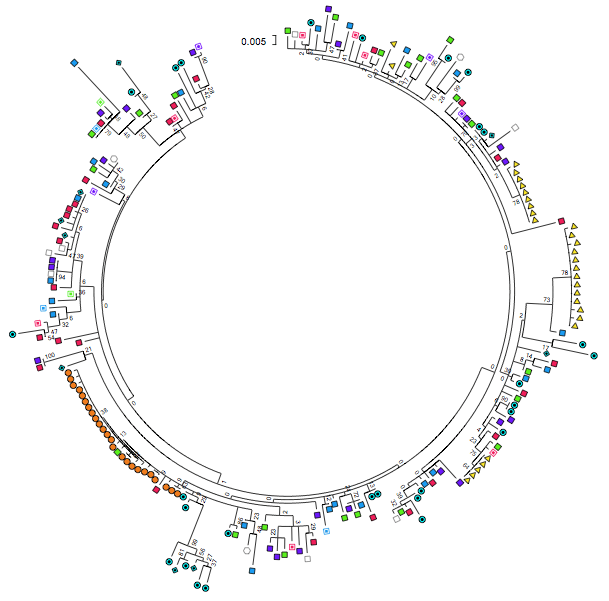
Я попытался выделить устройство с большим пространством, но я получил те же пропорции на дереве (кварц (ширина =12, высота =12)
1 ответ
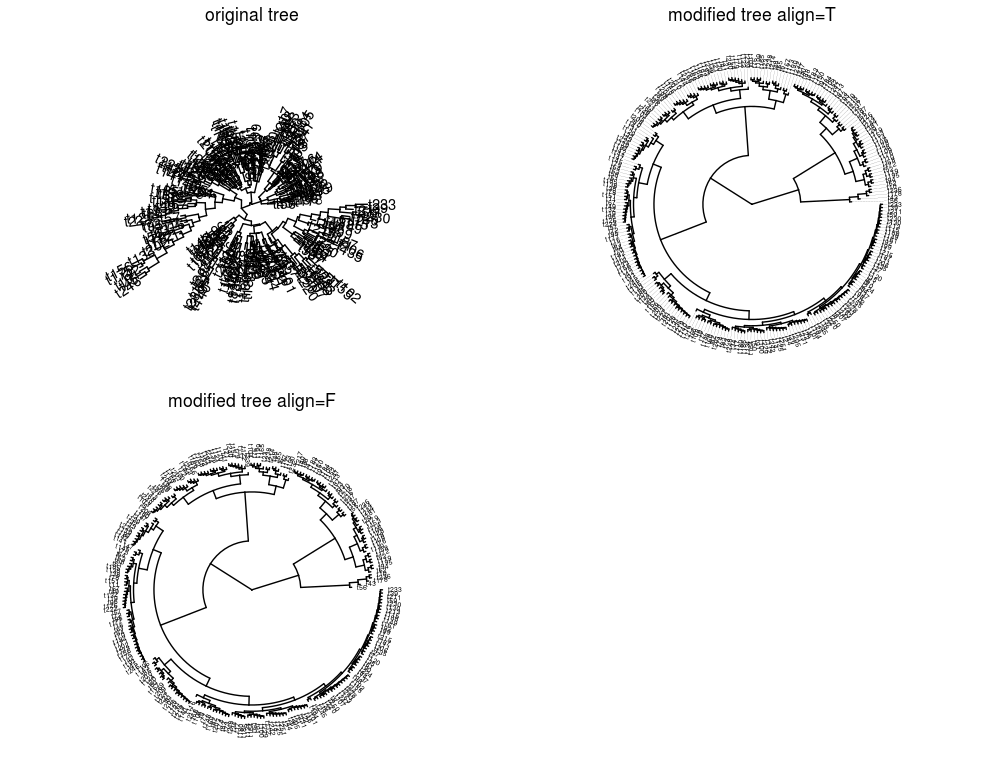
В моем подходе я воссоздал длины ребер, следуя отрицательному экспоненциальному распределению от внутренних к внешним ребрам по иерархическим уровням.
library(ggtree) # install as in:
#source("https://bioconductor.org/biocLite.R")
#biocLite("ggtree")
library(data.tree)
library(gridExtra)
library(rlist)
library(phangorn)
#create random tree
tree<-ape::rtree(251)
#its plot
originalplot<-ggtree(tree, layout="circular")+ geom_tiplab2() + labs(title="original tree") +
theme(plot.title = element_text(hjust = 0.5))
transformtree<-function(tree,radialparameter,repeatparameter,tiplength){
# radialparameter # # change this to collapse less(0.5) or more (3) and modify repeatparameter together
# repeatparameter # # i.e. increase if there are very small branches (levels)
#number of hierarchical levels in tree
dfr0<-as.data.frame(tree$edge)
tree2<-FromDataFrameNetwork(dfr0)# data.tree package
levels<-ToDataFrameTable(tree2, "level")
edgelevels<-max(levels)-1
# establish the hierarchy of nodes looking for the children of the children nodes
centralnode<-getMRCA(tree,1:length(tree$tip.label))
childrenlist<-list()
childrenlist[1]<-list(phangorn::Children(tree, centralnode))
for (i in 2:edgelevels){
childrenlist[i]<- list(unlist(lapply(unlist(childrenlist[i-1]), function(x) phangorn::Children(tree, x) ) ) )
}
# remove nodes of tips, we do not want to modify their length
childrentipsremoved<-lapply(childrenlist, function(x) x[!is.element(x,1:length(tree$tip.label))])
# list of inner nodes
groupedinnernodes<-rlist::list.clean(childrentipsremoved, fun = function(x) length(x) == 0L)
#this is the vector that will multiply the inner edges
transfvector<- rep(((c(1:(length(groupedinnernodes)/repeatparameter))^(-radialparameter) )*5),
each=repeatparameter)
# check length of groups of inner nodes and the transformation vector
lengths<-unlist(lapply(groupedinnernodes, function(x) length(x)) )
if(length(lengths)-length(transfvector)>0) {
for (i in 1:abs(length(lengths)-length(transfvector) ) ){
transfvector <- c(transfvector,transfvector[length(transfvector)])
} }
if(length(lengths)-length(transfvector)<0) {
for (i in 1:abs(length(lengths)-length(transfvector) ) ){
transfvector <- transfvector[-1] }}
# create the factor to transform the inner edges
vector1<-unlist(mapply(rep, transfvector,lengths) )
# discard length info, replace all edge length information by 1
size<-length(tree$edge.length)
tree$edge.length<-rep(1,size)
# replace edge length for the connecting inner nodes only
innernodes<-unlist(groupedinnernodes)
tree$edge.length[unlist(lapply(innernodes,function(x,y) which(y==x),y=tree$edge[,2]) )]<-
tree$edge.length[unlist(lapply(innernodes,function(x,y) which(y==x),y=tree$edge[,2]) )]*
vector1
# modify length of tip edges # optional decrease for big trees
tree$edge.length[tree$edge.length==1]<-tiplength
return(tree)
}
tree<-transformtree(tree,2.5,2,0.2)
# plot of modified tree aligned tips
newplot<-ggtree(tree, layout="circular")+ geom_tiplab2(align=T,linetype=1, linesize=.02,
size=1.8, offset=0.5) + labs(title="modified tree align=T")+
theme(plot.title = element_text(hjust = 0.5))
# plot of modified tree not aligned tips
newplot2<-ggtree(tree, layout="circular")+ geom_tiplab2(align=F,linetype=1, linesize=.02,
size=1.8, offset=0.5) + labs(title="modified tree align=F")+
theme(plot.title = element_text(hjust = 0.5))
#plots
gridExtra::grid.arrange(originalplot,newplot,newplot2,ncol=2)
Это можно сделать, указав xlim с участием layout = "circular", как это:
nwk <- system.file("extdata", "sample.nwk", package="treeio")
tree <- read.tree(nwk)
library(ggtree)
ggtree(tree, layout = "circular") +
geom_tippoint() +
geom_tiplab2(offset = 7) +
xlim(-150, NA)
Как описано в документации ggtree на рис. 4.3. Поиграйте сxlim значение, чтобы изменить диаметр центра.