Интерполяция временных рядов пропущенных значений в столбце в r
Я в настоящее время смотрел на imputeTS и пакеты зоопарка, но он не видит, чтобы работать Текущие данные..
group/timeseries(character)
1 2017-05-17 04:00:00
1 2017-05-17 04:01:00
1 NA
1 NA
1 2017-05-17 05:00:00
1 2017-05-17 06:00:00
2 NA
2 2017-05-17 04:31:00
2 NA
2 NA
2 NA
2 2017-05-17 05:31:00
Я хотел бы заполнить NA временными рядами интерполяции, чтобы время было средней точкой строки до и после. Также я должен отметить, что каждый временной ряд принадлежит группе. Значение времени сбрасывается для каждой группы.
Я предоставлю картину фактических данных, чтобы быть более четким 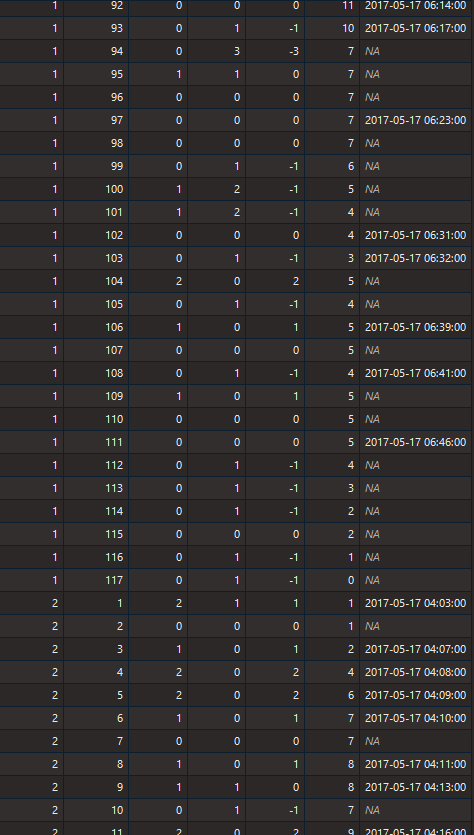
Спасибо за помощь в продвижении!
2 ответа
imputeTS и zoo не принимают символы или временные метки в качестве входных данных для своих функций интерполяции. (обычно интерполяция символов не имеет смысла)
Но вы можете дать символы в качестве входных данных для функции na.locf в zoo. (последнее наблюдение переносится с этой функцией)
Лучшее решение для вашей задачи должно быть следующим (я предполагаю, что у вас есть дата, указанная как POSIX.ct)
# Perform the imputation on numeric input
temp <- imputeTS::na.interpolation( as.numeric ( input ) )
# Transform the numeric values back to dates
as.POSIXct(temp, origin = "1960-01-01", tz = "UTC")
С "input" в первой строке будет ваш вектор с временными метками POSIX.ct. Настройки origin и tz (часовой пояс) во второй строке должны быть установлены в соответствии с вашими временными метками.
na.approx в пакете zoo это можно сделать, и группировка может быть выполнена без циклов, используя либо tapply в базе или как групповая операция в data.table.
Для вашего набора данных
df <- read.table(text=c("
group timeseries
1 '2017-05-17 04:00:00'
1 '2017-05-17 04:01:00'
1 NA
1 NA
1 '2017-05-17 05:00:00'
1 '2017-05-17 06:00:00'
2 NA
2 '2017-05-17 04:31:00'
2 NA
2 NA
2 NA
2 '2017-05-17 05:31:00'
"),
colClasses = c("integer", "POSIXct"),
header = TRUE)
Написать функцию, чтобы привести вектор к объекту зоопарка, интерполировать NA, извлечь результат
library(zoo)
foo <- function(x) coredata(na.approx(zoo(x), na.rm = FALSE))
Пример использования tapply в базе R для применения foo к каждой группе
df2 <- df #make a copy
df2$timeseries <- do.call(c, tapply(df2$timeseries, INDEX = df2$group, foo))
Пример использования group by в data.table для применения foo к каждой группе
library(data.table)
DT <- data.table(df)
DT[, timeseries := foo(timeseries), by = "group"]