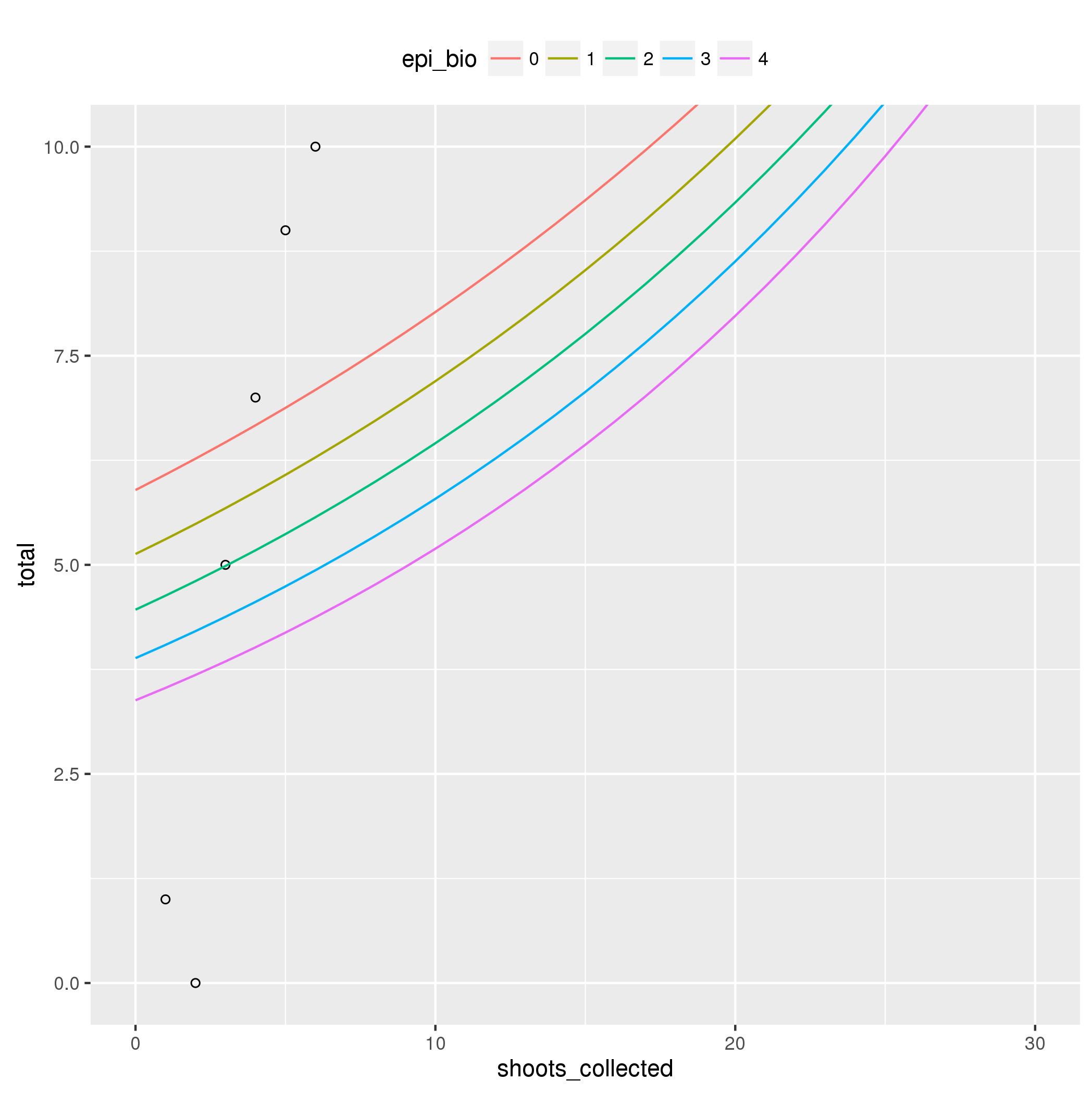
Расширение линий регрессии с помощью geom_line() ggplot2
Я использовал отрицательный блиномиальный глм, чтобы посмотреть на численность позвоночных в водорослях. Поскольку у меня был термин взаимодействия, я предсказал некоторые значения для численности рыбы. Я хотел бы, чтобы эти предсказанные линии регрессии достигли конца пространства графика. Прямо сейчас они все отключаются в разное время, как в примере ниже:
total<-c(1,0,5,7,9,10,23,45,78,100)
shoots_collected<-c(1,2,3,4,5,6,7,45,67,88)
epi_bio<-c(0.0,11,0.89,1.5,9,5,.04,6,7,.9)
Year<-c(1,1,1,1,2,2,2,2,1,1)
Year<-as.factor(Year)
intertidal<-data.frame(shoots_collected,Year,epi_bio, total)
glm.neg<-glm.nb(total~Year+shoots_collected+epi_bio+shoots_collected*epi_bio,
data=intertidal)
summary(glm.neg)
abun_shoots2015<-data.frame("shoots_collected"=rep(0:30, rep(5,31)),
"epi_bio"=rep(c(0,1,2,3,4), 31), "Year"=rep("1", 155))
# then extracted predicted values using:
p2015<-predict(glm.neg, newdata=abun_shoots2015, se.fit=TRUE, type='response')
abun_shoots2015$fit<-p2015$fit
ggplot(intertidal, aes(x=shoots_collected, y=total)) +
scale_x_continuous(limits = c(0, 30))+
scale_y_continuous(limits=c(0,10))+
geom_point(pch=1)+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==0.0000),], aes(x=shoots_collected, y=fit), col="red")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==1),], aes(x=shoots_collected, y=fit), col="green")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==2),], aes(x=shoots_collected, y=fit), col="blue")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==3),], aes(x=shoots_collected, y=fit), col="yellow")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==4),], aes(x=shoots_collected, y=fit), col="pink")
Я ранее использовал lines() команда, но перешел на geom_lines() чтобы я мог использовать fullrange=TRUE но это все равно не сработало. Я вижу, что у меня есть некоторые пропущенные значения, когда я пытаюсь построить линии, и я подозреваю, что именно поэтому некоторые из них обрезаются, но я не знаю, куда идти дальше.
1 ответ
Вы не хотите использовать scale_y_continuous и т. д. здесь, поскольку они имеют эффект полной обрезки данных, выходящих за установленные пределы. Вместо этого вы хотите ограничить диапазон графика, чтобы показать только часть данных. Это сделано с coord_cartesian(), как в:
ggplot(intertidal, aes(x=shoots_collected, y=total)) +
coord_cartesian(xlim = c(0, 30), ylim = c(0,10)) + ## KEY!
geom_point(pch=1)+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==0.0000),],
aes(x=shoots_collected, y=fit), col="red")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==1),],
aes(x=shoots_collected, y=fit), col="green")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==2),],
aes(x=shoots_collected, y=fit), col="blue")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==3),],
aes(x=shoots_collected, y=fit), col="yellow")+
geom_line(data=abun_shoots2015[which(abun_shoots2015$epi_bio==4),],
aes(x=shoots_collected, y=fit), col="pink")
Кроме того, я чувствую себя обязанным добавить, что ваш сюжет может быть лучше подготовлен epi_bio как фактор:
ggplot(intertidal, aes(x=shoots_collected, y=total)) +
coord_cartesian(xlim = c(0, 30), ylim = c(0,10)) + ## KEY!
geom_point(pch=1) +
geom_line(data = abun_shoots2015, aes(y = fit, colour = as.factor(epi_bio))) +
scale_colour_discrete(name = "epi_bio") +
theme(legend.position = "top")