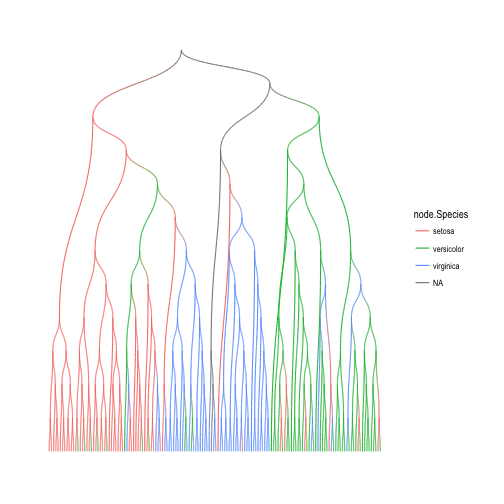
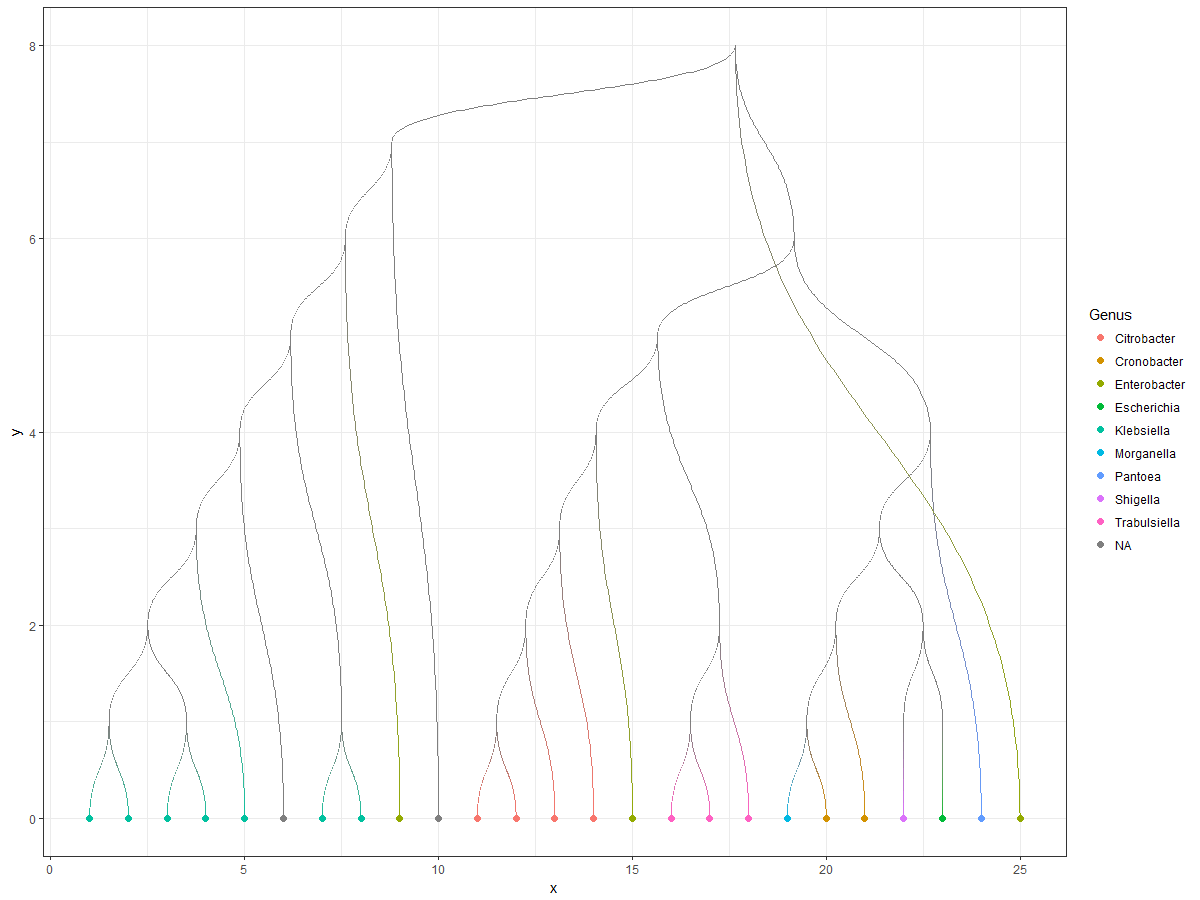
Распространение значения от детей с тидиграфом
У меня есть дерево, аннотированное на уровне рода (т. Е. У каждого листа есть имя), и я хочу распространять цвет листьев в ветвях / краях, пока у детей тот же род, как на этом графике:
Мое дерево здесь (извините, dput не работает...) и он выглядит так:
library(ggraph)
library(tidygraph)
load("tree_v3")
TBL %>% activate(nodes) %>% as_tibble
# A tibble: 50 x 2
leaf Genus
<lgl> <fctr>
1 FALSE NA
2 TRUE Klebsiella
3 TRUE Klebsiella
4 FALSE NA
5 TRUE Klebsiella
6 TRUE Klebsiella
7 FALSE NA
8 FALSE NA
9 TRUE Klebsiella
10 FALSE NA
# ... with 40 more rows
Я могу напечатать дерево с этим кодом, но, как вы можете видеть, цвета краев остаются рядом с листьями.
TBL %>%
ggraph('dendrogram') +
theme_bw() +
geom_edge_diagonal2(aes(color = node.Genus)) +
scale_edge_color_discrete(guide = FALSE) +
geom_node_point(aes(filter = leaf, color = Genus), size = 2)
В этом посте есть код, который отображает результаты поиска в этом блоге, но он не работает с моими данными, и я не понимаю, почему...
TBL2 <- TBL %>%
activate(nodes) %>%
mutate(Genus = map_bfs_back_chr(node_is_root(), .f = function(node, path, ...) {
nodes <- .N()
if (nodes$leaf[node]) return(nodes$Genus[node])
if (anyNA(unlist(path$result))) return(NA_character_)
path$result[[1]]
}))
Ошибка в mutate_impl(.data, dots): Ошибка оценки: невозможно привести значения к символу (1).
РЕДАКТИРОВАТЬ после ответа Марко Сандри
С mutate(Genus = as.character(Genus)) больше нет сообщений об ошибках, но род не распространяется правильно. Например, посмотрите третий и четвертый узлы, начиная справа: родительский элемент должен быть NA... (обратите внимание, что это не работает ни в сюжете поста в блоге).
1 ответ
Genus в TBL является фактором:
str(TBL %>% activate(nodes) %>% as_tibble)
# Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 50 obs. of 2 variables:
# $ leaf : logi FALSE TRUE TRUE FALSE TRUE TRUE ...
# $ Genus: Factor w/ 10 levels "","Citrobacter",..: NA 6 6 NA 6 6 NA NA 6 NA ...
но должен быть персонажем.
После конвертации Genus от фактора к символу, код работает.
TBL2 <- TBL %>%
activate(nodes) %>%
mutate(Genus = as.character(Genus)) %>%
mutate(Species = map_bfs_back_chr(node_is_root(), .f = function(node, path, ...) {
nodes <- .N()
if (nodes$leaf[node]) return(nodes$Genus[node])
if (anyNA(unlist(path$result))) return(NA_character_)
path$result[[1]]
}))