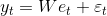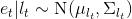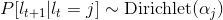Многомерная эмиссионная скрытая марковская модель в STAN
Я пытаюсь реализовать HMM, где наблюдения - это выбросы из HMM первого порядка, свернутые с вейвлетом.
То есть:
С:
а также
Код, который я имею до сих пор, следующий за одномерным случаем, изложенным здесь:
%%writefile HMM_code.stan
data {
int<lower=1> T; // number of observations (length)
int<lower=1> K; // number of hidden states
int<lower=1> H; // number of elements in wavelet vector
vector[K] dir_alpha;
real y[T]; // observations
matrix[T,H] W; // Wavelet matrix
cov_matrix[H] I; //prior covariance
}
parameters {
// Discrete state model
simplex[K] pi1; // initial state probabilities
simplex[K] A[K]; // transition probabilities
// A[i][j] = p(z_t = j | z_{t-1} = i)
// Continuous observation model
vector[H] Mu[K]; // observation means
real<lower=0> sigma; // observation standard deviations
cov_matrix[H] Sigma[K];
vector[T] eei[H]; //
}
transformed parameters {
vector[K] logalpha[T];
{ // Forward algorithm log p(z_t = j | y_{1:t})
real accumulator[K];
logalpha[1] = log(pi1) + multi_normal_lpdf(eei[1] | Mu[][1], Sigma[][1]);
for (t in 2:T) {
for (j in 1:K) { // j = current (t)
for (i in 1:K) { // i = previous (t-1)
// Murphy (2012) p. 609 eq. 17.48
// belief state + transition prob + local evidence at t
accumulator[i] = logalpha[t-1, i] + log(A[i, j]) + multi_normal_lpdf(eei[t] | Mu[][j], Sigma[][i]);
}
logalpha[t, j] = log_sum_exp(accumulator);
}
}
} // Forward
}
model{
Mu ~ multi_normal(rep_vector(0.0,H),I);
sigma ~ inv_gamma(.1,.1);
for(k in 1:K)
A[k] ~ dirichlet(dir_alpha);
for( j in 1:K)
Sigma[j] ~ inv_wishart(3.0, I); //Relevant part for this post
for (t in 1:T)
y[t] ~ normal(W[t,]*eei[t], pow(sigma,.5));// Likelihood
}
generated quantities {
vector[K] alpha[T];
vector[K] beta[T];
vector[K] gamma[T];
vector[K] loggamma[T];
int<lower=1, upper=K> zstar[T];
{ // Forward algortihm
for (t in 1:T)
alpha[t] = softmax(logalpha[t]);
} // Forward
{ // Backward algorithm log p(eei_{t+1:T} | z_t = j)
real accumulator[K];
vector[K] logbeta[T];
for (j in 1:K)
logbeta[T, j] = 1;
for (tforward in 0:(T-2)) {
int t;
t = T - tforward;
for (j in 1:K) { // j = previous (t-1)
for (i in 1:K) { // i = next (t)
// Murphy (2012) Eq. 17.58
// backwards t + transition prob + local evidence at t
accumulator[i] = logbeta[t, i] + log(A[j, i]) + multi_normal_lpdf(eei[t] | Mu[][i], Sigma[][i]);
}
logbeta[t-1, j] = log_sum_exp(accumulator);
}
}
for (t in 1:T)
beta[t] = softmax(logbeta[t]);
} // Backward
{ // forward-backward algorithm log p(z_t = j | eei_{1:T})
for(t in 1:T) {
loggamma[t] = alpha[t] .* beta[t];
}
for(t in 1:T)
gamma[t] = softmax(loggamma[t]);
} // forward-backward
{ // Viterbi algorithm
real logp_zstar;
int bpointer[T, K]; // backpointer to the most likely previous state on the most probable path
real delta[T, K]; // max prob for the sequence up to t
// that ends with an emission from state k
for (j in 1:K)
delta[1, K] = multi_normal_lpdf(eei[1] | Mu[][j], Sigma[][j]);
for (t in 2:T) {
for (j in 1:K) { // j = current (t)
delta[t, j] = negative_infinity();
for (i in 1:K) { // i = previous (t-1)
real logp;
logp = delta[t-1, i] + log(A[i, j]) + multi_normal_lpdf(eei[t] | Mu[][i], Sigma[][i]);
if (logp > delta[t, j]) {
bpointer[t, j] = i;
delta[t, j] = logp;
}
}
}
}
logp_zstar = max(delta[T]);
for (j in 1:K)
if (delta[T, j] == logp_zstar)
zstar[T] = j;
for (t in 1:(T - 1)) {
zstar[T - t] = bpointer[T - t + 1, zstar[T - t + 1]];
}
}
}
Скомпилировано с использованием:
%%time
sm = pystan.StanModel(file='HMM_code.stan')
И подходит с помощью:
%%time
fit = sm.sampling(data=HMM_data, iter=1, thin = 1, verbose = True)
Ошибка, которую я получаю, находится в первой строке при оценке функции multi_normal_pdf, говоря, что "LDLT_Factor параметра ковариации не является положительно определенным. Последняя условная дисперсия составляет 2.77556e-16"
1 ответ
Большие ковариационные матрицы легко могут быть численно сингулярными, даже в этом случае, когда они положительно определены по построению. Чтобы избежать этой проблемы и ненужных вычислений, в Stan обычно нужно объявить параметр как коэффициент Холецкого ковариационной или корреляционной матрицы (в этом случае вам также нужно объявить положительный вектор стандартных отклонений в параметрах). блок).
В этом случае вы бы назвали multi_normal_cholesky_lpdf функция, которая обусловливает фактор Холецкого ковариационной матрицы, а не саму ковариационную матрицу, которая, в свою очередь, может быть diag_pre_multiply вектора стандартных отклонений и коэффициент Холецкого корреляционной матрицы. Тем не менее, в этом случае вам нужно будет переключить приоры с обратного Wishart на lkj_corr_cholesky_lpdf для коэффициента Холецкого матрицы корреляции и все, что вы хотите (например, exponential_lpdf) для стандартных отклонений. Все это обсуждается в руководстве пользователя Stan.
Другой вариант - интегрировать ковариационные матрицы.