Цвет ветвей дендрограммы с использованием существующего столбца
У меня есть фрейм данных, который я пытаюсь кластеризовать. я использую hclust прямо сейчас. В моем фрейме данных есть FLAG столбец, который я хотел бы раскрасить дендрограмму. По полученной картине я пытаюсь выяснить сходство между различными FLAG категории. Мой фрейм данных выглядит примерно так:
FLAG ColA ColB ColC ColD
Я собираюсь на colA, colB, colC а также colD, Я хотел бы объединить их и раскрасить в соответствии с FLAG категории. Ex - красный цвет, если 1, синий, если 0 (у меня есть только две категории). Прямо сейчас я использую ванильную версию построения кластеров.
hc<-hclust(dist(data[2:5]),method='complete')
plot(hc)
Любая помощь в этом отношении будет принята с благодарностью.
2 ответа
Если вы хотите раскрасить ветви дендрограммы на основе определенной переменной, то следующий код (в основном взятый из справки по функции dendrapply) должен дать желаемый результат:
x<-1:100
dim(x)<-c(10,10)
groups<-sample(c("red","blue"), 10, replace=TRUE)
x.clust<-as.dendrogram(hclust(dist(x)))
local({
colLab <<- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
i <<- i+1
attr(n, "edgePar") <-
c(a$nodePar, list(col = mycols[i], lab.font= i%%3))
}
n
}
mycols <- groups
i <- 0
})
x.clust.dend <- dendrapply(x.clust, colLab)
plot(x.clust.dend)
Я думаю, что Архопала ответ хороший. Я взял на себя смелость сделать еще один шаг и добавил функцию assign_values_to_leaves_edgePar в пакет dendextend (начиная с версии 0.17.2, которая сейчас находится на github). Эта версия функции является более надежной и гибкой по сравнению с ответом Архопалы, поскольку:
- Это общая функция, которая может работать в разных задачах / настройках
- Функция может работать с другими параметрами edgePar (col, lwd, lty)
- Функция предлагает рециркуляцию частичных векторов и различные массажи предупреждений при необходимости.
Для установки пакета dendextend вы можете использовать install.packages('dendextend'), но для последней версии используйте следующий код:
require2 <- function (package, ...) {
if (!require(package)) install.packages(package); library(package)
}
## require2('installr')
## install.Rtools() # run this if you are using Windows and don't have Rtools installed (you must have it for devtools)
# Load devtools:
require2("devtools")
devtools::install_github('talgalili/dendextend')
Теперь, когда у нас установлен dendextend, вот второй взгляд на ответ Архопалы:
x<-1:100
dim(x)<-c(10,10)
set.seed(1)
groups<-sample(c("red","blue"), 10, replace=TRUE)
x.clust<-as.dendrogram(hclust(dist(x)))
x.clust.dend <- x.clust
x.clust.dend <- assign_values_to_leaves_edgePar(x.clust.dend, value = groups, edgePar = "col") # add the colors.
x.clust.dend <- assign_values_to_leaves_edgePar(x.clust.dend, value = 3, edgePar = "lwd") # make the lines thick
plot(x.clust.dend)
Вот результат:
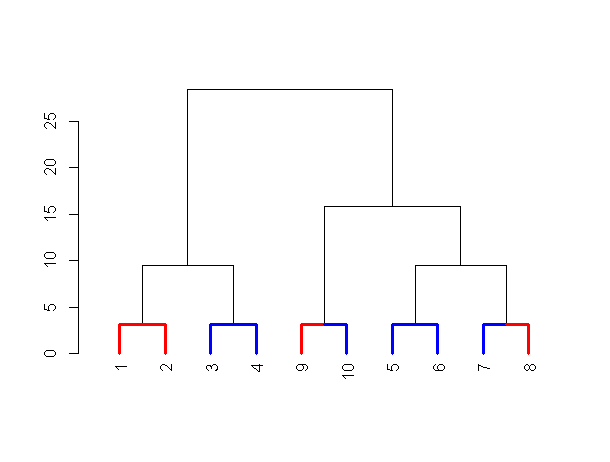
PS: я лично предпочитаю использовать каналы для этого типа кодирования (который даст тот же результат, что и выше, но легче читать):
x.clust <- x %>% dist %>% hclust %>% as.dendrogram
x.clust.dend <- x.clust %>%
assign_values_to_leaves_edgePar(value = groups, edgePar = "col") %>% # add the colors.
assign_values_to_leaves_edgePar(value = 3, edgePar = "lwd") # make the lines thick
plot(x.clust.dend)