Создание трехмерной каркасной модели коры головного мозга из шаблона мозга MNI с помощью Matlab с использованием MyRobustCrust
Я пытаюсь создать каркасную 3d-модель из шаблона мозга MNI, используя MyRobustCrust на matlab. Моя цель - определить координаты MNI на поверхности коры.
Для этого я следую инструкциям, перечисленным на этом сайте: http://soundsfromthenorth.com/?p=3
К сожалению, админ, похоже, больше не активен.
Следуя инструкциям сайта, я успешно извлек набор данных ("MNI152_T1_2mm_brain_mask.nii") из FSL. Однако, когда я запускаю MyRobustCrust, используя следующие данные (по указанию сайта), я получаю пустой график.
Ввод кода
>> [t,tnorm]=MyRobustCrust(p);
figure(1)
hold on
axis equal
h=trisurf(t,p(:,1),p(:,2),p(:,3),'facealpha',0.4,...
...'facecolor',[1 1 1],'edgecolor',[0.8 0.8 0.8]);
view(3);
axis vis3d
Вывод кода:
Added Shield: 0.0559 s
Delaunay Triangulation Time: 0.6515 s
Connectivity Time: 0.0796 s
Circumcenters Time: 0.0168 s
Warning: Brute continuation necessary
> In MyRobustCrust>Marking (line 380)
In MyRobustCrust (line 106)
Warning: 1000 th level was reached\n
> In MyRobustCrust>Marking (line 412)
In MyRobustCrust (line 106)
99.9941 % of Tetraedroms were checked
Walking Time: 0.2084 s
Manifold extraction Time: 0.3782 s
Total Time: 1.4809 s
view(3);
|
Error: Unbalanced or unexpected parenthesis or bracket.
Я, к сожалению, очень плохо знаком с нейровизуализацией и Matlab. У кого-нибудь есть какие-либо предложения относительно того, что я могу делать здесь не так? Сигнал об ошибке указывает на дисбаланс или неожиданные скобки в коде для MyRobustCrust или в данных?
1 ответ
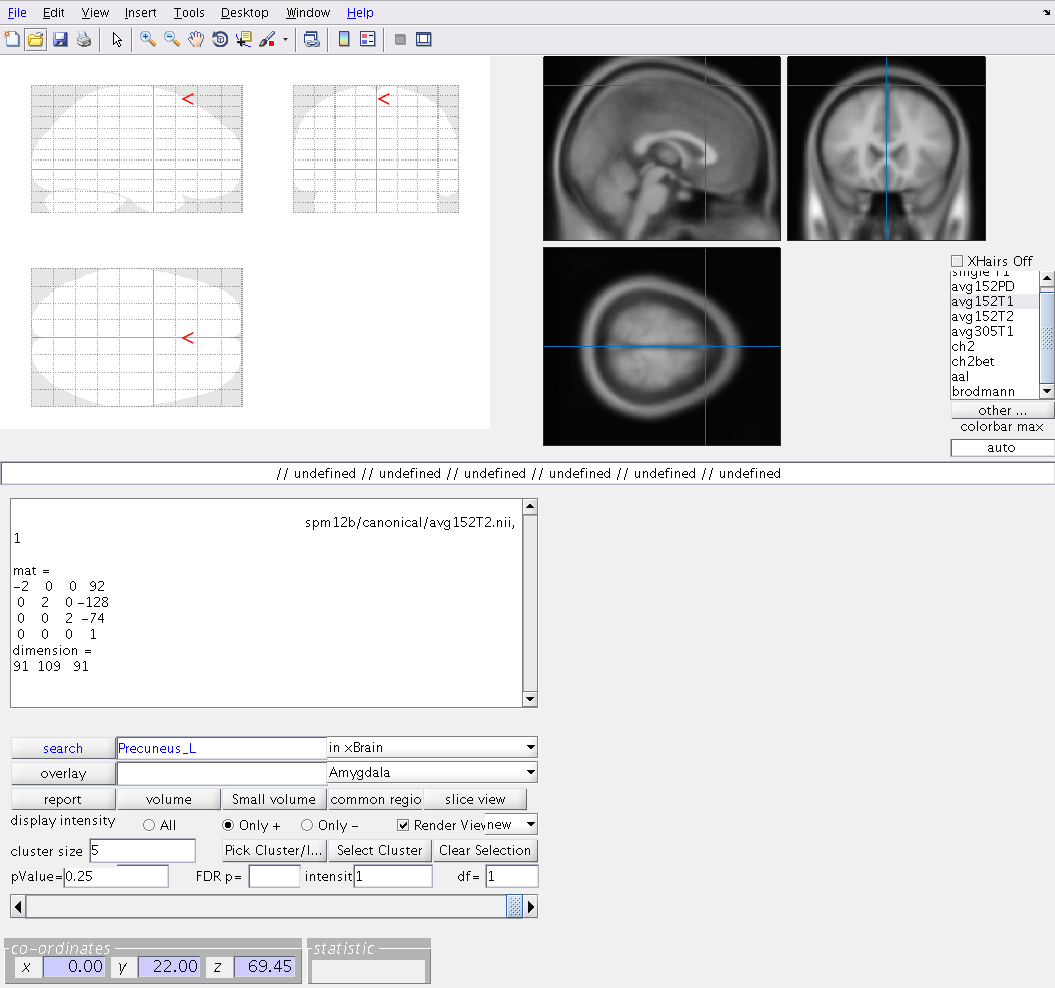
Надеюсь, я вас правильно понял, но мне кажется, что вы пытаетесь принять разметку поперечной плоскости (осевой плоскости) в координатах MNI.
Если это так, то, возможно, вам следует попробовать xjView (примечание: требуется SPM). Это графический интерфейс, который в основном используется для наложения контрастов на шаблон, среди тонов других функций (создание масок, создание таблиц статистики... и т. Д.). В вашем случае вы можете просто загрузить изображение MNI по умолчанию (avg152T1 или T2 из SPM) и щелкнуть вокруг 3-осевых представлений в правом верхнем углу; координаты MNI будут внизу слева.