GMM - логарифмическое правдоподобие не монотонно
Вчера я реализовал GMM (модель гауссовой смеси), используя алгоритм максимизации ожидания.
Как вы помните, он моделирует некое неизвестное распределение как смесь гауссиан, которые нам нужны для изучения его средних и дисперсионных значений, а также весов для каждого гауссиана.
это математика, стоящая за кодом (это не так уж сложно) http://mccormickml.com/2014/08/04/gaussian-mixture-models-tutorial-and-matlab-code/
это мой код:
import numpy as np
from scipy.stats import multivariate_normal
import matplotlib.pyplot as plt
#reference for this code is http://mccormickml.com/2014/08/04/gaussian-mixture-models-tutorial-and-matlab-code/
def expectation(data, means, covs, priors): #E-step. returns the updated probabilities
m = data.shape[0] #gets the data, means covariances and priors of all clusters
numOfClusters = priors.shape[0]
probabilities = np.zeros((m, numOfClusters))
for i in range(0, m):
for j in range(0, numOfClusters):
sum = 0
for l in range(0, numOfClusters):
sum += normalPDF(data[i, :], means[l], covs[l]) * priors[l, 0]
probabilities[i, j] = normalPDF(data[i, :], means[j], covs[j]) * priors[j, 0] / sum
return probabilities
def maximization(data, probabilities): #M-step. this updates the means, covariances, and priors of all clusters
m, n = data.shape
numOfClusters = probabilities.shape[1]
means = np.zeros((numOfClusters, n))
covs = np.zeros((numOfClusters, n, n))
priors = np.zeros((numOfClusters, 1))
for i in range(0, numOfClusters):
priors[i, 0] = np.sum(probabilities[:, i]) / m #update priors
for j in range(0, m): #update means
means[i] += probabilities[j, i] * data[j, :]
vec = np.reshape(data[j, :] - means[i, :], (n, 1))
covs[i] += probabilities[j, i] * np.dot(vec, vec.T) #update covs
means[i] /= np.sum(probabilities[:, i])
covs[i] /= np.sum(probabilities[:, i])
return [means, covs, priors]
def normalPDF(x, mean, covariance): #this is simply multivariate normal pdf
n = len(x)
mean = np.reshape(mean, (n, ))
x = np.reshape(x, (n, ))
var = multivariate_normal(mean=mean, cov=covariance,)
return var.pdf(x)
def initClusters(numOfClusters, data): #initialize all the gaussian clusters (means, covariances, priors
m, n = data.shape
means = np.zeros((numOfClusters, n))
covs = np.zeros((numOfClusters, n, n))
priors = np.zeros((numOfClusters, 1))
initialCovariance = np.cov(data.T)
for i in range(0, numOfClusters):
means[i] = np.random.rand(n) #the initial mean for each gaussian is chosen randomly
covs[i] = initialCovariance #the initial covariance of each cluster is the covariance of the data
priors[i, 0] = 1.0 / numOfClusters #the initial priors are uniformly distributed.
return [means, covs, priors]
def logLikelihood(data, probabilities): #data is our data. probabilities[i, j] = k means probability example i belongs in cluster j is 0 < k < 1
m = data.shape[0] #num of examples
examplesByCluster = np.zeros((m, 1))
for i in range(0, m):
examplesByCluster[i, 0] = np.argmax(probabilities[i, :])
examplesByCluster = examplesByCluster.astype(int) #examplesByCluster[i] = j means that example i belongs in cluster j
result = 0
for i in range(0, m):
result += np.log(probabilities[i, examplesByCluster[i, 0]]) #example i belongs in cluster examplesByCluster[i, 0]
return result
m = 2000 #num of training examples
n = 8 #num of features for each example
data = np.random.rand(m, n)
numOfClusters = 2 #num of gaussians
numIter = 30 #num of iterations of EM
cost = np.zeros((numIter, 1))
[means, covs, priors] = initClusters(numOfClusters, data)
for i in range(0, numIter):
probabilities = expectation(data, means, covs, priors)
[means, covs, priors] = maximization(data, probabilities)
cost[i, 0] = logLikelihood(data, probabilities)
plt.plot(cost)
plt.show()
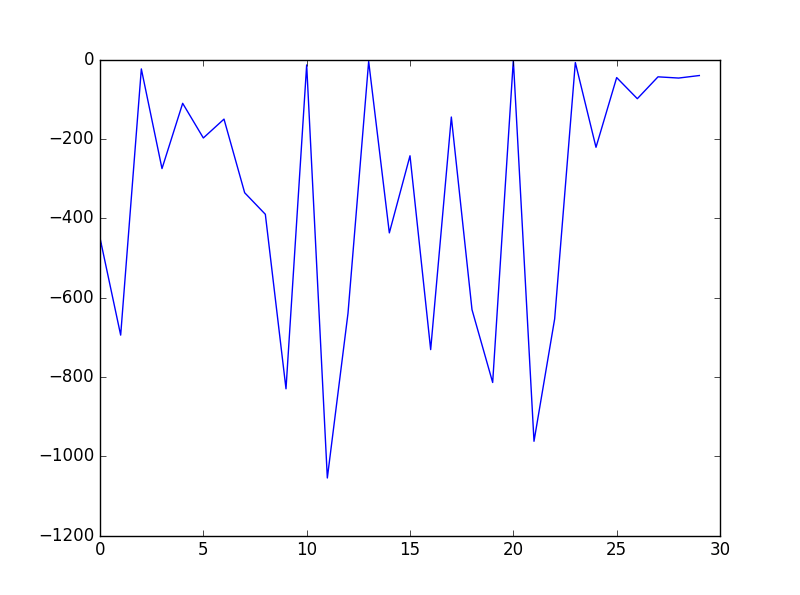
проблема в том, что логарифмическая вероятность ведет себя странно. Я ожидаю, что это будет монотонным увеличением. Но это не так.
Например, с 2000 примерами 8 объектов с 3 гауссовыми кластерами логарифмическое правдоподобие выглядит следующим образом (30 итераций) -
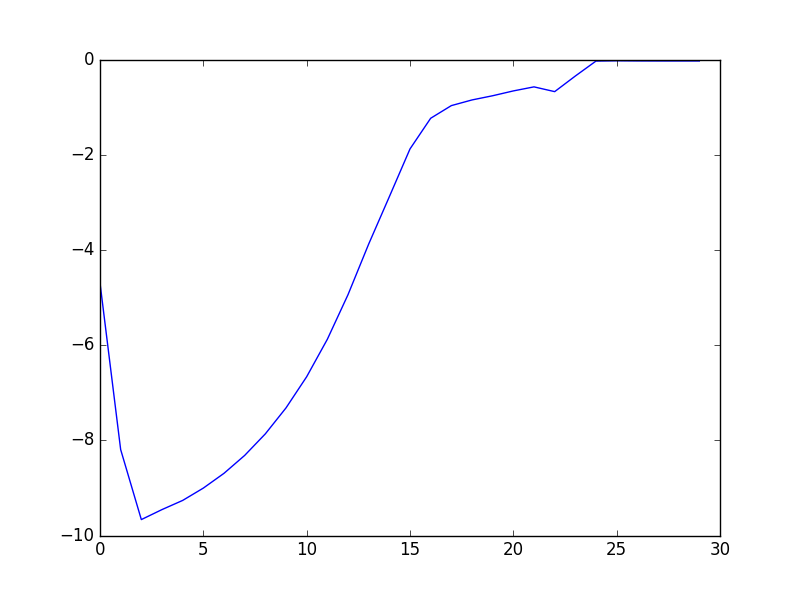
Так что это очень плохо. Но в других тестах, которые я запускал, например, один тест с 15 примерами из 2 функций и 2 кластеров, логарифмическая вероятность такова:
Лучше, но все же не идеально.
Почему это происходит и как я могу это исправить?
1 ответ
Проблема заключается в шаге максимизации.
Код использует means для расчета covs, Однако это делается в том же цикле, прежде чем делить means по сумме вероятностей.
Это приводит к взрыву предполагаемых ковариаций.
Вот предлагаемое исправление:
def maximization(data, probabilities): #M-step. this updates the means, covariances, and priors of all clusters
m, n = data.shape
numOfClusters = probabilities.shape[1]
means = np.zeros((numOfClusters, n))
covs = np.zeros((numOfClusters, n, n))
priors = np.zeros((numOfClusters, 1))
for i in range(0, numOfClusters):
priors[i, 0] = np.sum(probabilities[:, i]) / m #update priors
for j in range(0, m): #update means
means[i] += probabilities[j, i] * data[j, :]
means[i] /= np.sum(probabilities[:, i])
for i in range(0, numOfClusters):
for j in range(0, m): #update means
vec = np.reshape(data[j, :] - means[i, :], (n, 1))
covs[i] += probabilities[j, i] * np.multiply(vec, vec.T) #update covs
covs[i] /= np.sum(probabilities[:, i])
return [means, covs, priors]
И полученная функция стоимости (200 точек данных, 4 функции): 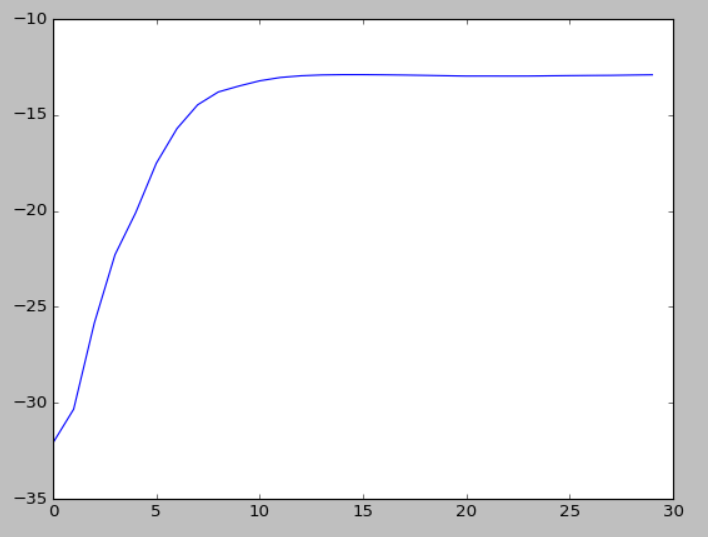
РЕДАКТИРОВАТЬ: я был убежден, что эта ошибка была единственной проблемой в коде, однако, запустив несколько дополнительных примеров, я все еще иногда вижу немонотонное поведение (хотя и менее ошибочное, чем раньше). Так что это, кажется, только часть проблемы.
РЕДАКТИРОВАТЬ 2: Была другая проблема в вычислении ковариации: умножение вектора должно быть поэлементным, а не точечным произведением - имейте в виду, что результатом должен быть вектор. Результаты кажутся постоянно монотонно растущими в настоящее время.