Связь между координатами графика дендрограммы и узлами кластера в scipy
Я ищу способ получить координаты точки кластера на графике дендрограммы, основываясь на его возвращении ClusterNode по to_tree.
Использование scipy для построения дендограммы из таких данных, как:
X = data
Y = pdist(X)
Z = linkage(Y)
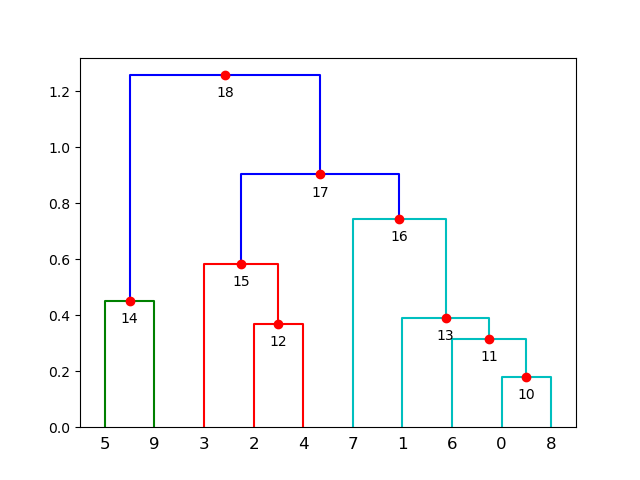
dend = dendrogram(Z)
rootnode, nodesList = to_tree(Z, rd=True)
Что я хотел бы сделать, это построить функцию get_coords(somClusterNode) что бы вернуть кортеж (x, y) указание положения узла на графике.
Благодаря этому ответу мне удалось выяснить, как получить позицию из возвращаемых значений дендрограммы, например:
i, d = list(zip(dend['icoord'], dend['dcoord']))[-1]
x = 0.5 * sum(i[1:3])
y = d[1]
plt.plot(x, y, 'ro')
Но я могу выяснить связь между порядком расположения узлов и списком icoord/dcoord порядок для того, чтобы сопоставить одно с другим.
У вас есть идеи, где я могу искать?
Спасибо за вашу помощь!
1 ответ
Каждая дендрограмма отображается только на одно дерево ClusterNodes, но любое дерево ClusterNodes может отображаться на бесконечное количество дендрограмм. Следовательно, ваше отображение от идентификатора узла до (x,y) позиций должно быть просто другим полем в вашей структуре данных дендрограммы, а не функцией ClusterNode. Вместо определения функции get_coordsЯ, следовательно, добавляет словарь dend который сопоставляет идентификаторы узлов с (x,y) координатами. Вы можете получить доступ к позициям с
x,y = dend['node_id_to_coord'][node_id] # node_id is an integer as returned by ClusterNode.id
Код:
import numpy as np
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import linkage, dendrogram, to_tree
from scipy.spatial.distance import pdist
# create some random data
X = np.random.rand(10, 3)
# get dendrogram
Z = linkage(pdist(X), method="ward")
dend = dendrogram(Z)
# ----------------------------------------
# get leave coordinates, which are at y == 0
def flatten(l):
return [item for sublist in l for item in sublist]
X = flatten(dend['icoord'])
Y = flatten(dend['dcoord'])
leave_coords = [(x,y) for x,y in zip(X,Y) if y==0]
# in the dendogram data structure,
# leave ids are listed in ascending order according to their x-coordinate
order = np.argsort([x for x,y in leave_coords])
id_to_coord = dict(zip(dend['leaves'], [leave_coords[idx] for idx in order])) # <- main data structure
# ----------------------------------------
# get coordinates of other nodes
# this should work but doesn't:
# # traverse tree from leaves upwards and populate mapping ID -> (x,y);
# # use linkage matrix to traverse the tree optimally
# # (each row in the linkage matrix corresponds to a row in dend['icoord'] and dend['dcoord'])
# root_node, node_list = to_tree(Z, rd=True)
# for ii, (X, Y) in enumerate(zip(dend['icoord'], dend['dcoord'])):
# x = (X[1] + X[2]) / 2
# y = Y[1] # or Y[2]
# node_id = ii + len(dend['leaves'])
# id_to_coord[node_id] = (x, y)
# so we need to do it the hard way:
# map endpoint of each link to coordinates of parent node
children_to_parent_coords = dict()
for i, d in zip(dend['icoord'], dend['dcoord']):
x = (i[1] + i[2]) / 2
y = d[1] # or d[2]
parent_coord = (x, y)
left_coord = (i[0], d[0])
right_coord = (i[-1], d[-1])
children_to_parent_coords[(left_coord, right_coord)] = parent_coord
# traverse tree from leaves upwards and populate mapping ID -> (x,y)
root_node, node_list = to_tree(Z, rd=True)
ids_left = range(len(dend['leaves']), len(node_list))
while len(ids_left) > 0:
for ii, node_id in enumerate(ids_left):
node = node_list[node_id]
if (node.left.id in id_to_coord) and (node.right.id in id_to_coord):
left_coord = id_to_coord[node.left.id]
right_coord = id_to_coord[node.right.id]
id_to_coord[node_id] = children_to_parent_coords[(left_coord, right_coord)]
ids_left = [node_id for node_id in range(len(node_list)) if not node_id in id_to_coord]
# plot result on top of dendrogram
ax = plt.gca()
for node_id, (x, y) in id_to_coord.iteritems():
if not node_list[node_id].is_leaf():
ax.plot(x, y, 'ro')
ax.annotate(str(node_id), (x, y), xytext=(0, -8),
textcoords='offset points',
va='top', ha='center')
dend['node_id_to_coord'] = id_to_coord
Есть еще один способ сделать это:
Идентификаторы дендограммы, по-видимому, генерируются обратным обходом дерева справа налево. Это позволяет нам построить перевод из node.id к индексу icoord а также dcoord следующее:
def rl_traversal(node):
# skipping leaves
if not node.is_leaf():
yield node.id
yield from rl_traversal(node.right)
yield from rl_traversal(node.left)
id_map = dict(zip( rl_traversal(root), reversed(range(root.get_count()-1))) ))
# id_map[node_id] = dendogram_id
Координаты узла могут быть получены с помощью dendo['icoord'][id_map[node_id]]