Как построить и аннотировать иерархические кластерные дендрограммы в Scipy/ Matplotlib
Я использую dendrogram от scipy построить иерархическую кластеризацию, используя matplotlib следующее:
mat = array([[1, 0.5, 0.9],
[0.5, 1, -0.5],
[0.9, -0.5, 1]])
plt.subplot(1,2,1)
plt.title("mat")
dist_mat = mat
linkage_matrix = linkage(dist_mat,
"single")
print "linkage2:"
print linkage(1-dist_mat, "single")
dendrogram(linkage_matrix,
color_threshold=1,
labels=["a", "b", "c"],
show_leaf_counts=True)
plt.subplot(1,2,2)
plt.title("1 - mat")
dist_mat = 1 - mat
linkage_matrix = linkage(dist_mat,
"single")
dendrogram(linkage_matrix,
color_threshold=1,
labels=["a", "b", "c"],
show_leaf_counts=True)
Мои вопросы: во-первых, почему mat а также 1-mat дать одинаковые кластеризации здесь? и во-вторых, как я могу аннотировать расстояние вдоль каждой ветви дерева, используя dendrogram так что расстояния между парами узлов можно сравнить?
наконец, кажется, что show_leaf_counts флаг игнорируется, есть ли способ включить его, чтобы отображалось количество объектов в каждом классе? Благодарю.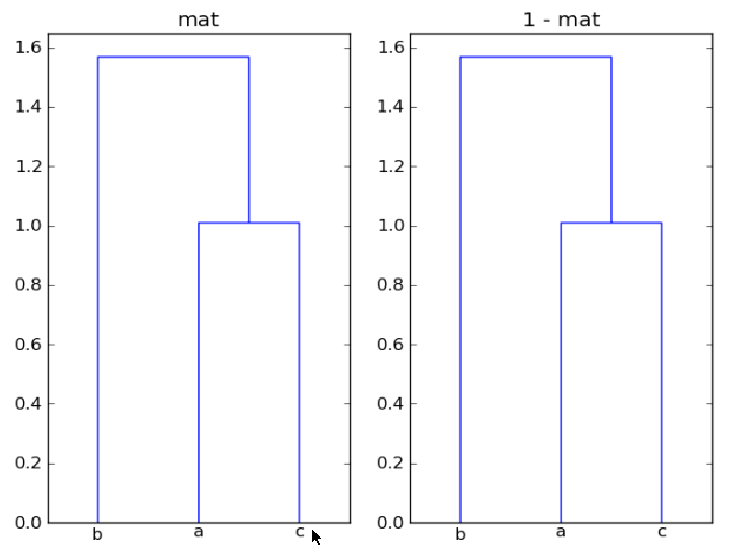
2 ответа
Вход в linkage() это либо массив n x m, представляющий n точек в m-мерном пространстве, либо одномерный массив, содержащий сжатую матрицу расстояний. В вашем примере mat 3 х 3, так что вы кластеризуете три трехмерные точки. Кластеризация основана на расстоянии между этими точками.
Почему мат и 1-мат дают одинаковую кластеризацию здесь?
Массивы mat а также 1-mat производить ту же кластеризацию, потому что кластеризация основана на расстояниях между точками, а не на отражении (-mat) ни перевод (mat + offset) из всего набора данных изменить относительные расстояния между точками.
Как можно аннотировать расстояние вдоль каждой ветви дерева с помощью дендрограммы, чтобы можно было сравнивать расстояния между парами узлов?
В приведенном ниже коде я показываю, как можно использовать данные, возвращаемые дендрограммой, для маркировки горизонтальных сегментов диаграммы с соответствующим расстоянием. Значения, связанные с ключами icoord а также dcoord дать координаты x и y каждого трехсегментного перевернутого U на рисунке. В augmented_dendrogram Эти данные используются для добавления метки расстояния (т. е. значения y) каждого сегмента горизонтальной линии в дендрограмме.
from scipy.cluster.hierarchy import dendrogram
import matplotlib.pyplot as plt
def augmented_dendrogram(*args, **kwargs):
ddata = dendrogram(*args, **kwargs)
if not kwargs.get('no_plot', False):
for i, d in zip(ddata['icoord'], ddata['dcoord']):
x = 0.5 * sum(i[1:3])
y = d[1]
plt.plot(x, y, 'ro')
plt.annotate("%.3g" % y, (x, y), xytext=(0, -8),
textcoords='offset points',
va='top', ha='center')
return ddata
Для тебя mat массив, расширенная дендрограмма
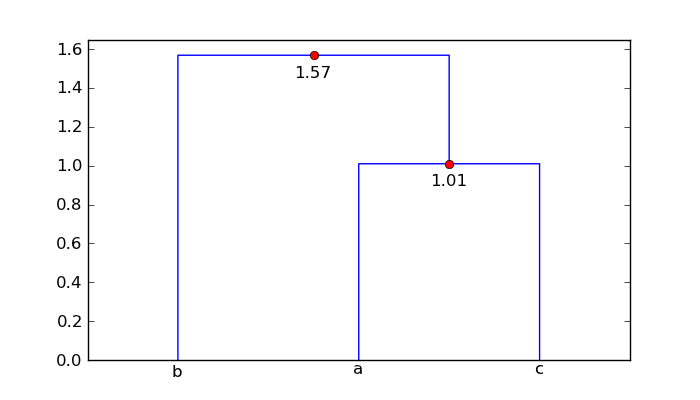
Таким образом, точки 'a' и 'c' находятся на расстоянии 1,01 единицы, а точка 'b' составляет 1,57 единицы от кластера ['a', 'c'].
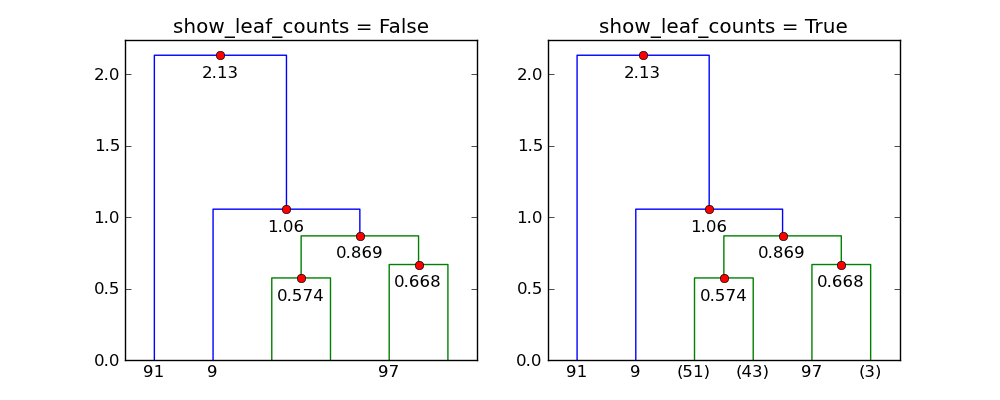
Кажется, что show_leaf_counts флаг игнорируется, есть ли способ включить его, чтобы отображалось количество объектов в каждом классе?
Флаг show_leaf_counts применяется только тогда, когда не все исходные точки данных отображаются в виде листьев. Например, когда trunc_mode = "lastp" только последний p узлы показаны.
Вот пример с 100 баллами:
import numpy as np
from scipy.cluster.hierarchy import linkage
import matplotlib.pyplot as plt
from augmented_dendrogram import augmented_dendrogram
# Generate a random sample of `n` points in 2-d.
np.random.seed(12312)
n = 100
x = np.random.multivariate_normal([0, 0], np.array([[4.0, 2.5], [2.5, 1.4]]),
size=(n,))
plt.figure(1, figsize=(6, 5))
plt.clf()
plt.scatter(x[:, 0], x[:, 1])
plt.axis('equal')
plt.grid(True)
linkage_matrix = linkage(x, "single")
plt.figure(2, figsize=(10, 4))
plt.clf()
plt.subplot(1, 2, 1)
show_leaf_counts = False
ddata = augmented_dendrogram(linkage_matrix,
color_threshold=1,
p=6,
truncate_mode='lastp',
show_leaf_counts=show_leaf_counts,
)
plt.title("show_leaf_counts = %s" % show_leaf_counts)
plt.subplot(1, 2, 2)
show_leaf_counts = True
ddata = augmented_dendrogram(linkage_matrix,
color_threshold=1,
p=6,
truncate_mode='lastp',
show_leaf_counts=show_leaf_counts,
)
plt.title("show_leaf_counts = %s" % show_leaf_counts)
plt.show()
Это точки в наборе данных:
С p=6 а также trunc_mode="lastp", dendrogram показывает только "вершину" дендрограммы. Ниже показано влияние show_leaf_counts,
Я думаю, что есть пара недоразумений относительно использования функций, которые вы пытаетесь использовать. Вот полностью рабочий фрагмент кода, чтобы проиллюстрировать мои идеи:
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram, linkage
from numpy import array
import numpy as np
mat = array([184, 222, 177, 216, 231,
45, 123, 128, 200,
129, 121, 203,
46, 83,
83])
dist_mat = mat
linkage_matrix = linkage(dist_mat, 'single')
print linkage_matrix
plt.figure(101)
plt.subplot(1, 2, 1)
plt.title("ascending")
dendrogram(linkage_matrix,
color_threshold=1,
truncate_mode='lastp',
labels=array(['a', 'b', 'c', 'd', 'e', 'f']),
distance_sort='ascending')
plt.subplot(1, 2, 2)
plt.title("descending")
dendrogram(linkage_matrix,
color_threshold=1,
truncate_mode='lastp',
labels=array(['a', 'b', 'c', 'd', 'e', 'f']),
distance_sort='descending')
def make_fake_data():
amp = 1000.
x = []
y = []
for i in range(0, 10):
s = 20
x.append(np.random.normal(30, s))
y.append(np.random.normal(30, s))
for i in range(0, 20):
s = 2
x.append(np.random.normal(150, s))
y.append(np.random.normal(150, s))
for i in range(0, 10):
s = 5
x.append(np.random.normal(-20, s))
y.append(np.random.normal(50, s))
plt.figure(1)
plt.title('fake data')
plt.scatter(x, y)
d = []
for i in range(len(x) - 1):
for j in range(i+1, len(x) - 1):
d.append(np.sqrt(((x[i]-x[j])**2 + (y[i]-y[j])**2)))
return d
mat = make_fake_data()
plt.figure(102)
plt.title("Three Clusters")
linkage_matrix = linkage(mat, 'single')
print "three clusters"
print linkage_matrix
dendrogram(linkage_matrix,
truncate_mode='lastp',
color_threshold=1,
show_leaf_counts=True)
plt.show()
Прежде всего, вычисление m -> m - 1 действительно не изменило ваш результат, поскольку матрица расстояний, которая в основном описывает относительные расстояния между всеми уникальными парами, не изменилась в вашем конкретном случае. (В приведенном выше примере кода все расстояния являются евклидовыми, поэтому все они положительны и согласованы с точками на двухмерной плоскости.)
Что касается вашего второго вопроса, вам, вероятно, нужно развернуть свою собственную процедуру аннотации, чтобы делать то, что вы хотите, так как я не думаю, что дендромграм изначально поддерживает это...
В последнем вопросе show_leaf_counts кажется работает только при попытке отобразить не-одиночные конечные узлы с параметром truncate_mode='lastp'. В основном листья сгруппированы так близко друг к другу, что их нелегко увидеть. Таким образом, у вас есть возможность просто показать лист, но у вас есть возможность показать (в скобках), сколько их сгруппировано в этом листе.
Надеюсь это поможет.