Создание 2d изображений молекул из данных FTP PubChem
Вместо того, чтобы сканировать веб-сайт PubChem, я бы предпочел быть хорошим и генерировать изображения локально с ftp-сайта PubChem:
ftp://ftp.ncbi.nih.gov/pubchem/specifications/
Единственная проблема заключается в том, что я ограничен OSX и Linux, и я не могу найти способ программно генерировать 2d изображения, которые они имеют на своем сайте. Смотрите этот пример:
https://pubchem.ncbi.nlm.nih.gov/compound/6
Под заголовком "2D Структура" у нас есть это изображение здесь:
https://pubchem.ncbi.nlm.nih.gov/image/imgsrv.fcgi?cid=6&t=l
Это то, что я пытаюсь создать.
2 ответа
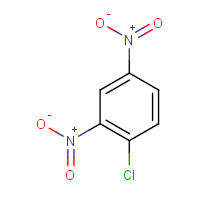
Если вы хотите что-то нестандартное, я бы предложил использовать molconvert от ChevAxon's Marvin ( https://www.chemaxon.com/products/marvin/), который является бесплатным для академиков. Его можно легко использовать из командной строки, и он поддерживает множество форматов ввода и вывода. Так что для вашего примера это будет:
molconvert "png" -s "C1=CC(=C(C=C1[N+](=O)[O-])[N+](=O)[O-])Cl" -o cdnb.png
В результате на следующем изображении:
Он также позволяет устанавливать такие параметры, как ширина, высота, качество, цвет фона и так далее.
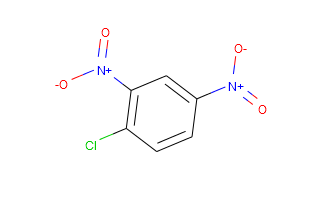
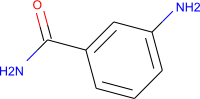
Однако, если вы программист, я бы определенно рекомендовал RDKit. Следует код, который генерирует изображения для пары соединений, представленных в виде улыбок.
from rdkit import Chem
from rdkit.Chem import Draw
ms_smis = [["C1=CC(=C(C=C1[N+](=O)[O-])[N+](=O)[O-])Cl", "cdnb"],
["C1=CC(=CC(=C1)N)C(=O)N", "3aminobenzamide"]]
ms = [[Chem.MolFromSmiles(x[0]), x[1]] for x in ms_smis]
for m in ms: Draw.MolToFile(m[0], m[1] + ".svg", size=(800, 800))
Это дает вам следующие изображения:
Так что я также написал ребятам из PubChem по электронной почте, и они очень быстро ответили мне:
Единственный доступ к изображениям, который у нас есть, - через службу загрузки: https://pubchem.ncbi.nlm.nih.gov/pc_fetch/pc_fetch.cgi
Вы можете запросить до 50000 изображений одновременно.
Что лучше, чем я ожидал, но все же не удивительно, поскольку требует загрузки вещей, которые я теоретически мог бы генерировать локально. Так что я оставляю этот вопрос открытым, пока какая-то добрая душа не напишет библиотеку с открытым исходным кодом, чтобы сделать то же самое.
Редактировать:
Я полагаю, что с тем же успехом могу спасти людей, если они будут делать то же самое, что и я. Я создал Ruby Gem на механизированной основе для автоматизации загрузки изображений. Пожалуйста, будьте добры к своим серверам и загружайте только то, что вам нужно.
https://github.com/zachaysan/pubchem
gem install pubchem
Вариант с открытым исходным кодом - Indigo Toolkit, который также содержит предварительно скомпилированные пакеты для Linux, Windows и MacOS и языковые привязки для библиотек Python, Java, .NET и C. Я выбрал бету 1.4.0.
У меня был такой же интерес, как и у вас, в преобразовании SMILES в 2D-структуры, и я адаптировал свой Python для ответа на ваш вопрос и сбора информации о времени. Он использует загрузку PubChem FTP (Compound/Extras) CID-SMILES.gz. Следующий сценарий представляет собой реализацию локального преобразователя SMILES в 2D-структуру, который считывает диапазон строк из файла PubChem CID-SMILES изомерных SMILES (который содержит более 102 миллионов составных записей) и преобразует SMILES в изображения PNG 2D-конструкции. В трех тестах с преобразованием 1000 SMILES в структуру потребовалось 35, 50 и 60 секунд для преобразования 1000 SMILES при смещении строки файла 0, 100 000 и 10 000 000 на моем ноутбуке с Windows 10 (процессор Intel i7-7500U, 2,70 ГГц) с твердотельным накопителем и запущенным Python 3.7.4. Общий размер 3000 файлов составил 100 МБ.
from indigo import *
from indigo.renderer import *
import subprocess
import datetime
def timerstart():
# start timer and print time, return start time
start = datetime.datetime.now()
print("Start time =", start)
return start
def timerstop(start):
# end timer and print time and elapsed time, return elapsed time
endtime = datetime.datetime.now()
elapsed = endtime - start
print("End time =", endtime)
print("Elapsed time =", elapsed)
return elapsed
numrecs = 1000
recoffset = 0 # 10000000 # record offset
starttime = timerstart()
indigo = Indigo()
renderer = IndigoRenderer(indigo)
# set render options
indigo.setOption("render-atom-color-property", "color")
indigo.setOption("render-coloring", True)
indigo.setOption("render-comment-position", "bottom")
indigo.setOption("render-comment-offset", "20")
indigo.setOption("render-background-color", 1.0, 1.0, 1.0)
indigo.setOption("render-output-format", "png")
# set data path (including data file) and output file path
datapath = r'../Download/CID-SMILES'
pngpath = r'./2D/'
# read subset of rows from data file
mycmd = "head -" + str(recoffset+numrecs) + " " + datapath + " | tail -" + str(numrecs)
print(mycmd)
(out, err) = subprocess.Popen(mycmd, stdout=subprocess.PIPE, shell=True).communicate()
lines = str(out.decode("utf-8")).split("\n")
count = 0
for line in lines:
try:
cols = line.split("\t") # split on tab
key = cols[0] # cid in cols[0]
smiles = cols[1] # smiles in cols[1]
mol = indigo.loadMolecule(smiles)
s = "CID=" + key
indigo.setOption("render-comment", s)
#indigo.setOption("render-image-size", 200, 250)
#indigo.setOption("render-image-size", 400, 500)
renderer.renderToFile(mol, pngpath + key + ".png")
count += 1
except:
print("Error processing line after", str(count), ":", line)
pass
elapsedtime = timerstop(starttime)
print("Converted", str(count), "SMILES to PNG")