Объединить филогенетическое дерево с графиком x,y
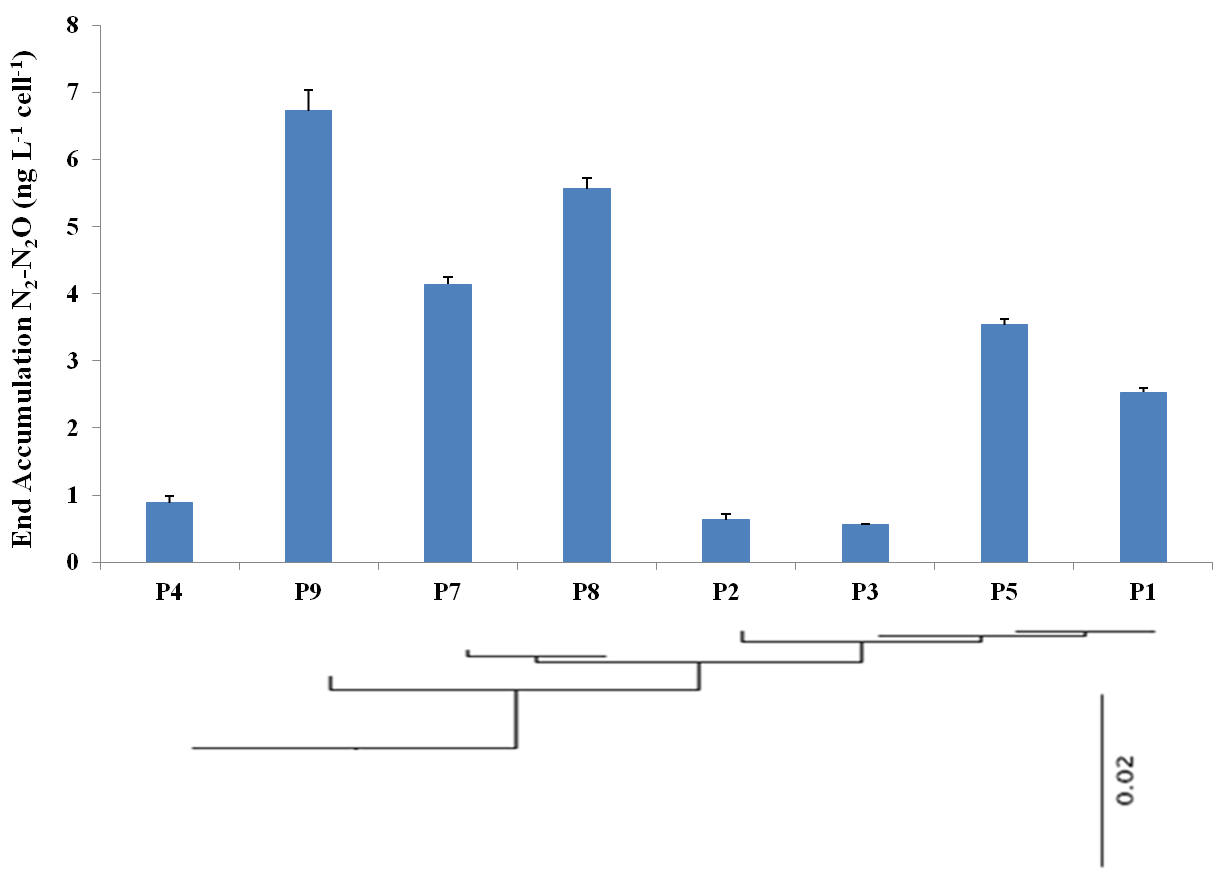
Я пытаюсь расположить филогенетическое дерево на графике, показывающем физиологические данные для набора родственных организмов. Нечто подобное на картинке ниже. Это было собрано в PowerPoint из 2 отдельных графиков. Я предполагаю, что это выполнит работу, но я надеялся создать единственное изображение, которое, я думаю, будет легче отформатировать в документ. Я могу создать график, который я хочу, используя ggplot2, и импортировать дерево, используя ape. Я думал, что должен быть способ сохранить дерево как графический объект, а затем упорядочить его по графику, используя функцию gridarrange в gridExtra. Проблема в том, что ape не позволит мне сохранить дерево как графический объект, например,
p2<-plot(tree, dir = "u", show.tip.label = FALSE)
просто строит дерево и когда вы вызываете p2, он просто дает список аргументов. Мне интересно, есть ли у кого-нибудь советы.
Спасибо!
2 ответа
Я не уверен, что это будет работать с gtable из CRAN
require(ggplot2)
require(gridBase)
require(gtable)
p <- qplot(1,1)
g <- ggplotGrob(p)
g <- gtable_add_rows(g, unit(2,"in"), nrow(g))
g <- gtable_add_grob(g, rectGrob(),
t = 7, l=4, b=7, r=4)
grid.newpage()
grid.draw(g)
#grid.force()
#grid.ls(grobs=F, viewports=T)
seekViewport("layout.7-4-7-4")
par(plt=gridPLT(), new=TRUE)
plot(rtree(10), "c", FALSE, direction = "u")
upViewport()
Во-первых, я хотел бы поблагодарить baptiste за ВСЕ его множественные ответы, которые решили большинство моих проблем с ggplot2.
во-вторых, у меня был похожий вопрос, который заключался в том, чтобы включить дерево из обезьяны в тепловую карту, полученную с помощью ggplot2. Баптист сделал мой день, и хотя моя упрощенная версия могла помочь. Я использовал только то, что было полезно для меня (убрав добавление gg_rows).
library(ape)
tr <- read.tree("mytree.tree")
# heat is the heatmap ggplot, using geom_tile
g <- ggplotGrob(heat)
grid.newpage()
grid.draw(g)
# use oma to reduce the tree so it fits
par(new = TRUE, oma = c(5, 4, 5, 38))
plot(tr)
nodelabels(tr$node.label, cex = 1, frame = "none", col = "black", adj = c(-0.3, 0.5))
add.scale.bar()
# use dev.copy2pdf and not ggsave
dev.copy2pdf(file = "heatmap_prob.pdf")
результат здесь 