Биоинформатика: как сделать контраст Лиммы с 3 группами (случай / контроль, возраст и пол)
Я студент, и у меня есть сомнения относительно того, как использовать Limma для сравнения дифференциальной экспрессии генов, оценивая мои три переменные: болезнь (случай / контроль), пол (мужчина / женщина) и возраст.
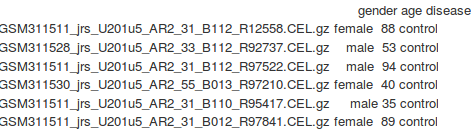
Мои данные таковы: (три столбца с полом, возрастом и заболеванием)
головка (PDATA (GSE))
Данные здесь:
Я знаю, строю свой эксперимент над моей переменной болезнью:
design= model.matrix(~ pData(gse)[,"disease"])
colnames(design)= c("control", "case")
fit= lmFit(gse, fesign)
fit1= eBayes(fit)
topTable(fit1, coef=2, adjust="BH", number= as.numeric(dim(gse)[1]))
Но как я могу сделать контрастную Лимму с моими 3 переменными? Спасибо за помощь!
age= pData(gse)[,"age"]
gender= pData(gse)[,"gender"]
disease= pData(gse)[,"disease"]
age.group= ifelse(age>40,"over40", "under40")
design = model.matrix(~0 + disease + gender + gender:age)
contr.matrix <- makeContrasts( **???**