rdkit: Как показать количество атомов в молекулярной среде
Здравствуйте! Я пытался использовать пакет rdkit, чтобы завершить работу по отображению чисел атомов молекулы в Jupyter Notebook, "import IPython.core.interactiveshell" и "import InteractiveShell", а также "из rdkit.Chem.Draw импортировать DrawingOptions", затем Я использовал "DrawingOptions.includeAtomNumbers=True" для его работы, но в результате индекс атомов вообще не отображался. Я не знаю, в чем причина того, что число атомов не показывалось. Поэтому я хочу порадовать вас дать подходящий ответ. Большое спасибо!
3 ответа
Есть три способа показать номера атомов в молекуле.
from rdkit import Chem
from rdkit.Chem.Draw import IPythonConsole
def show_atom_number(mol, label):
for atom in mol.GetAtoms():
atom.SetProp(label, str(atom.GetIdx()+1))
return mol
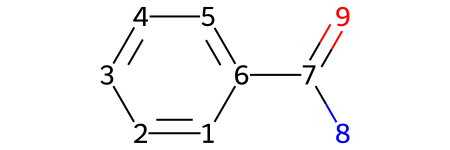
1. Вместо атомов
mol = Chem.MolFromSmiles('c1ccccc(C(N)=O)1')
show_atom_number(mol, 'atomLabel')
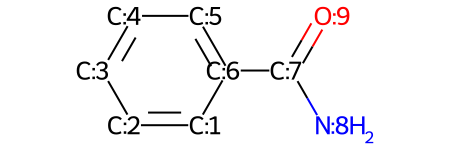
2. Наряду с атомами
mol = Chem.MolFromSmiles('c1ccccc(C(N)=O)1')
show_atom_number(mol, 'molAtomMapNumber')
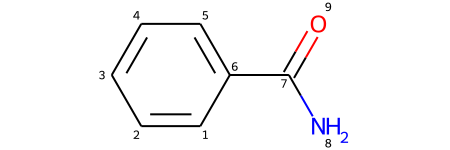
3. Поверх атомов
mol = Chem.MolFromSmiles('c1ccccc(C(N)=O)1')
show_atom_number(mol, 'atomNote')
Если вы хотите изменить отображаемые числа, измените часть
str(atom.GetIdx()+1) согласно вашим требованиям.
Это работает для меня в ноутбуке Jupyter:
from rdkit import Chem
from rdkit.Chem.Draw import IPythonConsole
from rdkit.Chem import Draw
smiles = 'O=C(C)Oc1ccccc1C(=O)O'
mol = Chem.MolFromSmiles(smiles)
Draw.MolToImage(mol, includeAtomNumbers=True)
В версии 2020.03.2.0 вы могли попробовать
from rdkit.Chem.Draw import rdMolDraw2D
from rdkit import Chem
mol = Chem.MolFromSmiles('c1ccccc1O')
d = rdMolDraw2D.MolDraw2DCairo(250, 200)
d.drawOptions().addAtomIndices = True
d.DrawMolecule(mol)
d.FinishDrawing()
with open('atom_annotation_1.png', 'wb') as f:
f.write(d.GetDrawingText())