R: Тьюки posthoc тестирует для nnet multinom многочленовой подгонки, чтобы проверить общие различия в многочленовом распределении
Я установил мутиномную модель, используя nnet"s multinom Использование функции (в данном случае по данным, отдавающим предпочтение в рационе мужского и женского пола и разного размера классов аллигаторов в разных озерах):
data=read.csv("https://www.dropbox.com/s/y9elunsbv74p2h6/alligator.csv?dl=1")
head(data)
id size sex lake food
1 1 <2.3 male hancock fish
2 2 <2.3 male hancock fish
3 3 <2.3 male hancock fish
4 4 <2.3 male hancock fish
5 5 <2.3 male hancock fish
6 6 <2.3 male hancock fish
library(nnet)
fit=multinom(food~lake+sex+size, data = data, Hess = TRUE)
Общее значение моих факторов я могу использовать
library(car)
Anova(fit, type="III") # type III tests
Analysis of Deviance Table (Type III tests)
Response: food
LR Chisq Df Pr(>Chisq)
lake 50.318 12 1.228e-06 ***
sex 2.215 4 0.696321
size 17.600 4 0.001477 **
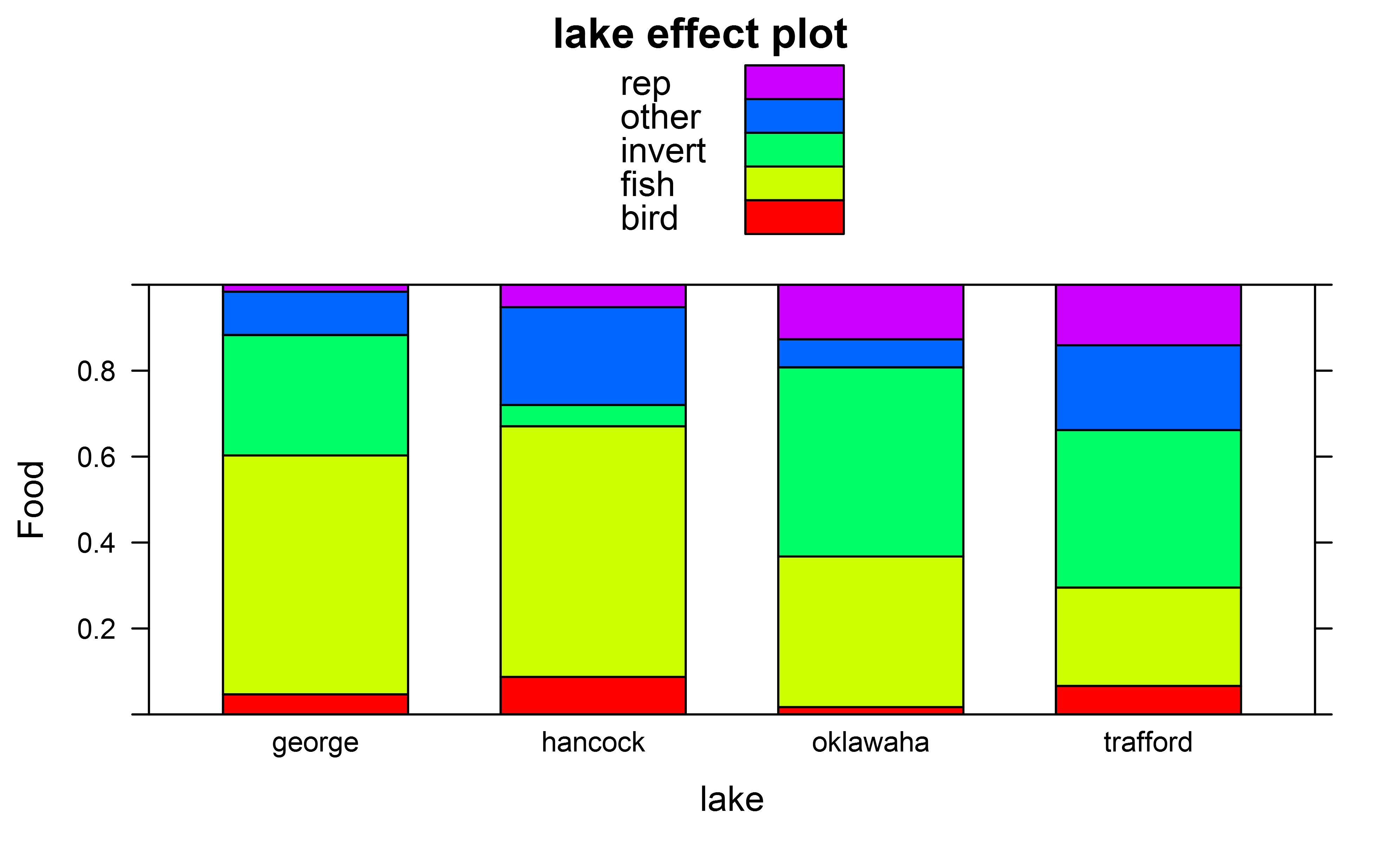
И графики эффекта, которые я получил, например, для фактора "озеро", используя
library(effects)
plot(effect(fit,term="lake"),ylab="Food",type="probability",style="stacked",colors=rainbow(5))
В дополнение к общим тестам Anova я также хотел бы также провести попарные постхок-тесты Тьюки, хотя для проверки общих различий в полиномиальном распределении продуктов, которые едят, например, по разным парам озер.
Я впервые подумал об использовании функции glht в упаковке multcomp но это не похоже на работу, например, для фактора lake:
library(multcomp)
summary(glht(fit, mcp(lake = "Tukey")))
Error in summary(glht(fit, mcp(lake = "Tukey"))) :
error in evaluating the argument 'object' in selecting a method for function 'summary': Error in glht.matrix(model = list(n = c(6, 0, 5), nunits = 12L, nconn = c(0, :
‘ncol(linfct)’ is not equal to ‘length(coef(model))’
Альтернативой было использовать пакет lsmeans за то, за что я пытался
lsmeans(fit, pairwise ~ lake | food, adjust="tukey", mode = "prob")
$contrasts
food = bird:
contrast estimate SE df t.ratio p.value
george - hancock -0.04397388 0.05451515 24 -0.807 0.8507
george - oklawaha 0.03680712 0.03849268 24 0.956 0.7751
george - trafford -0.02123255 0.05159049 24 -0.412 0.9760
hancock - oklawaha 0.08078100 0.04983303 24 1.621 0.3863
hancock - trafford 0.02274133 0.06242724 24 0.364 0.9831
oklawaha - trafford -0.05803967 0.04503128 24 -1.289 0.5786
food = fish:
contrast estimate SE df t.ratio p.value
george - hancock -0.02311955 0.09310322 24 -0.248 0.9945
george - oklawaha 0.19874095 0.09273047 24 2.143 0.1683
george - trafford 0.32066789 0.08342262 24 3.844 0.0041
hancock - oklawaha 0.22186050 0.09879102 24 2.246 0.1396
hancock - trafford 0.34378744 0.09088119 24 3.783 0.0047
oklawaha - trafford 0.12192695 0.08577365 24 1.421 0.4987
food = invert:
contrast estimate SE df t.ratio p.value
george - hancock 0.23202865 0.06111726 24 3.796 0.0046
george - oklawaha -0.13967425 0.08808698 24 -1.586 0.4053
george - trafford -0.07193252 0.08346283 24 -0.862 0.8242
hancock - oklawaha -0.37170290 0.07492749 24 -4.961 0.0003
hancock - trafford -0.30396117 0.07129577 24 -4.263 0.0014
oklawaha - trafford 0.06774173 0.09384594 24 0.722 0.8874
food = other:
contrast estimate SE df t.ratio p.value
george - hancock -0.12522495 0.06811177 24 -1.839 0.2806
george - oklawaha 0.03499241 0.05141930 24 0.681 0.9035
george - trafford -0.08643898 0.06612383 24 -1.307 0.5674
hancock - oklawaha 0.16021736 0.06759887 24 2.370 0.1103
hancock - trafford 0.03878598 0.08135810 24 0.477 0.9635
oklawaha - trafford -0.12143138 0.06402725 24 -1.897 0.2560
food = rep:
contrast estimate SE df t.ratio p.value
george - hancock -0.03971026 0.03810819 24 -1.042 0.7269
george - oklawaha -0.13086622 0.05735022 24 -2.282 0.1305
george - trafford -0.14106384 0.06037257 24 -2.337 0.1177
hancock - oklawaha -0.09115595 0.06462624 24 -1.411 0.5052
hancock - trafford -0.10135358 0.06752424 24 -1.501 0.4525
oklawaha - trafford -0.01019762 0.07161794 24 -0.142 0.9989
Results are averaged over the levels of: sex, size
P value adjustment: tukey method for comparing a family of 4 estimates
Это выполняет тесты на различия в пропорции каждого определенного типа продукта, хотя.
Мне было интересно, можно ли было бы так или иначе получить тесты Tukey posthoc, в которых общие полиномиальные распределения сравниваются по разным озерам, т. Е. Где проверяются различия в пропорции любого из съеденных продуктов добычи? Я пробовал с
lsmeans(fit, pairwise ~ lake, adjust="tukey", mode = "prob")
но это не похоже на работу:
$contrasts
contrast estimate SE df t.ratio p.value
george - hancock 3.252607e-19 1.879395e-10 24 0 1.0000
george - oklawaha -8.131516e-19 1.861245e-10 24 0 1.0000
george - trafford -1.843144e-18 2.504062e-10 24 0 1.0000
hancock - oklawaha -1.138412e-18 NaN 24 NaN NaN
hancock - trafford -2.168404e-18 NaN 24 NaN NaN
oklawaha - trafford -1.029992e-18 NaN 24 NaN NaN
Какие-нибудь мысли?
Или кто-нибудь знает, как glht можно заставить работать на multinom модели?
1 ответ
Только что получил доброе послание Расса Лента, и он подумал, что синтаксис для этих попарных сравнений между озерами позволяет проверить различия в многочленовом распределении продуктов, которые едят аллигаторы.
lsm = lsmeans(fit, ~ lake|food, mode = "latent")
cmp = contrast(lsm, method="pairwise", ref=1)
test = test(cmp, joint=TRUE, by="contrast")
There are linearly dependent rows - df are reduced accordingly
test
contrast df1 df2 F p.value
george - hancock 4 24 3.430 0.0236
george - oklawaha 4 24 2.128 0.1084
george - trafford 4 24 3.319 0.0268
hancock - oklawaha 4 24 5.820 0.0020
hancock - trafford 4 24 5.084 0.0041
oklawaha - trafford 4 24 1.484 0.2383
Спасибо, Русс!