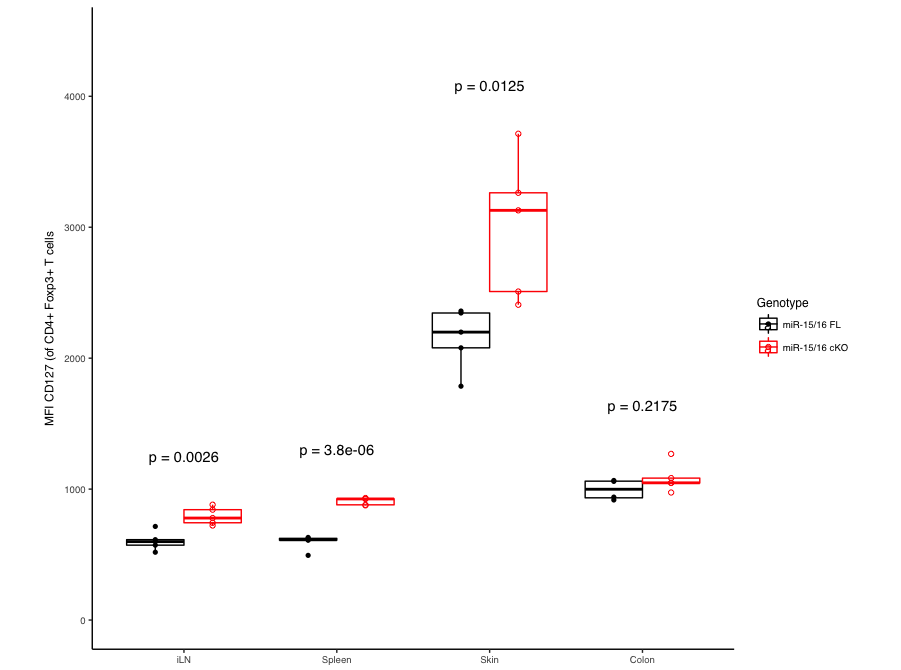
Как добавить бары сравнения на график, чтобы обозначить, какое значение a p сравнения соответствует
Я использую следующий фрейм данных:
df1 <- structure(list(Genotype = structure(c(1L, 1L, 1L, 1L, 1L,
2L,2L,2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L, 1L, 1L, 1L,
1L,1L, 2L, 2L, 2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L),
.Label= c("miR-15/16 FL", "miR-15/16 cKO"), class = "factor"),
Tissue = structure(c(1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L,
1L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L, 3L,
3L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L,
4L), .Label = c("iLN", "Spleen", "Skin", "Colon"), class = "factor"),
`Cells/SC/Live/CD8—,, CD4+/Foxp3+,Median,<BV421-A>,CD127` = c(518L,
715L, 572L, 599L, 614L, 881L, 743L, 722L, 779L, 843L, 494L,
610L, 613L, 624L, 631L, 925L, 880L, 932L, 876L, 926L, 1786L,
2079L, 2199L, 2345L, 2360L, 2408L, 2509L, 3129L, 3263L, 3714L,
917L, NA, 1066L, 1059L, 939L, 1269L, 1047L, 974L, 1048L,
1084L)),
.Names = c("Genotype", "Tissue", "Cells/SC/Live/CD8—,,CD4+/Foxp3+,Median,<BV421-A>,CD127"),
row.names = c(NA, -40L), class = c("tbl_df", "tbl", "data.frame"))
и пытаясь построить график, используя ggplot2, где отображаются поля и точки, сгруппированные по "Tissue" и чередующиеся по "Genotype". Значения значимости отображаются правильно, но я хотел бы добавить строки для обозначения проводимых сравнений, чтобы они начинались в центре каждого квадрата "miR-15/16 FL" и заканчивались в центре каждого "miR-15 /". 16 cKO"окно графика и сидеть прямо под значениями значимости. Ниже приведен код, который я использую для создания графика:
library(ggplot2)
library(ggpubr)
color.groups <- c("black","red")
names(color.groups) <- unique(df1$Genotype)
shape.groups <- c(16, 1)
names(shape.groups) <- unique(df1$Genotype)
ggplot(df1, aes(x = Tissue, y = df1[3], color = Genotype, shape = Genotype)) +
geom_boxplot(position = position_dodge(), outlier.shape = NA) +
geom_point(position=position_dodge(width=0.75)) +
ylim(0,1.2*max(df1[3], na.rm = TRUE)) +
ylab('MFI CD127 (of CD4+ Foxp3+ T cells') +
scale_color_manual(values=color.groups) +
scale_shape_manual(values=shape.groups) +
theme_bw() + theme(panel.border = element_blank(), panel.grid.major = element_blank(),
panel.grid.minor = element_blank(), axis.line = element_line(colour = "black"),
axis.title.x=element_blank(), aspect.ratio = 1,
text = element_text(size = 9)) +
stat_compare_means(show.legend = FALSE, label = 'p.format', method = 't.test',
label.y = c(0.1*max(df1[3], na.rm = TRUE) + max(df1[3][c(1:10),], na.rm = TRUE),
0.1*max(df1[3], na.rm = TRUE) + max(df1[3][c(11:20),], na.rm = TRUE),
0.1*max(df1[3], na.rm = TRUE) + max(df1[3][c(21:30),], na.rm = TRUE),
0.1*max(df1[3], na.rm = TRUE) + max(df1[3][c(31:40),], na.rm = TRUE)))
Спасибо за любую помощь!
1 ответ
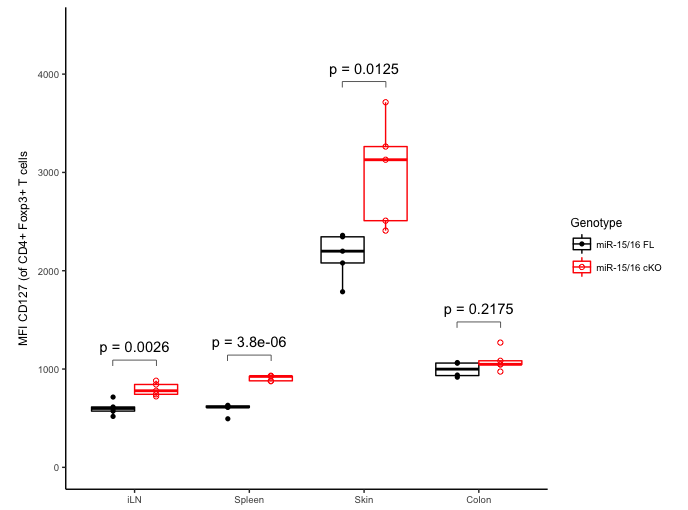
Я создал скобки с тремя вызовами geom_segment, Эти звонки используют новый dmax фрейм данных, созданный для предоставления опорных значений y для размещения скобок и меток p-значения. Ценности e а также r для настройки этих позиций.
Я сделал несколько других изменений в вашем коде.
Измените имя третьего столбца на
tempи использовать это имяy=tempв вызове ggplot. Ваш оригинальный код используетy=df1[3]который, по существу, выходит за пределы сюжетаdf1объект в родительской среде, что может вызвать проблемы. Кроме того, короткое имя, на которое можно сослаться, облегчает созданиеdmaxфрейм данных и ссылки на его столбцы.Использовать
dmaxфрейм данных дляlabel.yпозиции вstat_compare_means, что уменьшает количество кода, необходимого. (Incidently,stat_compare_meansкажется, требует жесткоlabel.yпозиции, а не получать их отaesотображение данных.)Расположите метки p-value как абсолютное расстояние над каждой парой прямоугольников (используя значение
e), а не мультипликативное расстояние. Это облегчает поддержание согласованности между метками p-значения, скобками и графиками.
# Use a short column name for the third column
names(df1)[3] = "temp"
# Generate data frame of reference y-values for p-value labels and bracket positions
dmax = df1 %>% group_by(Tissue) %>%
summarise(temp=max(temp, na.rm=TRUE),
Genotype=NA)
# For tweaking position of brackets
e = 350
r = 0.6
w = 0.19
bcol = "grey30"
ggplot(df1, aes(x = Tissue, y = temp, color = Genotype, shape = Genotype)) +
geom_boxplot(position = position_dodge(), outlier.shape = NA) +
geom_point(position=position_dodge(width=0.75)) +
ylim(0,1.2*max(df1[3], na.rm = TRUE)) +
ylab('MFI CD127 (of CD4+ Foxp3+ T cells') +
scale_color_manual(values=color.groups) +
scale_shape_manual(values=shape.groups) +
theme_bw() + theme(panel.border = element_blank(), panel.grid.major = element_blank(),
panel.grid.minor = element_blank(), axis.line = element_line(colour = "black"),
axis.title.x=element_blank(), aspect.ratio = 1,
text = element_text(size = 9)) +
stat_compare_means(show.legend = FALSE, label = 'p.format', method = 't.test',
label.y = e + dmax$temp) +
geom_segment(data=dmax,
aes(x=as.numeric(Tissue)-w, xend=as.numeric(Tissue)+w,
y=temp + r*e, yend=temp + r*e), size=0.3, color=bcol, inherit.aes=FALSE) +
geom_segment(data=dmax,
aes(x=as.numeric(Tissue) + w, xend=as.numeric(Tissue) + w,
y=temp + r*e, yend=temp + r*e - 60), size=0.3, color=bcol, inherit.aes=FALSE) +
geom_segment(data=dmax,
aes(x=as.numeric(Tissue) - w, xend=as.numeric(Tissue) - w,
y=temp + r*e, yend=temp + r*e - 60), size=0.3, color=bcol, inherit.aes=FALSE)
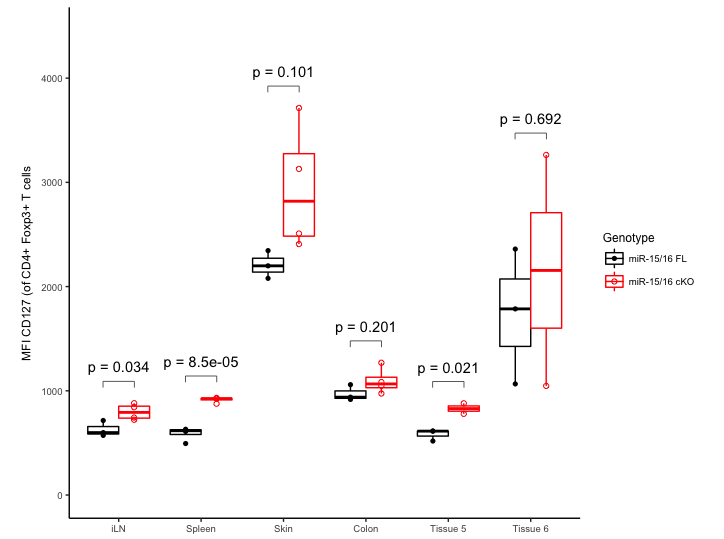
Чтобы ответить на ваш комментарий, вот пример, показывающий, что вышеописанный метод настраивается на любое количество x-категорий.
Давайте начнем с добавления двух новых категорий тканей:
library(forcats)
df1$Tissue = fct_expand(df1$Tissue, "Tissue 5", "Tissue 6")
df1$Tissue[seq(1,20,4)] = "Tissue 5"
df1$Tissue[seq(21,40,4)] = "Tissue 6"
dmax = df1 %>% group_by(Tissue) %>%
summarise(temp=max(temp, na.rm=TRUE),
Genotype=NA)
Теперь запустите точно такой же код, указанный выше, чтобы получить следующий график: