Используя Tidygraph, чтобы объединить два ребра из тех же двух узлов в один
Я изо всех сил пытаюсь выяснить, как сложить 2 ребра между теми же 2 узлами в 1, а затем вычислить сумму этих ребер.
Я считаю, что есть способ сделать это в igraph:
simplify(gcon, edge.attr.comb = list(weight = "sum", function(x)length(x)))
но я бы хотел сделать это с tidygraph если возможно, так как я имел успех в реализации до этого момента с tidygraph и я гораздо лучше знаком с tidyverse способ работы.
Мои данные выглядят так:
from to Strength Dataframe Question Topic
1 0 32 4 weekly 1 Connection Frequency
2 0 19 5 weekly 1 Connection Frequency
3 0 8 3 weekly 1 Connection Frequency
4 0 6 5 weekly 1 Connection Frequency
5 0 2 4 weekly 1 Connection Frequency
6 0 14 5 weekly 1 Connection Frequency
И то, и другое "от" и "до" содержит одинаковые идентификаторы (например, от-до; 0-1 и 1-0). Я хотел бы сжать так, чтобы существовала только одна итерация отношения 0-1 с суммированием Strength рассчитывается.
Вот мой код до сих пор:
graph <- data %>%
filter(Dataframe == "weekly" & Question == 1) %>%
as_tbl_graph(directed = FALSE) %>%
activate(edges) %>% # first manipulate edges
filter(!edge_is_loop()) %>% # remove any loops
activate(nodes) %>% # now manipulate nodes
left_join(node.group, by = "name") %>%
mutate(
Popularity = centrality_degree(mode = 'in'),
Centre = node_is_center(),
Keyplayer = node_is_keyplayer(k = 5))
Можно ли объединить два соответствующих ребра в одно ребро? Я искал форум, но наткнулся на ссылки, где одни и те же узлы повторяются в одних и тех же столбцах (то есть 0-1 в нескольких строках).
2 ответа
library(tidygraph) # v1.2.0
library(dplyr) # v0.8.5
library(purrr) # v0.3.4
dat <- data.frame(
from = c("a", "a", "b", "c"),
to = c("b", "b", "a", "b"),
n = 1:4
)
Вызов to_simple() в convert()свернуть параллельные края. Соответствующие ребра и веса хранятся в.orig_dataкак список блюд. Затем извлеките сумму веса сжатых ребер из.orig_data.
dat %>%
as_tbl_graph() %>%
convert(to_simple) %>%
activate(edges) %>%
mutate(n_sum = map_dbl(.orig_data, ~ sum(.x$n)))
# A tbl_graph: 3 nodes and 3 edges
#
# A directed simple graph with 1 component
#
# Edge Data: 3 x 5 (active)
from to .tidygraph_edge_index .orig_data n_sum
<int> <int> <list> <list> <dbl>
1 1 2 <int [2]> <tibble [2 x 3]> 3
2 2 1 <int [1]> <tibble [1 x 3]> 3
3 3 2 <int [1]> <tibble [1 x 3]> 4
#
# Node Data: 3 x 2
name .tidygraph_node_index
<chr> <int>
1 a 1
2 b 2
3 c 3
Я тоже борюсь с этой проблемой. Мое решение до сих пор состоит в том, чтобы сложить пары каждого узла, а затем суммировать веса. Что-то вроде этого:
require(dplyr)
require(tidyr)
pasteCols = function(x, y, sep = ":"){
stopifnot(length(x) == length(y))
return(lapply(1:length(x), function(i){paste0(sort(c(x[i], y[i])), collapse = ":")}) %>% unlist())
}
data = data %>%
mutate(col_pairs = pasteCols(from, to, sep = ":")) %>%
group_by(col_pairs) %>% summarise(sum_weight = sum(weight)) %>%
tidyr::separate(col = col_pairs, c("from", "to"), sep = ":")
Вы можете свернуть несколько ребер в графе g, перейдя к взвешенной метрике смежности и обратно в график igraph следующим образом:
gg <- graph.adjacency(get.adjacency(g), mode="undirected", weighted=TRUE)
Сейчас gg будет содержать атрибут edge $weight соответствует числу ребер, которые произошли между каждой парой вершин в g,
Я не очень знаком с тидиграфом, но я создал этот педагогический код, чтобы облегчить твой путь.
# A graph from sample data
sample_el <- cbind(c(1,1,1,2,2,2,3,3,3,4,4,5,5,6,6,6,7,7,7,7,8,8),
c(2,2,3,6,6,4,4,6,8,5,5,6,8,7,7,2,6,8,3,6,4,4))
g <- graph_from_edgelist(sample_el, directed=F)
# Always plot graphs with this same layout
mylaoyt <- layout_(g, as_star())
plot(g, layout = mylaoyt)
# Merge all duplicate edges using a weighted adjacency matric that
# is converted back to a graph
gg <- graph.adjacency(get.adjacency(g), mode="undirected", weighted=TRUE)
# function to return a weighted edgelist from a graph
get.weighted.edgelist <- function(graph){cbind(get.edgelist(gg), E(gg)$weight)}
# compare your two edge-lists. el has duplicates, wel is weighted
el <- get.edgelist(g)
wel<- get.weighted.edgelist(gg)
el
wel
# Plot the two graphs to see what el and wel would look like:
par(mfrow=c(1,2))
plot(g, layout=mylaoyt, vertex.label=NA, vertex.size=10)
plot(gg, layout=mylaoyt, vertex.label=NA, vertex.size=10, edge.width=E(gg)$weight * 3)
Выход в el а также wel это визуализировать так:
Надеюсь, что вы можете вырезать то, что вам нужно.
tidygraph может упростить графики, когда в morphсостояние Эд с simplify_to вызов, но он возвращается к исходному, когда unmorphing.
Это простой способ:
data <- read.table(header=TRUE, text="
from to weight
0 14 5
0 1 1
1 0 1
")
original <- as_tbl_graph(data)
Вход:
> original
# A tbl_graph: 3 nodes and 3 edges
#
# A directed simple graph with 1 component
#
# Node Data: 3 x 1 (active)
name
<chr>
1 0
2 1
3 14
#
# Edge Data: 3 x 3
from to weight
<int> <int> <int>
1 1 3 5
2 1 2 1
3 2 1 1
Решение:
modified <- original %>% activate(edges) %>%
# create a temporary grouping & filtering variable by sorting from/to IDs
mutate(temp = ifelse(from > to, paste0(to, from), paste0(from, to))) %>%
group_by(temp) %>%
mutate(weight = sum(weight)) %>%
ungroup() %>%
dplyr::distinct(temp, .keep_all = TRUE) %>%
select(-temp)
Выход:
> modified
# A tbl_graph: 3 nodes and 2 edges
#
# A rooted tree
#
# Edge Data: 2 x 3 (active)
from to weight
<int> <int> <int>
1 1 3 5
2 1 2 2
#
# Node Data: 3 x 1
name
<chr>
1 0
2 1
3 14
Вот один подход. Оно используетtidygraph, который использует igraph под капотом.
library(tidygraph)
#>
#> Attaching package: 'tidygraph'
#> The following object is masked from 'package:stats':
#>
#> filter
library(igraph)
#>
#> Attaching package: 'igraph'
#> The following object is masked from 'package:tidygraph':
#>
#> groups
#> The following objects are masked from 'package:stats':
#>
#> decompose, spectrum
#> The following object is masked from 'package:base':
#>
#> union
library(ggraph)
#> Loading required package: ggplot2
library(tidyverse)
g <- tibble(from = sample(letters[1:5], 25, T),
to = sample(letters[1:5], 25, T)) %>%
as_tbl_graph()
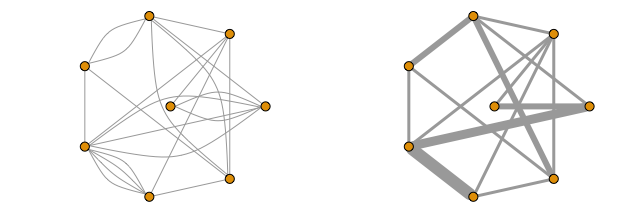
ggraph(g)+
geom_edge_parallel(arrow = arrow(type = 'closed'),
start_cap = circle(7.5, 'mm'),
end_cap = circle(7.5, 'mm'))+
geom_node_label(aes(label = name))+
labs(title = 'Multiple edges shown between node pairs')
#> Using `stress` as default layout

# Add the weigths as counts in the original dataframe
g_weights <- g %>%
activate(edges) %>%
as_tibble() %>%
mutate(link = glue::glue('{from}_{to}')) %>%
add_count(link) %>%
distinct(link, n, .keep_all = T) %>%
select(from, to, n) %>%
as_tbl_graph()
ggraph(g_weights)+
geom_edge_parallel(arrow = arrow(type = 'closed'),
start_cap = circle(7.5, 'mm'),
end_cap = circle(7.5, 'mm'),
aes(width = n))+
geom_node_label(aes(label = name))+
labs(title = 'Single edges shown between node pairs',
subtitle = 'Weights used as edge width')+
scale_edge_width(range = c(.5, 2), name = 'Weight')
#> Using `stress` as default layout

Создано 03.09.2019 пакетом REPEX (v0.3.0)