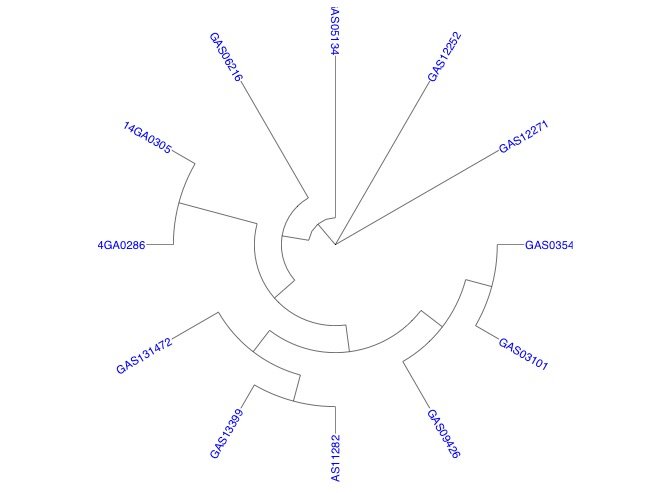
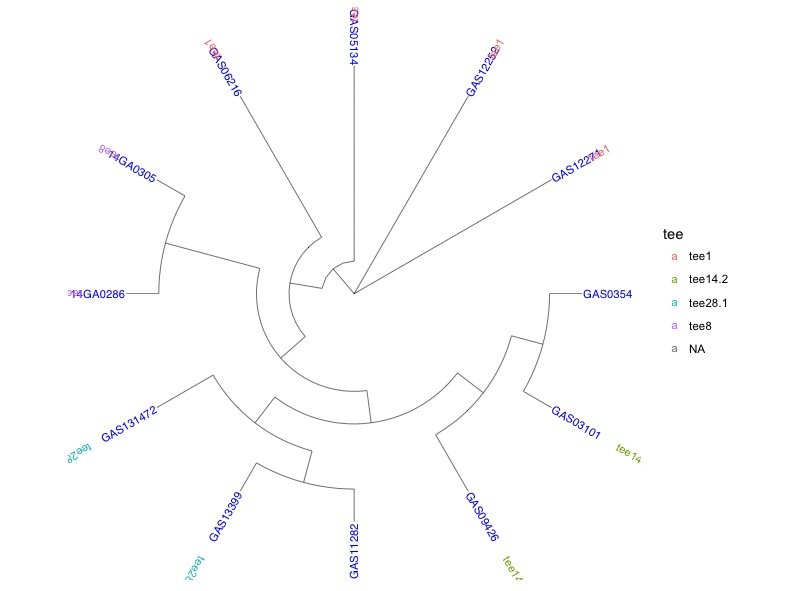
Можно ли кодировать geom_text в gg_tree, чтобы не нарушать расстояние от узлов?
Я выровнял некоторые аминокислотные последовательности в R и импортировал матрицу расстояний (dist_mat) для использования в ggtree, используя tree <- ape::nj(dist_mat), Это выглядит примерно так:
node parent branch.length x y label isTip branch angle
1 1 14 0.000000000 0.00000000 3.000000 GAS05134 TRUE 0.000000000 90.0000
2 2 13 0.000000000 0.00000000 2.000000 GAS12252 TRUE 0.000000000 60.0000
3 3 13 0.000000000 0.00000000 1.000000 GAS12271 TRUE 0.000000000 30.0000
4 4 15 0.004565217 0.02000000 4.000000 GAS06216 TRUE 0.017717391 120.0000
5 5 18 0.060110914 0.85012362 7.000000 GAS131472 TRUE 0.820068164 210.0000
6 6 19 0.000000000 0.84990179 8.000000 GAS13399 TRUE 0.849901793 240.0000
7 7 19 0.000000000 0.84990179 9.000000 GAS11282 TRUE 0.849901793 270.0000
8 8 21 0.000000000 0.92485325 11.000000 GAS03101 TRUE 0.924853253 330.0000
9 9 21 0.000000000 0.92485325 12.000000 GAS0354 TRUE 0.924853253 360.0000
10 10 20 0.000000000 0.92485325 10.000000 GAS09426 TRUE 0.924853253 300.0000
11 11 22 0.000000000 0.91032609 5.000000 14GA0305 TRUE 0.910326087 150.0000
12 12 22 0.000000000 0.91032609 6.000000 14GA0286 TRUE 0.910326087 180.0000
13 13 13 0.000000000 0.00000000 2.447917 <NA> FALSE 0.000000000 73.4375
14 14 13 0.000000000 0.00000000 4.343750 <NA> FALSE 0.000000000 130.3125
15 15 14 0.015434783 0.01543478 5.687500 <NA> FALSE 0.007717391 170.6250
16 16 15 0.454136361 0.46957114 7.375000 <NA> FALSE 0.242502963 221.2500
17 17 16 0.031992271 0.50156341 9.250000 <NA> FALSE 0.485567279 277.5000
18 18 17 0.288449292 0.79001271 7.750000 <NA> FALSE 0.645788061 232.5000
19 19 18 0.059889086 0.84990179 8.500000 <NA> FALSE 0.819957250 255.0000
20 20 17 0.423289838 0.92485325 10.750000 <NA> FALSE 0.713208334 322.5000
21 21 20 0.000000000 0.92485325 11.500000 <NA> FALSE 0.924853253 345.0000
22 22 16 0.440754944 0.91032609 5.500000 <NA> FALSE 0.689948615 165.0000
Базовое представление в gg_tree выглядит так:
> gg_tree <- ggtree(size=0.2,tree, layout = "circular", branch.length = "none") + geom_tiplab2(color='blue', size=3)
Затем я добавляю некоторые данные, чтобы добавить эстетику из исходного фрейма данных:
> gg_tree <- gg_tree %<+% DF
> head(DF, 12)
# A tibble: 12 x 4
id emm tee `50aa_HVR_peptide`
<chr> <chr> <chr> <chr>
1 GAS05134 emm1.0 tee1 NGDGNPREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA
2 GAS12252 emm1.0 tee1 NGDGNPREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA
3 GAS12271 emm1.0 tee1 NGDGNPREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA
4 GAS06216 emm1.19 tee1 NGDGNLREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA
5 GAS131472 emm100.0 tee28.1 RVTTRSQAQDAAGLKEKADKYEVRNHELEHNNEKLKTENSDLKTENSKLT
6 GAS13399 emm100.5 tee28.1 RVTTRSQAQDAAGLKEKADKYEVRNHELEHNNEKLKTENSKLTSEKEELT
7 GAS11282 emm100.5 tee28.1 RVTTRSQAQDAAGLKEKADKYEVRNHELEHNNEKLKTENSKLTSEKEELT
8 GAS03101 emm101.0 tee14.2 ADHPSYTAAKDEVLSKFSVPGHVWAHEREKNDKLSSENEGLKAGLQEKEQ
9 GAS0354 emm101.0 tee14.2 ADHPSYTAAKDEVLSKFSVPGHVWAHEREKNDKLSSENEGLKAGLQEKEQ
10 GAS09426 emm101.0 tee14.2 ADHPSYTAAKDEVLSKFSVPGHVWAHEREKNDKLSSENEGLKAGLQEKEQ
11 14GA0305 emm103.0 tee8 DSPRDVTSDLTTSMWKKKAEEAEAKASKFEKQLEDYKKAQKDYYEIEEKL
12 14GA0286 emm103.0 tee8 DSPRDVTSDLTTSMWKKKAEEAEAKASKFEKQLEDYKKAQKDYYEIEEKL
Затем я хотел бы добавить информацию в столбце "тройник" в виде цветного текста на внешней стороне дерева, моя попытка заключается в следующем:
> gg_tree + geom_text(size = 3,aes(angle=angle, color=tee, label=tee), hjust=-2)+
theme(legend.position="right")
Как вы можете видеть, я пытался использовать "hjust", чтобы слой "geom_text" не перекрывался со слоем "geom_tiplab2", но расстояние для каждого текста "tee" до метки наконечника, кажется, существенно отличается.
Кто-нибудь может подсказать, как сделать так, чтобы текст "тройника" плавно проходил по внешней стороне дерева, следуя формам подсказок? Примечание: это происходит и с прямоугольными деревьями, а не только с круглыми.
> sessionInfo()
R version 3.4.3 (2017-11-30)
Platform: x86_64-apple-darwin15.6.0 (64-bit)
Running under: macOS High Sierra 10.13.2
Matrix products: default
BLAS: /System/Library/Frameworks/Accelerate.framework/Versions/A/Frameworks/vecLib.framework/Versions/A/libBLAS.dylib
LAPACK: /Library/Frameworks/R.framework/Versions/3.4/Resources/lib/libRlapack.dylib
locale:
[1] en_GB.UTF-8/en_GB.UTF-8/en_GB.UTF-8/C/en_GB.UTF-8/en_GB.UTF-8
attached base packages:
[1] stats4 parallel stats graphics grDevices utils datasets methods base
other attached packages:
[1] ggtree_1.10.2 treeio_1.2.1 ggplot2_2.2.1 readxl_1.0.0
[5] readr_1.1.1 DECIPHER_2.6.0 RSQLite_2.0 Biostrings_2.46.0
[9] XVector_0.18.0 IRanges_2.12.0 S4Vectors_0.16.0 BiocGenerics_0.24.0
loaded via a namespace (and not attached):
[1] Rcpp_0.12.14 pillar_1.0.1 compiler_3.4.3 cellranger_1.1.0 plyr_1.8.4
[6] tools_3.4.3 zlibbioc_1.24.0 digest_0.6.13 bit_1.1-12 jsonlite_1.5
[11] memoise_1.1.0 tibble_1.4.1 gtable_0.2.0 nlme_3.1-131 lattice_0.20-35
[16] pkgconfig_2.0.1 rlang_0.1.6 cli_1.0.0 rstudioapi_0.7 DBI_0.7
[21] rvcheck_0.0.9 hms_0.4.0 bit64_0.9-7 grid_3.4.3 glue_1.2.0
[26] R6_2.2.2 purrr_0.2.4 tidyr_0.7.2 blob_1.1.0 magrittr_1.5
[31] scales_0.5.0 assertthat_0.2.0 colorspace_1.3-2 ape_5.0 labeling_0.3
[36] utf8_1.1.3 lazyeval_0.2.1 munsell_0.4.3 crayon_1.3.4
1 ответ
hjust а также vjust не так хорошо с coord_polar один трюк, чтобы переместить geom_text Метки от центра слишком добавить значение к x координата:
library(ggtree)
gg_tree + geom_text(size = 3, aes(angle = angle,
color = tee,
label = tee,
x = x + 0.4), hjust = 0)+
theme(legend.position = "right")
установить gg_tree:
source("https://bioconductor.org/biocLite.R")
biocLite("ggtree")
используемые данные:
> dput(DF)
structure(list(id = structure(c(5L, 9L, 10L, 6L, 11L, 12L, 8L,
3L, 4L, 7L, 2L, 1L), .Label = c("14GA0286", "14GA0305", "GAS03101",
"GAS0354", "GAS05134", "GAS06216", "GAS09426", "GAS11282", "GAS12252",
"GAS12271", "GAS131472", "GAS13399"), class = "factor"), emm = structure(c(1L,
1L, 1L, 2L, 3L, 4L, 4L, 5L, 5L, 5L, 6L, 6L), .Label = c("emm1.0",
"emm1.19", "emm100.0", "emm100.5", "emm101.0", "emm103.0"), class = "factor"),
tee = structure(c(1L, 1L, 1L, 1L, 3L, 3L, 3L, 2L, 2L, 2L,
4L, 4L), .Label = c("tee1", "tee14.2", "tee28.1", "tee8"), class = "factor"),
X.50aa_HVR_peptide. = structure(c(4L, 4L, 4L, 3L, 5L, 6L,
6L, 1L, 1L, 1L, 2L, 2L), .Label = c("ADHPSYTAAKDEVLSKFSVPGHVWAHEREKNDKLSSENEGLKAGLQEKEQ",
"DSPRDVTSDLTTSMWKKKAEEAEAKASKFEKQLEDYKKAQKDYYEIEEKL", "NGDGNLREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA",
"NGDGNPREVIEDLAANNPAIQNIRLRHENKDLKARLENAMEVAGRDFKRA", "RVTTRSQAQDAAGLKEKADKYEVRNHELEHNNEKLKTENSDLKTENSKLT",
"RVTTRSQAQDAAGLKEKADKYEVRNHELEHNNEKLKTENSKLTSEKEELT"), class = "factor")), .Names = c("id",
"emm", "tee", "X.50aa_HVR_peptide."), class = "data.frame", row.names = c("1",
"2", "3", "4", "5", "6", "7", "8", "9", "10", "11", "12"))
> dput(tree)
structure(list(node = 1:22, parent = c(14L, 13L, 13L, 15L, 18L,
19L, 19L, 21L, 21L, 20L, 22L, 22L, 13L, 13L, 14L, 15L, 16L, 17L,
18L, 17L, 20L, 16L), branch.length = c(0, 0, 0, 0.004565217,
0.060110914, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0.015434783, 0.454136361,
0.031992271, 0.288449292, 0.059889086, 0.423289838, 0, 0.440754944
), x = c(0, 0, 0, 0.02, 0.85012362, 0.84990179, 0.84990179, 0.92485325,
0.92485325, 0.92485325, 0.91032609, 0.91032609, 0, 0, 0.01543478,
0.46957114, 0.50156341, 0.79001271, 0.84990179, 0.92485325, 0.92485325,
0.91032609), y = c(3, 2, 1, 4, 7, 8, 9, 11, 12, 10, 5, 6, 2.447917,
4.34375, 5.6875, 7.375, 9.25, 7.75, 8.5, 10.75, 11.5, 5.5), label = structure(c(6L,
10L, 11L, 7L, 12L, 13L, 9L, 4L, 5L, 8L, 3L, 2L, 1L, 1L, 1L, 1L,
1L, 1L, 1L, 1L, 1L, 1L), .Label = c("<NA>", "14GA0286", "14GA0305",
"GAS03101", "GAS0354", "GAS05134", "GAS06216", "GAS09426", "GAS11282",
"GAS12252", "GAS12271", "GAS131472", "GAS13399"), class = "factor"),
isTip = c(TRUE, TRUE, TRUE, TRUE, TRUE, TRUE, TRUE, TRUE,
TRUE, TRUE, TRUE, TRUE, FALSE, FALSE, FALSE, FALSE, FALSE,
FALSE, FALSE, FALSE, FALSE, FALSE), branch = c(0, 0, 0, 0.017717391,
0.820068164, 0.849901793, 0.849901793, 0.924853253, 0.924853253,
0.924853253, 0.910326087, 0.910326087, 0, 0, 0.007717391,
0.242502963, 0.485567279, 0.645788061, 0.81995725, 0.713208334,
0.924853253, 0.689948615), angle = c(90, 60, 30, 120, 210,
240, 270, 330, 360, 300, 150, 180, 73.4375, 130.3125, 170.625,
221.25, 277.5, 232.5, 255, 322.5, 345, 165)), .Names = c("node",
"parent", "branch.length", "x", "y", "label", "isTip", "branch",
"angle"), class = "data.frame", row.names = c("1", "2", "3",
"4", "5", "6", "7", "8", "9", "10", "11", "12", "13", "14", "15",
"16", "17", "18", "19", "20", "21", "22"))