R Как сгенерировать вектор вероятностей, который обычно распределяется для использования на chisq.test
У меня есть вектор из 30 выборок. Я хочу проверить гипотезу о том, что выборка из популяции, которая обычно распределяется.
> N.concentration
[1] 0.164 0.045 0.069 0.100 0.050 0.080 0.043 0.036 0.057 0.154 0.133 0.193
[13] 0.129 0.121 0.081 0.178 0.041 0.040 0.116 0.078 0.104 0.095 0.116 0.038
[25] 0.141 0.100 0.104 0.078 0.121 0.104
Я сделал частотный вектор, используя hist
> N.hist <- hist(N.concentration, breaks=10)
> N.freq <- N.hist$count
[1] 3 5 4 4 5 4 2 2 1
я использую chisq.test проверить пригодность N.freq однако для нормального распределения функция требует аргумента p = вектор вероятностей такой же длины x, как определено в документации chisq.test. Я пытаюсь создать вектор для него, но, честно говоря, я не знаю точно, что генерировать. я пытаюсь
> d <- length(N.freq$count)%/%2
> p <- dnorm(c(-d:d))
> p
[1] 0.0001338302 0.0044318484 0.0539909665 0.2419707245 0.3989422804
[6] 0.2419707245 0.0539909665 0.0044318484 0.0001338302
> chisq.test(N.freq, p = p)
Error in chisq.test(p1$count, p = p) :
probabilities must sum to 1.
Я думал об использовании rescale.p=TRUE но я не уверен, что это даст действительный тест.
РЕДАКТИРОВАТЬ: если я использую rescale.p, я получил предупреждение
> chisq.test(N.freq, p=p, rescale.p=TRUE)
Chi-squared test for given probabilities
data: N.freq
X-squared = 2697.7, df = 8, p-value < 2.2e-16
Warning message:
In chisq.test(N.freq, p = p, rescale.p = TRUE) :
Chi-squared approximation may be incorrect
2 ответа
Как я уже сказал, чтобы проверить нормальность, мы должны знать среднее значение и стандартную ошибку нормального распределения в нулевой гипотезе. Поскольку данных значений нет, мы должны оценить их по вашим 30 данным.
x <- c(0.164, 0.045, 0.069, 0.1, 0.05, 0.08, 0.043, 0.036, 0.057,
0.154, 0.133, 0.193, 0.129, 0.121, 0.081, 0.178, 0.041, 0.04,
0.116, 0.078, 0.104, 0.095, 0.116, 0.038, 0.141, 0.1, 0.104,
0.078, 0.121, 0.104)
mu <- mean(x)
sig <- sd(x)
Теперь, как и то, что вы сделали, нам нужно скопировать данные:
h <- hist(x, breaks = 10)
#List of 6
# $ breaks : num [1:10] 0.02 0.04 0.06 0.08 0.1 0.12 0.14 0.16 0.18 0.2
# $ counts : int [1:9] 3 5 4 4 5 4 2 2 1
# $ density : num [1:9] 5 8.33 6.67 6.67 8.33 ...
# $ mids : num [1:9] 0.03 0.05 0.07 0.09 0.11 0.13 0.15 0.17 0.19
# $ xname : chr "x"
# $ equidist: logi TRUE
# - attr(*, "class")= chr "histogram"
Чтобы получить истинную вероятность при нулевой гипотезе, нам нужна вероятность для каждой ячейки бина, то есть между перерывами.
p <- diff(pnorm(h$breaks, mu, sig))
#[1] 0.05675523 0.10254734 0.15053351 0.17953337 0.17396679 0.13696059 0.08760419
#[8] 0.04552387 0.01921839
Я склонен не доверять критерию хи-квадрат только с 30 данными. Но вот как мы можем использовать chisq.test:
chisq.test(h$counts, p = p, rescale.p = TRUE)
#
# Chi-squared test for given probabilities
#
#data: h$counts
#X-squared = 3.1476, df = 8, p-value = 0.9248
#
#Warning message:
#In chisq.test(h$counts, p, rescale.p = TRUE) :
# Chi-squared approximation may be incorrect
Часто вам не нужно беспокоиться о предупреждении. Если вы хотите избавиться от него, установите simulate.p.value = TRUE:
chisq.test(h$counts, p = p, rescale.p = TRUE, simulate.p.value = TRUE)
#
# Chi-squared test for given probabilities with simulated p-value (based
# on 2000 replicates)
#
#data: h$counts
#X-squared = 3.1476, df = NA, p-value = 0.942
Существует множество статистических тестов, разработанных для проверки отклонения от нормальности указанного набора данных (например, см. Пакет nortest). Однако вы должны знать, что многие статистики считают, что тестирование нормальности "по сути бесполезно": в частности (из ответа на связанный вопрос CrossValidated):
Ученые часто задают вопрос, на который ответит тест нормальности: достаточно ли отклоняются данные от гауссовского идеала, чтобы "запретить" использование теста, предполагающего гауссово распределение? Ученые часто хотят, чтобы тест на нормальность был тем судьей, который решает, когда следует отказаться от обычных (ANOVA и т. Д.) Тестов и вместо этого проанализировать преобразованные данные или использовать непараметрический тест на основе рангов или метод повторной выборки или начальной загрузки. Для этого тесты на нормальность не очень полезны.
Однако, продолжая и используя тест Шапиро-Уилка от базы R (согласно странице Википедии, Шапиро-Уилк обладает хорошей силой - но заметьте из обсуждения выше, что высокая мощность не обязательно является тем, чего мы действительно хотим в этом случае...)
d <- c(0.164,0.045,0.069,0.100,0.050,0.080,0.043,0.036,0.057,0.154,
0.133,0.193,0.129,0.121,0.081,0.178,0.041,0.040,0.116,0.078,
0.104,0.095,0.116,0.038,0.141,0.100,0.104,0.078,0.121,0.104)
shapiro.test(d)
## Shapiro-Wilk normality test
##
## data: d
## W = 0.9547, p-value = 0.2255
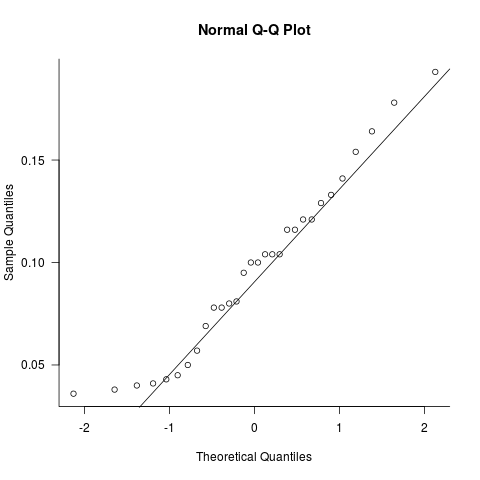
Графический подход:
par(las=1,bty="l")
qqnorm(d)
qqline(d)
Точки достаточно хорошо следуют за линией, и наибольшие отклонения (три наименьших точки в наборе данных) на самом деле больше, чем ожидалось, что означает, что набор данных немного тонкохвостый на нижнем конце, что означает, что тесты основаны на предположении нормальности, как правило, будет слегка консервативным.