Ограничения для линейной модели с использованием Quadprog для диапазонов / пределов коэффициентов
Я делаю некоторые эксперименты, и я, конечно, знаю, почему ограничение коэффициентов требуется редко, но здесь идет.
В следующих данных я использовал quadprog для решения линейной модели. Обратите внимание, что X1 просто перехват.
X1 <- 1
X2 <- c(4.374, 2.3708, -7.3033, 12.0803, -0.4098, 53.0631, 13.1304, 7.3617, 16.6252, 27.3394)
X3 <- c(2.6423, 2.6284, 36.9398, 15.9278, 18.3124, 54.5039, 3.764, 19.0552, 25.4906, 13.0112)
X4 <- c(4.381, 3.144, 9.506, 15.329, 21.008, 38.091, 22.399, 13.223, 17.419, 19.87)
X <- as.matrix(cbind(X1,X2,X3,X4))
Y <- as.matrix(c(37.7,27.48,24.08,25.97,16.65,73.77,45.10,53.35,61.95,71.15))
M1 <- solve.QP(t(X) %*% X, t(Y) %*% X, matrix(0, 4, 0), c())$solution
Задача состоит в том, чтобы ограничить определенные коэффициенты. Я знаю, что я должен изменить параметры Amet и bvac (согласно линейной регрессии с ограничениями на коэффициенты). Однако я не уверен, как его настроить, чтобы были соблюдены следующие ограничения.
Выходные данные читаются как [1] 37.3468790 1.2872473 -0.0177749 -0.5988443, где значения будут прогнозируемыми подобранными значениями X1, X2, X3 и X4.
Ограничения (при условии)…
X2 <= .899
0 <= X3 <= .500
0 <= X4 <= .334
1 ответ
Просто чтобы уточнить: вы даете ожидаемый результат "где значения будут предсказаны подогнанные значения X1, X2, X3 и X4". Я предполагаю, что вы имеете в виду приблизительные (не установленные) значения параметров.
Это довольно просто реализовать ограниченные параметры при моделировании данных в rstan, rstan интерфейс R для Stan, который, в свою очередь, является вероятностным языком программирования для статистического байесовского вывода.
Вот пример на основе предоставленных вами образцов данных.
Во-первых, давайте определимся с нашей моделью. Обратите внимание, что мы ограничиваем параметры
beta2,beta3а такжеbeta4согласно вашим требованиямmodel <- " data { int<lower=1> n; // number of observations int<lower=0> k; // number of parameters matrix[n, k] X; // data vector[n] y; // response } parameters { real beta1; // X1 unconstrained real<lower=negative_infinity(), upper=0.899> beta2; // X2 <= .899 real<lower=0, upper=0.5> beta3; // 0 <= X3 <= 0.5 real<lower=0, upper=0.334> beta4; // 0 <= X4 <= 0.334 real sigma; // residuals } model { // Likelihood y ~ normal(beta1 * X[, 1] + beta2 * X[, 2] + beta3 * X[, 3] + beta4 * X[, 4], sigma); }"Далее мы подгоняем модель к вашим образцам данных.
library(rstan) rstan_options(auto_write = TRUE) options(mc.cores = parallel::detectCores()) df <- cbind.data.frame(Y, X) fit <- stan( model_code = model, data = list( n = nrow(df), k = ncol(df[, -1]), X = df[, -1], y = df[, 1]))Наконец, давайте напечатаем оценки параметров аккуратно
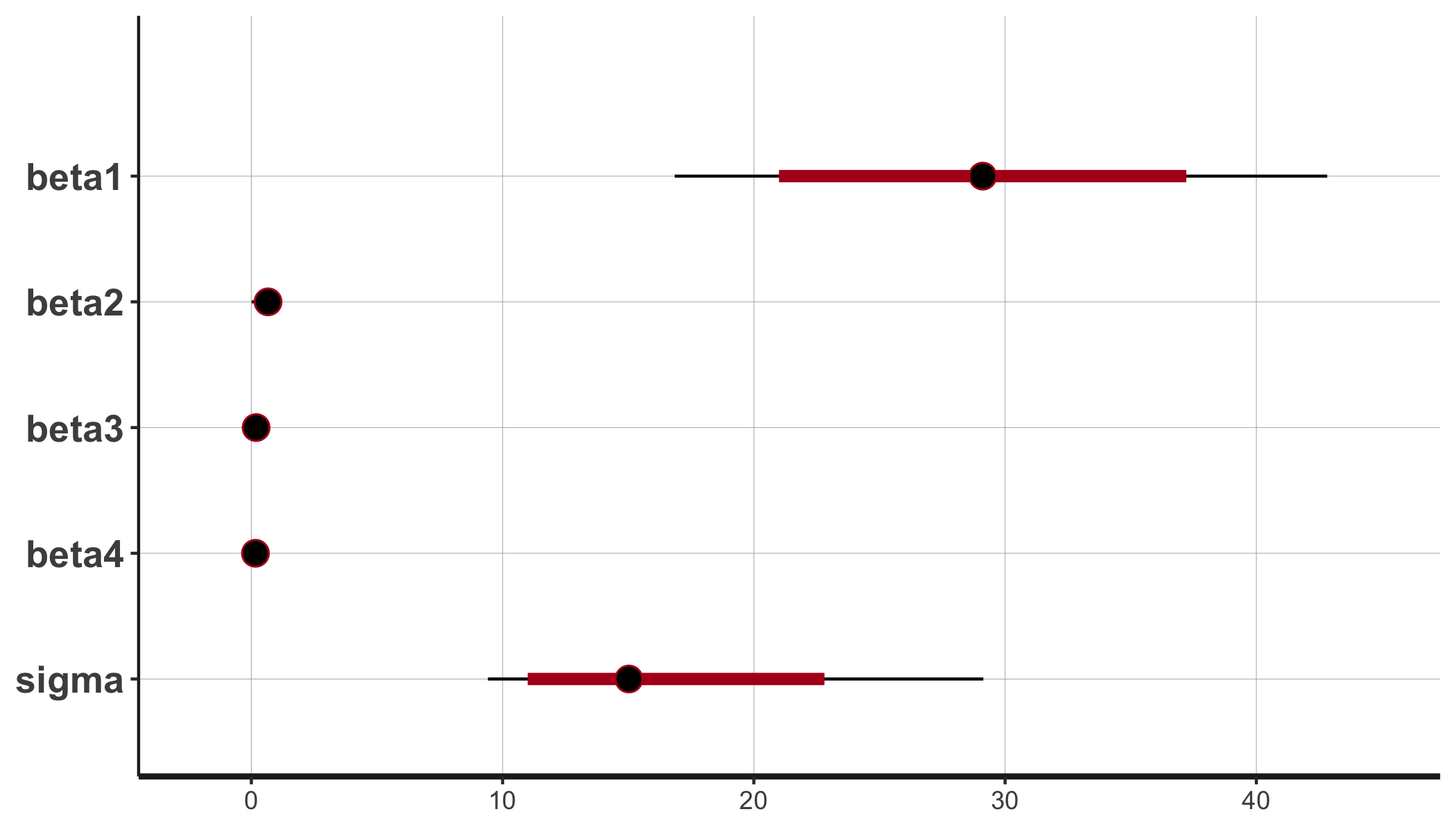
library(broom) tidy(fit, conf.int = TRUE) ## A tibble: 5 x 5 # term estimate std.error conf.low conf.high # <chr> <dbl> <dbl> <dbl> <dbl> #1 beta1 29.2 6.53 16.9 42.8 #2 beta2 0.609 0.234 0.0149 0.889 #3 beta3 0.207 0.138 0.00909 0.479 #4 beta4 0.164 0.0954 0.00780 0.326 #5 sigma 16.2 5.16 9.42 29.1Мы также можем построить оценки параметров, включая CI.
Обратите внимание, что оценки параметров согласуются с наложенными ограничениями.