Отредактировано: Как написать цикл для теста PostHoc Tukey и множественного сравнения
Это мой образец большой матрицы данных, и каждый столбец назван с несколькими данными и разделен подчеркиванием.
structure(list(Gene = c("AGI4120.1_UBQ", "AGI570.1_Acin"), WT_Tissue_0T_1 = c(0.886461437, 1.093164915), WT_Tissue_0T_2 = c(1.075140682, 1.229862834), WT_Tissue_0T_3 = c(0.632903012, 1.094003128), WT_Tissue_1T_1 = c(0.883151274, 1.26322126), WT_Tissue_1T_2 = c(1.005627276, 0.962729188), WT_Tissue_1T_3 = c(0.87123469, 0.968078993), WT_Tissue_3T_1 = c(0.723601456, 0.633890322), WT_Tissue_3T_2 = c(0.392585237, 0.534819363), WT_Tissue_3T_3 = c(0.640185369, 1.021934772), WT_Tissue_5T_1 = c(0.720291294, 0.589244505), WT_Tissue_5T_2 = c(0.362131744, 0.475251717), WT_Tissue_5T_3 = c(0.549486925, 0.618177919), mut1_Tissue_0T_1 = c(1.464415756, 1.130533457), mut1_Tissue_0T_2 = c(1.01489573, 1.114915728), mut1_Tissue_0T_3 = c(1.171797418, 1.399956009), mut1_Tissue_1T_1 = c(0.927507448, 1.231911575), mut1_Tissue_1T_2 = c(1.089705396, 1.256782289 ), mut1_Tissue_1T_3 = c(0.993048659, 0.999044465), mut1_Tissue_3T_1 = c(1.000993049, 1.103486794), mut1_Tissue_3T_2 = c(1.062562066, 0.883617224 ), mut1_Tissue_3T_3 = c(1.037404833, 0.851875438), mut1_Tissue_5T_1 = c(0.730883813, 0.437440083), mut1_Tissue_5T_2 = c(0.480635551, 0.298762126 ), mut1_Tissue_5T_3 = c(0.85468388, 0.614923997)), row.names = c(NA, -2L), class = c("tbl_df", "tbl", "data.frame"), spec = structure(list( cols = list(Gene = structure(list(), class = c("collector_character", "collector")), WT_Tissue_0T_1 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_0T_2 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_0T_3 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_1T_1 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_1T_2 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_1T_3 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_3T_1 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_3T_2 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_3T_3 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_5T_1 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_5T_2 = structure(list(), class = c("collector_double", "collector")), WT_Tissue_5T_3 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_0T_1 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_0T_2 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_0T_3 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_1T_1 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_1T_2 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_1T_3 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_3T_1 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_3T_2 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_3T_3 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_5T_1 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_5T_2 = structure(list(), class = c("collector_double", "collector")), mut1_Tissue_5T_3 = structure(list(), class = c("collector_double", "collector"))), default = structure(list(), class = c("collector_guess", "collector"))), class = "col_spec"))
Я хочу следовать тесту Тьюки и построить гистограммы для каждого гена (ответ против времени; заполненный обоими генотипами) с несколькими буквами сравнения.
Синтаксис
df1 <- df %>%
gather(var, response, WT_Tissue_0T_1:mut1_Tissue_5T_3) %>%
separate(var, c("Genotype", "Tissue", "Time"), sep = "_") %>%
arrange(desc(Gene))
df2 <- df1 %>%
group_by(`Gene`,Genotype,Tissue,Time) %>%
mutate(Response=mean(response),n=n(),se=sd(response)/sqrt(n))
Двухсторонний ANOVA
fit1 <- aov(Response ~ Genotype*Time, df2)
Далее я хочу перейти к тесту Тьюки (множественное сравнение), например, для гена "AGI4120.1_UBQ", построить график зависимости ответа от времени и посмотреть, как ведет себя каждый генотип (WT & mut1) в каждой временной точке (0T, 1T, 3T). и 5т)? если ответ существенно отличается или нет и обозначается буквами на графике.
Как показано ниже, синтаксис lsmeans объединяет все гены в один и дает вывод, как я могу сделать так, чтобы он зацикливался для каждого гена отдельно (то есть "AGI4120.1_UBQ", "AGI570.1_Acin") и получал буквы, чтобы показать статистически различные группы (иначе) компактный буквенный дисплей ")
lsmeans(fit1, pairwise ~ Genotype | Time)
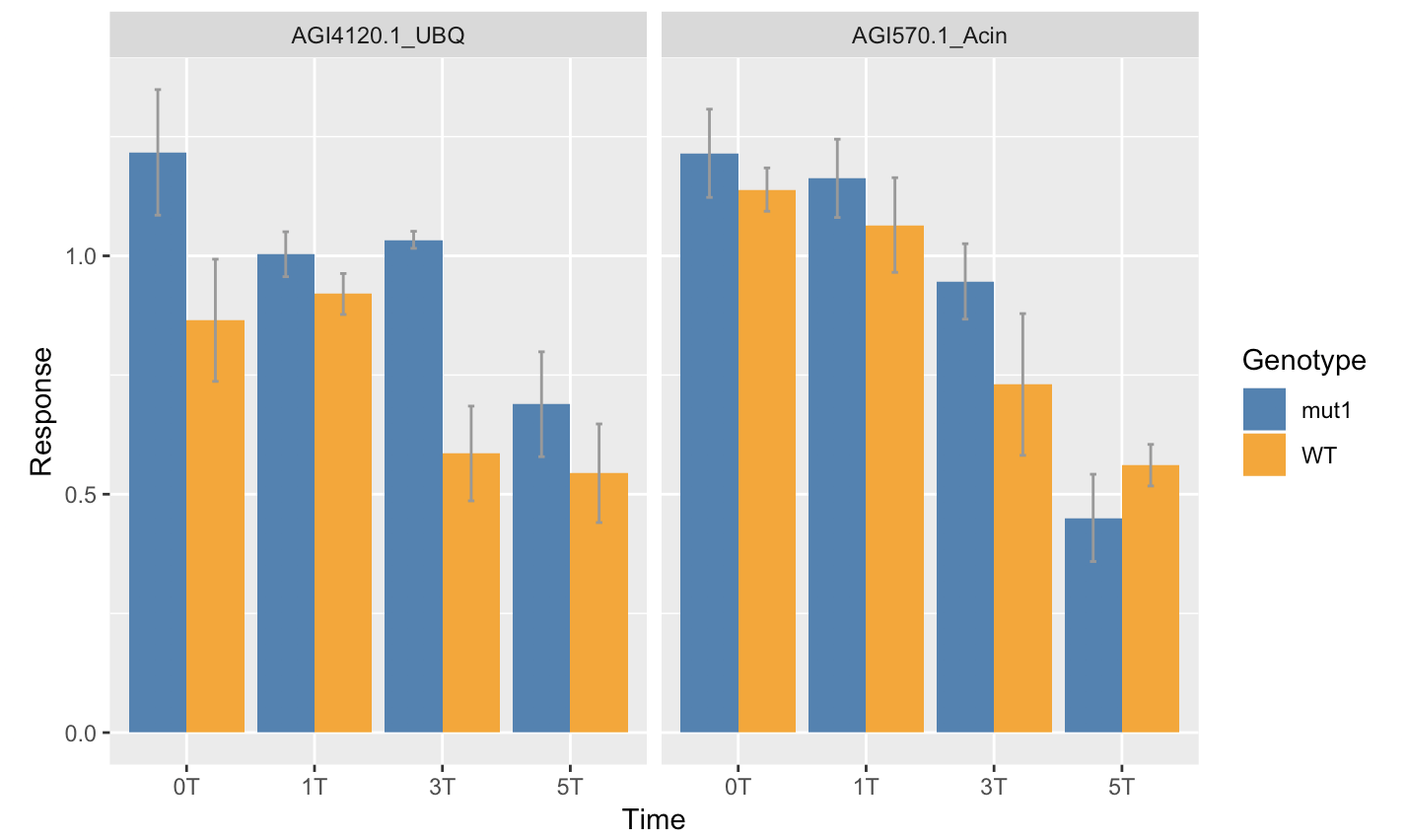
Моя конечная цель состоит в том, чтобы построить каждый ген, как на графике ниже, и обозначить значимые буквы.
df2$genotype <- factor(df2$genotype, levels = c("WT","mut1")) colours <- c('#336600','#ffcc00')library(ggplot2)ggplot(df2,aes( x=Time, y=Response, fill=Genotype))+geom_bar(stat='identity', position='dodge')+scale_fill_manual(values=colours)+geom_errorbar(aes(ymin=average_measure-se, ymax=average_measure+se)+facet_wrap(~`Gene`)+labs(x='Time', y='Response')
Ожидается График для обозначения компактного отображения букв
Буду признателен за помощь, если это возможно.
1 ответ
Есть несколько проблем с вашим кодом. Я бы сказал, что это не совсем подходящая статья для Stackru, поскольку проблемы здесь разнообразны и не могут быть расширены за пределы этого специфического сочетания ошибок и синтаксических проблем. Но я отправлю несколько предложений в качестве ответа, чтобы вы начали - надеюсь, они помогут.
1. lsmeans() :
Эта функция ожидает подгонки модели (например, из lm()) или ref.grid объект. Но вы передаете ему фрейм данных без каких-либо свойств регрессии, необходимых для вычисления наименьших квадратов. (Подумай: как lsmeans() знать, какой должна быть зависимая переменная, когда вы запрашиваете парное сравнение с Genes в качестве независимой переменной?) Проверьте использование lsmeans документация для более подробной информации.
Исходя из ваших данных, я бы сказал, что вы, вероятно, хотите запустить многоуровневую регрессию, используя что-то вроде lme4 пакет, с Gene а также Genotype и возможно Time как вложенные уровни группировки.
Но для демонстрации я буду держать это просто с lm(), Передача подогнанного объекта регрессии lsmeans() работает как задумано:
fit <- lm(Response ~ Gene + Genotype + Time, data=df2)
lsmeans(fit, pairwise ~ Gene)[[2]]
# Output
contrast estimate SE df t.ratio p.value
AGI4120.1_UBQ - AGI570.1_Acin -0.0515123 0.0299492 42 -1.72 0.0928
Results are averaged over the levels of: Genotype, Time
2. ggplot() :
Вы не определили colours или же average_measure в коде, который вы предоставили; вызов этих необъявленных переменных приведет к сбою.
Конструктивно я бы предложил вам использовать df1 и разрешить ggplot делать группировку, а не group_by в df2, Тогда используйте stat="summary" а также fun.y="mean" для достижения summarise() вычисления, которые вы сделали в df2, Это также позволяет вам использовать mean_se Функция для ваших ошибок. Как это:
ggplot(df1,aes( x=Time, y=response, fill=Genotype))+
geom_bar(stat='summary', fun.y='mean', position=position_dodge(0.9))+
stat_summary(fun.data = mean_se, geom = "errorbar",
color="gray60", width=.1, position=position_dodge(0.9)) +
scale_fill_manual(values=c("steelblue","orange"))+
facet_wrap(~`Gene`)+
labs(x='Time', y='Response')
Наконец, обратите внимание, что ваше использование separate() в df1 выдаст предупреждение, хотя и не будет ошибкой, так как при разбиении на sep="_", Вы можете избежать этого (если это вызывает путаницу), добавив уровень для захвата окончательного значения (которое выглядит как временной индекс):
separate(var, c("Genotype", "Tissue", "Time", "Index"), sep = "_")