Как быстро загрузить данные в R?
У меня есть несколько сценариев R, где я должен загрузить несколько кадров данных в R как можно быстрее. Это очень важно, так как чтение данных является самой медленной частью процедуры. Например: построение графиков с разных информационных фреймов. Я получаю данные в сохраненном (SPSS) формате, но я могу преобразовать их в любой формат, как предложено. Слияние фреймов данных, к сожалению, не вариант.
Какой может быть самый быстрый способ загрузки данных? Я думал о следующем:
- преобразовать из sav в двоичный объект R (Rdata) в первый раз, а затем всегда загружать его, так как это кажется намного быстрее, чем
read.spss, - преобразование из сохраненных в CSV- файлы и чтение данных из файлов с заданными параметрами, которые обсуждались в этом разделе
- или стоит установить бэкэнд MySQL на localhost и загружать данные с него? Может ли это быть быстрее? Если да, могу ли я также сохранить любой пользовательский
attrзначения переменных (например, variable.labels из импортированных файлов Spss)? Или это должно быть сделано в отдельной таблице?
Любые другие мысли приветствуются. Спасибо за каждое предложение заранее!
Ниже я провел небольшой эксперимент на основе ответов, которые вы дали, а также добавил (24/01/2011) довольно "хакерское", но очень быстрое решение, загружающее только несколько переменных / столбцов из специального двоичного файла. Последний, похоже, самый быстрый метод, который я могу себе представить, поэтому я создал (05/03/2011: версия 0.3) небольшой пакет с именем saves для работы с этой функцией. Пакет находится в стадии "тяжелой" разработки, любая рекомендация приветствуется!
Я скоро опубликую виньетку с точными результатами тестов с помощью пакета микробенчмарков.
4 ответа
Это зависит от того, что вы хотите сделать и как вы обрабатываете данные дальше. В любом случае загрузка из двоичного объекта R всегда будет быстрее, если вам всегда нужен один и тот же набор данных. Ограничение скорости здесь - это скорость вашего жесткого диска, а не R. Бинарная форма - это внутреннее представление кадра данных в рабочей области, поэтому преобразование больше не требуется.
Любой вид текстового файла - это отдельная история, поскольку вы всегда включаете накладные расходы: каждый раз, когда вы читаете текстовый файл, данные должны быть преобразованы в двоичный объект R. Я забуду о них. Они полезны только для переноса наборов данных из одного приложения в другое.
Настройка бэкэнда MySQL очень полезна, если вам нужны разные части данных или разные подмножества в разных комбинациях. Особенно при работе с огромными наборами данных тот факт, что вам не нужно загружать весь набор данных, прежде чем вы сможете начать выбирать строки / столбцы, может выиграть у вас довольно много времени. Но это работает только с огромными наборами данных, поскольку чтение двоичного файла происходит немного быстрее, чем поиск в базе данных.
Если данные не слишком велики, вы можете сохранить разные кадры данных в одном файле RData, что даст вам возможность упростить процесс. У меня часто есть набор данных в списке или в отдельной среде (см. Также ?environment за несколько простых примеров). Это позволяет lapply / eapply решения для обработки нескольких кадров данных одновременно.
Спасибо всем за советы и ответы, я сделал некоторые резюме и эксперимент на основе этого.
См. Небольшой тест с общедоступной базой данных ( ESS 2008 в Венгрии) ниже. В базе данных 1508 наблюдений и 508 переменных, так что это могут быть данные среднего размера. Это может быть хорошим примером для проведения теста (для меня), но, конечно, особые потребности потребуют эксперимента с адекватными данными.
Чтение данных из сохраненного файла SPSS без каких-либо изменений:
> system.time(data <- read.spss('ESS_HUN_4.sav'))
user system elapsed
2.214 0.030 2.376
Загрузка с преобразованным двоичным объектом:
> save('data',file='ESS_HUN_4.Rdata')
> system.time(data.Rdata <- load('ESS_HUN_4.Rdata'))
user system elapsed
0.28 0.00 0.28
Попытка с CSV:
> write.table(data, file="ESS_HUN_4.csv")
> system.time(data.csv <- read.csv('ESS_HUN_4.csv'))
user system elapsed
1.730 0.010 1.824
Попытка с "точно настроенной" загрузкой CSV:
> system.time(data.csv <- read.table('ESS_HUN_4.csv', comment.char="", stringsAsFactors=FALSE, sep=","))
user system elapsed
1.296 0.014 1.362
Также с пакетом sqldf, который, кажется, загружает CSV-файлы намного быстрее:
> library(sqldf)
> f <- file("ESS_HUN_4.csv")
> system.time(bigdf <- sqldf("select * from f", dbname = tempfile(), file.format = list(header = T, row.names = F, sep="\t")))
user system elapsed
0.939 0.106 1.071
А также загрузка данных из базы данных MySQL, работающей на localhost:
> library(RMySQL)
> con <- dbConnect(MySQL(), user='root', dbname='test', host='localhost', password='')
> dbWriteTable(con, "data", as.data.frame(data), overwrite = TRUE)
> system.time(data <- dbReadTable(con, 'data'))
user system elapsed
0.583 0.026 1.055
> query <-('SELECT * FROM data')
> system.time(data.sql <- dbGetQuery(con, query))
user system elapsed
0.270 0.020 0.473
Здесь, я думаю, мы должны добавить два system.time Сообщается, что подключение к данным также считается в нашем случае. Пожалуйста, прокомментируйте, если я что-то не так понял.
Но давайте посмотрим, запрашивает ли только некоторые переменные, например. при построении графика нам не нужны все кадры данных в большинстве случаев, и для создания хорошего графика их достаточно запросить только две переменные:
> query <-('SELECT c1, c19 FROM data')
> system.time(data.sql <- dbGetQuery(con, query))
user system elapsed
0.030 0.000 0.112
Который кажется действительно отличным! Конечно, только после загрузки таблицы с dbReadTable
Резюме: ничто не сравнится с чтением целых данных из двоичного файла, но считывание только нескольких столбцов (или других отфильтрованных данных) из одной и тойже таблицы базы данных может быть также взвешено в некоторых особых случаях.
Среда тестирования: ноутбук HP 6715b (AMD X2 2 ГГц, 4 Гб DDR2) с недорогим твердотельным накопителем.
ОБНОВЛЕНИЕ (24/01/2011): я добавил довольно хакерский, но довольно "креативный" способ загрузки только нескольких столбцов двоичного объекта - который выглядит намного быстрее, чем любой метод, рассмотренный выше.
Имейте в виду: код будет выглядеть очень плохо, но все же очень эффективно:)
Сначала я сохраняю все столбцы объекта data.frame в разные двоичные объекты с помощью следующего цикла:
attach(data)
for (i in 1:length(data)) {
save(list=names(data)[i],file=paste('ESS_HUN_4-', names(data)[i], '.Rdata', sep=''))
}
detach(data)
И тогда я загружаю два столбца данных:
> system.time(load('ESS_HUN_4-c19.Rdata')) +
> system.time(load('ESS_HUN_4-c1.Rdata')) +
> system.time(data.c1_c19 <- cbind(c1, c19))
user system elapsed
0.003 0.000 0.002
Который выглядит как "сверхбыстрый" метод!:) Примечание: он был загружен в 100 раз быстрее, чем самый быстрый (загружающий весь двоичный объект) метод, описанный выше.
Я составил очень крошечный пакет (названный: saves), поищите подробности в github, если интересно.
ОБНОВЛЕНИЕ (03/03/2011): новая версия моего маленького пакета ( сохранения) была загружена в CRAN, в которой можно сохранять и загружать переменные еще быстрее - если только пользователю требуется только подмножество доступных переменных в фрейм данных или список. Подробности смотрите в виньетке в источниках пакета или на моей домашней странице, и позвольте мне также представить хороший блокпост с некоторыми сделанными тестами:
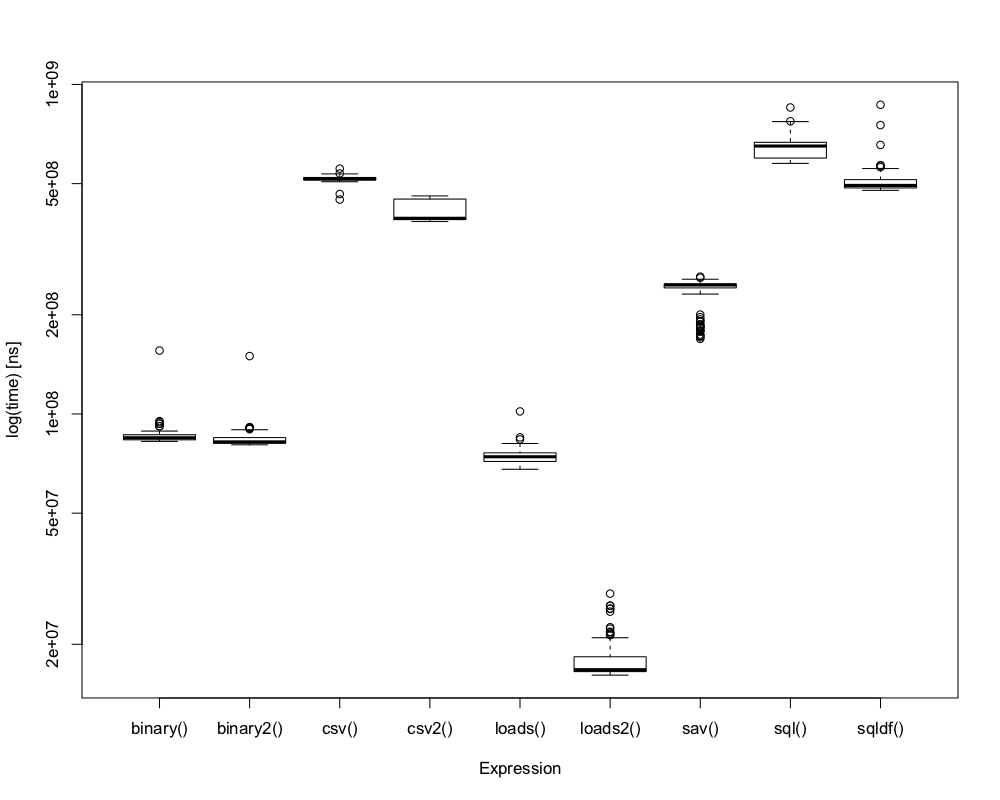
Этот блок-график показывает преимущество использования пакета saves для загрузки только поднабора переменных против load а также read.table или же read.csv с базы, read.spss от иностранного или sqldf или же RMySQL пакеты.
Я очень доволен RMySQL. Я не уверен, правильно ли я понял ваш вопрос, но ярлыки не должны быть проблемой. Есть несколько вспомогательных функций, которые просто используют таблицу SQL и имена строк по умолчанию, но, конечно, вы можете использовать некоторые операторы SQL.
Я бы сказал (кроме больших наборов данных, которые оправдывают суету), одной из основных причин использования RMySQL является более знакомое с синтаксисом SQL, чем с функциями жонглирования данными R. Лично я предпочитаю GROUP BY по совокупности. Обратите внимание, что использование хранимых процедур внутри R не работает особенно хорошо.
Итог... настройка локального хоста MySQL не так уж много усилий - попробуйте! Я не могу сказать точно о скорости, но у меня есть ощущение, что есть шанс, что это быстрее. Тем не менее, я постараюсь вернуться сюда.
РЕДАКТИРОВАТЬ: вот тест... и победитель: spacedman
# SQL connection
source("lib/connect.R")
dbQuery <- "SELECT * FROM mytable"
mydata <- dbGetQuery(con,dbQuery)
system.time(dbGetQuery(con,dbQuery))
# returns
#user system elapsed
# 0.999 0.213 1.715
save.image(file="speedtest.Rdata")
system.time(load("speedtest.Rdata"))
#user system elapsed
#0.348 0.006 0.358
Размер файла здесь был всего около 1 МБ. MacBook Pro, 4 ГБ, оперативная память 2,4 ГГц, Intel Core Duo, Mac OSX 10.6.4, MySQL 5.0.41. Никогда не пробовал, потому что я обычно работаю с большим набором данных, и загрузка - это не проблема, а скорее обработка... если есть проблемы со временем совсем. +1 за Q!
Если это вообще возможно, преобразовать данные в csv или другой "простой" формат, чтобы сделать чтение максимально быстрым (см. ответ Йориса). Я импортирую csv файлы в массе с apply функция, что-то вроде:
list.of.files <- as.list(list.files("your dir"))
lapply(list.of.files, FUN = function(x) {
my.object <- read.table(...) # or some other function, like read.spss
})