Быстрое чтение очень больших таблиц как данных
У меня есть очень большие таблицы (30 миллионов строк), которые я хотел бы загрузить в виде фреймов данных в R. read.table() имеет много удобных функций, но кажется, что в реализации есть много логики, которая замедлит процесс. В моем случае, я предполагаю, что знаю типы столбцов заранее, таблица не содержит заголовков столбцов или имен строк и не содержит патологических символов, о которых мне нужно беспокоиться.
Я знаю, что чтение в таблице в виде списка, используя scan() может быть довольно быстрым, например:
datalist <- scan('myfile',sep='\t',list(url='',popularity=0,mintime=0,maxtime=0)))
Но некоторые из моих попыток преобразовать это в фрейм данных, похоже, снижают производительность вышеупомянутого в 6 раз:
df <- as.data.frame(scan('myfile',sep='\t',list(url='',popularity=0,mintime=0,maxtime=0))))
Есть ли лучший способ сделать это? Или, может быть, совершенно другой подход к проблеме?
12 ответов
Обновление, несколько лет спустя
Этот ответ старый, и R перешел. доводка read.table бегать немного быстрее имеет очень мало пользы. Ваши варианты:
С помощью
freadвdata.tableдля импорта данных из CSV / Tab-файлов с разделителями непосредственно в R. См . ответ mnel.С помощью
read_tableвreadr(на КРАНе с апреля 2015 года). Это работает так же, какfreadвыше. Readme в ссылке объясняет разницу между двумя функциями (readrв настоящее время утверждает, что "в 1,5-2 раза медленнее", чемdata.table::fread).read.csv.rawотiotoolsпредоставляет третий вариант для быстрого чтения файлов CSV.Попытка сохранить как можно больше данных в базах данных, а не в простых файлах. (Помимо того, что это лучший постоянный носитель данных, данные передаются в и из R в двоичном формате, что быстрее).
read.csv.sqlвsqldfПакет, как описано в ответе Дж. Д. Лонга, импортирует данные во временную базу данных SQLite и затем считывает их в R. См. также:RODBCпакет, и обратное зависит от разделаDBIстраница пакета.MonetDB.Rдает вам тип данных, который выглядит как фрейм данных, но на самом деле представляет собой MonetDB, что повышает производительность. Импорт данных с егоmonetdb.read.csvфункция.dplyrпозволяет работать непосредственно с данными, хранящимися в нескольких типах баз данных.Хранение данных в двоичных форматах также может быть полезно для повышения производительности. использование
saveRDS/readRDS(см. ниже),h5или жеrhdf5пакеты для формата HDF5, илиwrite_fst/read_fstотfstпакет.
Оригинальный ответ
Есть несколько простых вещей, которые вы можете попробовать, используете ли вы read.table или scan.
Задавать
nrows= количество записей в ваших данных (nmaxвscan).Удостоверься что
comment.char=""отключить интерпретацию комментариев.Явно определите классы каждого столбца, используя
colClassesвread.table,настройка
multi.line=FALSEможет также улучшить производительность при сканировании.
Если ничего из этого не работает, то используйте один из пакетов профилирования, чтобы определить, какие строки замедляют работу. Возможно, вы можете написать урезанную версию read.table на основании результатов.
Другой альтернативой является фильтрация ваших данных, прежде чем читать их в R.
Или, если проблема заключается в том, что вы должны регулярно читать его, затем используйте эти методы для чтения данных за один раз, а затем сохраните кадр данных в виде двоичного двоичного объекта с savesaveRDS затем в следующий раз вы сможете получить его быстрее с loadreadRDS,
Вот пример, который использует fread от data.table 1.8.7
Примеры приходят со страницы справки на fread, с временами на моем Windows XP Core 2 Duo E8400.
library(data.table)
# Demo speedup
n=1e6
DT = data.table( a=sample(1:1000,n,replace=TRUE),
b=sample(1:1000,n,replace=TRUE),
c=rnorm(n),
d=sample(c("foo","bar","baz","qux","quux"),n,replace=TRUE),
e=rnorm(n),
f=sample(1:1000,n,replace=TRUE) )
DT[2,b:=NA_integer_]
DT[4,c:=NA_real_]
DT[3,d:=NA_character_]
DT[5,d:=""]
DT[2,e:=+Inf]
DT[3,e:=-Inf]
стандартный read.table
write.table(DT,"test.csv",sep=",",row.names=FALSE,quote=FALSE)
cat("File size (MB):",round(file.info("test.csv")$size/1024^2),"\n")
## File size (MB): 51
system.time(DF1 <- read.csv("test.csv",stringsAsFactors=FALSE))
## user system elapsed
## 24.71 0.15 25.42
# second run will be faster
system.time(DF1 <- read.csv("test.csv",stringsAsFactors=FALSE))
## user system elapsed
## 17.85 0.07 17.98
оптимизированный read.table
system.time(DF2 <- read.table("test.csv",header=TRUE,sep=",",quote="",
stringsAsFactors=FALSE,comment.char="",nrows=n,
colClasses=c("integer","integer","numeric",
"character","numeric","integer")))
## user system elapsed
## 10.20 0.03 10.32
Fread
require(data.table)
system.time(DT <- fread("test.csv"))
## user system elapsed
## 3.12 0.01 3.22
sqldf
require(sqldf)
system.time(SQLDF <- read.csv.sql("test.csv",dbname=NULL))
## user system elapsed
## 12.49 0.09 12.69
# sqldf as on SO
f <- file("test.csv")
system.time(SQLf <- sqldf("select * from f", dbname = tempfile(), file.format = list(header = T, row.names = F)))
## user system elapsed
## 10.21 0.47 10.73
фф / ффдф
require(ff)
system.time(FFDF <- read.csv.ffdf(file="test.csv",nrows=n))
## user system elapsed
## 10.85 0.10 10.99
В итоге:
## user system elapsed Method
## 24.71 0.15 25.42 read.csv (first time)
## 17.85 0.07 17.98 read.csv (second time)
## 10.20 0.03 10.32 Optimized read.table
## 3.12 0.01 3.22 fread
## 12.49 0.09 12.69 sqldf
## 10.21 0.47 10.73 sqldf on SO
## 10.85 0.10 10.99 ffdf
Сначала я не видел этот вопрос и через несколько дней задал похожий вопрос. Я собираюсь снять свой предыдущий вопрос, но я решил добавить ответ, чтобы объяснить, как я использовал sqldf() сделать это.
Там было немного дискуссий о том, как лучше всего импортировать 2 ГБ или более текстовых данных во фрейм данных R. Вчера я написал сообщение в блоге об использовании sqldf() импортировать данные в SQLite в качестве промежуточной области, а затем всасывать их из SQLite в R. Это очень хорошо работает для меня. Я смог получить 2 ГБ (3 столбца, 40 мм строки) данных за < 5 минут. В отличие от read.csv Команда бежала всю ночь и никогда не выполнялась.
Вот мой тестовый код:
Настройте тестовые данные:
bigdf <- data.frame(dim=sample(letters, replace=T, 4e7), fact1=rnorm(4e7), fact2=rnorm(4e7, 20, 50))
write.csv(bigdf, 'bigdf.csv', quote = F)
Я перезапустил R перед запуском следующей процедуры импорта:
library(sqldf)
f <- file("bigdf.csv")
system.time(bigdf <- sqldf("select * from f", dbname = tempfile(), file.format = list(header = T, row.names = F)))
Я позволил следующей строке работать всю ночь, но она так и не завершилась:
system.time(big.df <- read.csv('bigdf.csv'))
Как ни странно, никто не отвечал на нижнюю часть вопроса в течение многих лет, хотя это важный вопрос - data.frameЭто просто списки с правильными атрибутами, поэтому если у вас есть большие данные, которые вы не хотите использовать as.data.frame или подобное для списка. Гораздо быстрее просто "превратить" список в фрейм данных на месте:
attr(df, "row.names") <- .set_row_names(length(df[[1]]))
class(df) <- "data.frame"
Это не делает копию данных, поэтому она немедленная (в отличие от всех других методов). Предполагается, что вы уже установили names() в списке соответственно.
[Что касается загрузки больших данных в R - лично я дам их по столбцам в двоичные файлы и использую readBin() - это самый быстрый метод (кроме mmapping), который ограничен только скоростью диска. Анализ файлов ASCII по своей сути медленен (даже в C) по сравнению с двоичными данными.]
Ранее об этом спрашивали в R-Help, так что стоит ознакомиться.
Одно из предложений было использовать readChar() а затем выполните строковые манипуляции с результатом с strsplit() а также substr(), Вы можете видеть, что логика readChar намного меньше, чем read.table.
Я не знаю, является ли здесь проблема с памятью, но вы также можете взглянуть на пакет HadoopStreaming. При этом используется Hadoop, который представляет собой инфраструктуру MapReduce, предназначенную для работы с большими наборами данных. Для этого вы бы использовали функцию hsTableReader. Это пример (но у него есть кривая обучения, чтобы выучить Hadoop):
str <- "key1\t3.9\nkey1\t8.9\nkey1\t1.2\nkey1\t3.9\nkey1\t8.9\nkey1\t1.2\nkey2\t9.9\nkey2\"
cat(str)
cols = list(key='',val=0)
con <- textConnection(str, open = "r")
hsTableReader(con,cols,chunkSize=6,FUN=print,ignoreKey=TRUE)
close(con)
Основная идея здесь состоит в том, чтобы разбить импорт данных на куски. Вы могли бы даже пойти так далеко, чтобы использовать одну из параллельных структур (например, Snow) и запустить импорт данных параллельно, сегментируя файл, но, скорее всего, для больших наборов данных, которые не помогут, поскольку вы столкнетесь с ограничениями памяти, именно поэтому карта-сокращение является лучшим подходом.
Альтернативой является использование vroom пакет. Теперь на CRAN.vroom не загружает весь файл, он индексирует, где находится каждая запись, и читается позже, когда вы его используете.
Платите только за то, что вы используете.
См. Введение в vroom, Начало работы с vroom и тесты vroom.
Основной обзор заключается в том, что первоначальное чтение огромного файла будет намного быстрее, а последующие изменения данных могут быть немного медленнее. Таким образом, в зависимости от того, что вы используете, это может быть лучшим вариантом.
Посмотрите упрощенный пример из тестов vroom ниже, ключевые моменты, которые нужно увидеть, - это супер быстрое время чтения, но немного ускоряющие операции, такие как агрегирование и т. Д.
package read print sample filter aggregate total
read.delim 1m 21.5s 1ms 315ms 764ms 1m 22.6s
readr 33.1s 90ms 2ms 202ms 825ms 34.2s
data.table 15.7s 13ms 1ms 129ms 394ms 16.3s
vroom (altrep) dplyr 1.7s 89ms 1.7s 1.3s 1.9s 6.7s
Я очень быстро читаю данные с помощью нового arrowпакет. Похоже, он находится на довольно ранней стадии.
В частности, я использую столбчатый формат паркета. Это преобразуется обратно вdata.frameв R, но вы можете получить еще большее ускорение, если этого не сделаете. Этот формат удобен, так как его можно использовать и из Python.
Мой основной вариант использования для этого - на довольно ограниченном сервере RShiny. По этим причинам я предпочитаю хранить данные прикрепленными к приложениям (т. Е. Вне SQL), и поэтому требую небольшой размер файла, а также скорость.
В этой связанной статье представлен сравнительный анализ и хороший обзор. Ниже я привел несколько интересных моментов.
https://ursalabs.org/blog/2019-10-columnar-perf/
Размер файла
То есть файл Parquet вдвое меньше даже сжатого с помощью gzip CSV. Одна из причин того, что файл Parquet такой малый, - это кодирование по словарю (также называемое "словарным сжатием"). Сжатие словаря может дать значительно лучшее сжатие, чем использование компрессора байтов общего назначения, такого как LZ4 или ZSTD (которые используются в формате FST). Parquet был разработан для создания очень маленьких файлов, которые быстро читаются.
Скорость чтения
При управлении по типу вывода (например, при сравнении всех выходных данных R data.frame друг с другом) мы видим, что производительность Parquet, Feather и FST находится в относительно небольшом диапазоне друг от друга. То же самое и с выводами pandas.DataFrame. data.table::fread впечатляюще конкурирует с размером файла 1,5 ГБ, но отстает от других на 2,5 ГБ CSV.
Независимый тест
Я провел несколько независимых сравнительных тестов на смоделированном наборе данных из 1000000 строк. В основном я перетасовал кучу вещей, чтобы попытаться оспорить сжатие. Также я добавил короткое текстовое поле из случайных слов и два смоделированных фактора.
Данные
library(dplyr)
library(tibble)
library(OpenRepGrid)
n <- 1000000
set.seed(1234)
some_levels1 <- sapply(1:10, function(x) paste(LETTERS[sample(1:26, size = sample(3:8, 1), replace = TRUE)], collapse = ""))
some_levels2 <- sapply(1:65, function(x) paste(LETTERS[sample(1:26, size = sample(5:16, 1), replace = TRUE)], collapse = ""))
test_data <- mtcars %>%
rownames_to_column() %>%
sample_n(n, replace = TRUE) %>%
mutate_all(~ sample(., length(.))) %>%
mutate(factor1 = sample(some_levels1, n, replace = TRUE),
factor2 = sample(some_levels2, n, replace = TRUE),
text = randomSentences(n, sample(3:8, n, replace = TRUE))
)
Прочти и напиши
Запись данных проста.
library(arrow)
write_parquet(test_data , "test_data.parquet")
# you can also mess with the compression
write_parquet(test_data, "test_data2.parquet", compress = "gzip", compression_level = 9)
Считывать данные также легко.
read_parquet("test_data.parquet")
# this option will result in lightning fast reads, but in a different format.
read_parquet("test_data2.parquet", as_data_frame = FALSE)
Я протестировал чтение этих данных с несколькими конкурирующими вариантами и получил несколько иные результаты, чем в статье выше, чего и следовало ожидать.
Этот файл далеко не такой большой, как статья о тесте, так что, возможно, в этом разница.
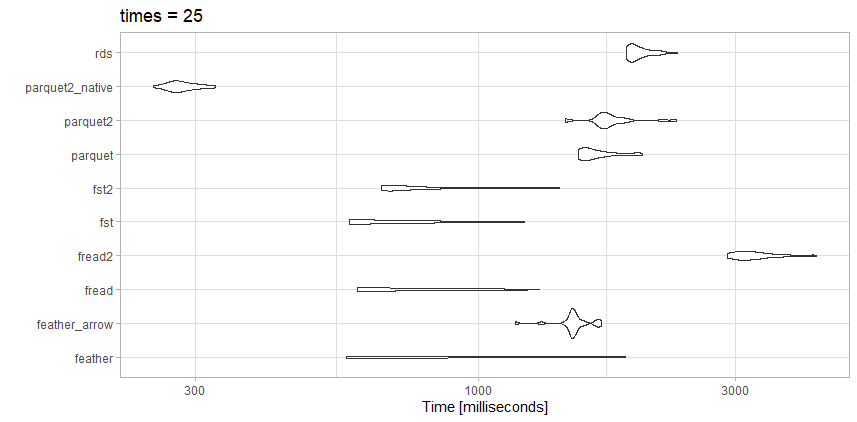
Тесты
- rds : test_data.rds (20,3 МБ)
- parquet2_native: (14,9 МБ с большим сжатием и
as_data_frame = FALSE) - parquet2: test_data2.parquet (14,9 МБ с большим сжатием)
- паркет: test_data.parquet (40,7 МБ)
- fst2: test_data2.fst (27,9 МБ с большим сжатием)
- fst : test_data.fst (76,8 МБ)
- fread2: test_data.csv.gz (23,6 МБ)
- fread: test_data.csv (98,7 МБ)
- Feather_arrow: test_data.feather (157,2 МБ при чтении
arrow) - перо: test_data.feather (157,2 МБ при чтении
feather)
Наблюдения
Для этого конкретного файла freadна самом деле очень быстро. Мне нравится небольшой размер файла из сильно сжатогоparquet2контрольная работа. Я могу потратить время на работу с собственным форматом данных, а не сdata.frame если мне действительно нужна скорость.
Вот fstтоже отличный выбор. Я бы использовал сильно сжатыйfst формат или сильно сжатый parquet в зависимости от того, нужен ли мне компромисс между скоростью или размером файла.
Незначительные дополнительные моменты стоит упомянуть. Если у вас очень большой файл, вы можете на лету рассчитать количество строк (если нет заголовка), используя (где bedGraph имя вашего файла в вашем рабочем каталоге):
>numRow=as.integer(system(paste("wc -l", bedGraph, "| sed 's/[^0-9.]*\\([0-9.]*\\).*/\\1/'"), intern=T))
Затем вы можете использовать это либо в read.csv, read.table...
>system.time((BG=read.table(bedGraph, nrows=numRow, col.names=c('chr', 'start', 'end', 'score'),colClasses=c('character', rep('integer',3)))))
user system elapsed
25.877 0.887 26.752
>object.size(BG)
203949432 bytes
Часто я думаю, что это хорошая практика - хранить большие базы данных внутри базы данных (например, Postgres). Я не использую ничего слишком большого, чем (nrow * ncol) ncell = 10M, что довольно мало; но я часто нахожу, что хочу, чтобы R создавал и содержал графики, интенсивно использующие память, только когда я выполняю запросы из нескольких баз данных В будущем 32-ГБ ноутбуки некоторые из этих типов проблем с памятью исчезнут. Но привлекательность использования базы данных для хранения данных и последующего использования памяти R для получения результатов и графиков запросов все еще может быть полезной. Некоторые преимущества:
(1) Данные остаются загруженными в вашу базу данных. Вы просто переподключаетесь в pgadmin к нужным базам данных, когда снова включаете свой ноутбук.
(2) Это правда, что R может выполнять намного больше изящных статистических и графических операций, чем SQL. Но я думаю, что SQL лучше предназначен для запросов больших объемов данных, чем R.
# Looking at Voter/Registrant Age by Decade
library(RPostgreSQL);library(lattice)
con <- dbConnect(PostgreSQL(), user= "postgres", password="password",
port="2345", host="localhost", dbname="WC2014_08_01_2014")
Decade_BD_1980_42 <- dbGetQuery(con,"Select PrecinctID,Count(PrecinctID),extract(DECADE from Birthdate) from voterdb where extract(DECADE from Birthdate)::numeric > 198 and PrecinctID in (Select * from LD42) Group By PrecinctID,date_part Order by Count DESC;")
Decade_RD_1980_42 <- dbGetQuery(con,"Select PrecinctID,Count(PrecinctID),extract(DECADE from RegistrationDate) from voterdb where extract(DECADE from RegistrationDate)::numeric > 198 and PrecinctID in (Select * from LD42) Group By PrecinctID,date_part Order by Count DESC;")
with(Decade_BD_1980_42,(barchart(~count | as.factor(precinctid))));
mtext("42LD Birthdays later than 1980 by Precinct",side=1,line=0)
with(Decade_RD_1980_42,(barchart(~count | as.factor(precinctid))));
mtext("42LD Registration Dates later than 1980 by Precinct",side=1,line=0)
Я хотел внести свой вклад в решение на основе Spark в самой простой форме:
# Test Data ---------------------------------------------------------------
set.seed(123)
bigdf <-
data.frame(
dim = sample(letters, replace = T, 4e7),
fact1 = rnorm(4e7),
fact2 = rnorm(4e7, 20, 50)
)
tmp_csv <- fs::file_temp(pattern = "big_df", ext = ".csv")
readr::write_csv(x = bigdf, file = tmp_csv)
# Spark -------------------------------------------------------------------
# Installing if needed
# sparklyr::spark_available_versions()
# sparklyr::spark_install()
library("sparklyr")
sc <- spark_connect(master = "local")
# Uploading CSV
system.time(tbl_big_df <- spark_read_csv(sc = sc, path = tmp_csv))
Spark дал довольно хорошие результаты:
>> system.time(tbl_big_df <- spark_read_csv(sc = sc, path = tmp_csv))
user system elapsed
0.278 0.034 11.747
Это было проверено на MacBook Pro с 32 ГБ оперативной памяти.
Примечания
Spark, как правило , не должен «выигрывать» с пакетами, оптимизированными для скорости. Тем не менее, я хотел дать ответ, используя Spark:
- Для некоторых комментариев и ответов, где процесс не работал, использование Spark может быть жизнеспособной альтернативой.
- В долгосрочной перспективе загрузка как можно большего количества данных может оказаться проблематичной позже, когда с этим объектом будут предприняты другие операции, и они достигнут предела производительности архитектуры.
Я думаю, что для таких вопросов, где задача состоит в том, чтобы обрабатывать 1e7 или более строк, Spark следует учитывать. Даже если есть возможность «вбить» эти данные в единый
data.frameэто просто неправильно. Скорее всего, с этим объектом будет сложно работать, возникнут проблемы при развертывании моделей и т. д.
Я перепробовал все вышеперечисленное, и [readr][1] справился лучше всех. У меня всего 8гб оперативной памяти
Цикл на 20 файлов по 5гб, 7 столбцов:
read_fwf(arquivos[i],col_types = "ccccccc",fwf_cols(cnpj = c(4,17), nome = c(19,168), cpf = c(169,183), fantasia = c(169,223), sit.cadastral = c(224,225), dt.sitcadastral = c(226,233), cnae = c(376,382)))
Вместо обычного read.table я чувствую, что fread - более быстрая функция. Задание дополнительных атрибутов, таких как выбор только необходимых столбцов, указание классов и строк в качестве факторов, уменьшит время, необходимое для импорта файла.
data_frame <- fread("filename.csv",sep=",",header=FALSE,stringsAsFactors=FALSE,select=c(1,4,5,6,7),colClasses=c("as.numeric","as.character","as.numeric","as.Date","as.Factor"))