Дерево решений Анализ вопроса
В настоящее время я работаю над проблемой с R. Я хочу применить дерево классификации к набору данных, но результат кажется неправильным, так как я уже решил ту же проблему, используя Weka, и получил другие результаты.
Я получил набор данных, содержащийся в CSV-файле следующим образом:
age,menopause,tumor.size,inv.nodes,node.caps,deg.malig,breast,breast.quad,irradiat,class
40-49,premeno,15-19,0-2,yes,3,right,left_up,no,recurrence-events
50-59,ge40,15-19,0-2,no,1,right,central,no,no-recurrence-events
50-59,ge40,35-39,0-2,no,2,left,left_low,no,recurrence-events
40-49,premeno,35-39,0-2,yes,3,right,left_low,yes,no-recurrence-events
40-49,premeno,30-34,3-5,yes,2,left,right_up,no,recurrence-events
и это сценарий:
#Open r file
cancer = read.csv("cancer.csv")
#Data Exploration
summary(cancer)
str(cancer)
#Divide into test and train sets
set.seed(1234)
ind <- sample(2, nrow(cancer), replace=TRUE, prob=c(0.7, 0.3))
trainData <- cancer[ind==1,]
testData <- cancer[ind==2,]
#Build the model
library(party)
cancermodel <- class ~ age + menopause + tumor.size + inv.nodes + node.caps + deg.malig + breast + breast.quad + irradiat
cancertree <- ctree(cancermodel,trainData)
table(predict(cancertree),trainData$class)
#Draw tree
plot(cancertree, type="simple")
#Testset
testPred <- predict(cancertree, newdata = testData)
table(testPred, testData$class)
2 ответа
Деревья решений имеют много разных реализаций алгоритмов в R (tree, rpart, party) и в Weka (J48, LMT, DecisionStump), и разные алгоритмы могут создавать разные деревья решений для одного и того же набора данных (некоторые работают, максимизируя прирост информации / индекс Джини некоторые используют тесты гипотез с хи-квадрат статистикой).
Даже любой данный алгоритм будет создавать разные деревья решений с разными входными параметрами (сокращенные / необработанные, минимальные # точки данных в узле для разделения и т. Д.).
Кроме того, как отмечает @RomRom, дерево решений не является очень надежной моделью, в том смысле, что небольшое изменение в обучающих данных может привести к совершенно другому дереву.
Учитывая все это, трудно создать одно и то же дерево решений в R и Weka, и даже если вы можете, вам нужно очень тщательно настроить параметры модели, что может потребовать большого количества экспериментов.
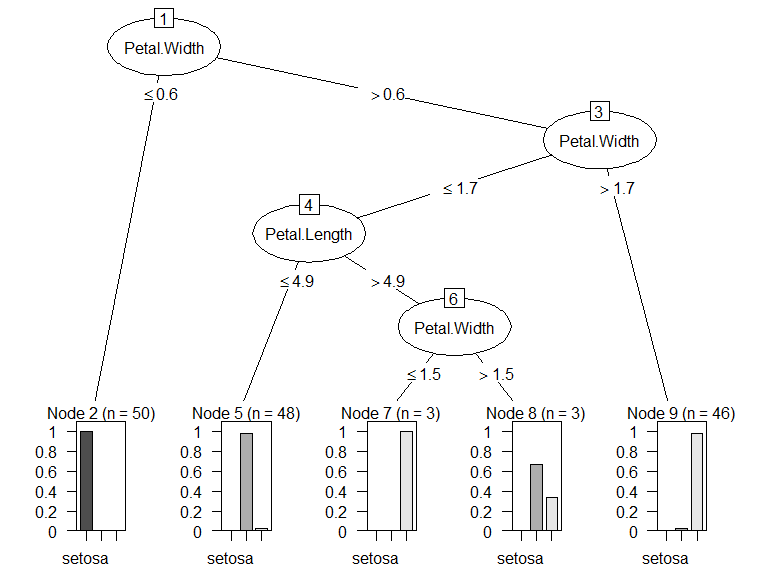
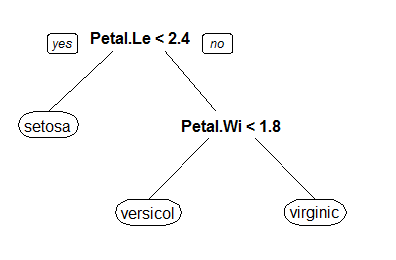
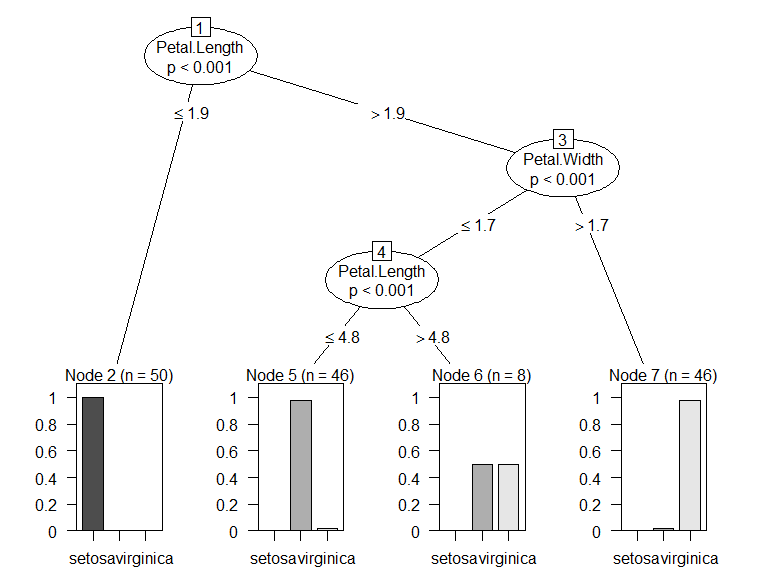
Ниже приведен пример набора данных радужной оболочки с несколькими моделями дерева решений R и моделями дерева решений RWeka (как видно из построенных деревьев, разные модели генерируют разные деревья с одинаковой радужной оболочкой набора данных).
library(RWeka)
m1 <- J48(Species ~ ., data = iris)
if(require("partykit", quietly = TRUE)) plot(m1)
library(rpart)
m2 <- rpart(Species ~ ., data = iris)
library(rpart.plot)
prp(m2)
library(party)
m3 <- ctree(Species ~ ., data = iris)
plot(m3)
Вы выбрали случайную выборку здесь в своем коде в пределах R: ind <- sample (2, nrow (рак), replace = TRUE, prob = c (0.7, 0.3))
как вы копировали и использовали одну и ту же случайную выборку в Weka? деревья - очень ненадежные модели и могут быстро меняться в зависимости от разных файлов данных.