Колеблющаяся скорость обработки в скрипте Python с использованием pysam.TabixFile для аннотирования операций чтения
Начальный вопрос
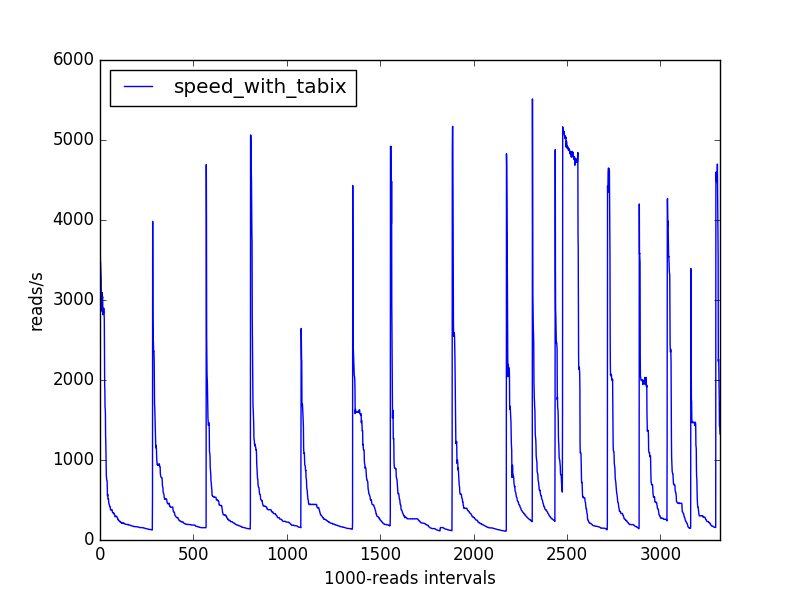
Я пишу скрипт биоинформатики в python (3.5), который анализирует большой (отсортированный и проиндексированный) файл bam, представляющий секвенирующие чтения, выровненные по геному, связывает геномную информацию ("аннотации") с этими чтениями и подсчитывает типы встреченных аннотаций., Я измеряю скорость, с которой мой сценарий обрабатывает выровненные операции чтения (более 1000 операций чтения), и получаю следующие вариации скорости:
Чем можно объяснить эту закономерность?
Моя интуиция заставила бы меня сделать ставку на некоторую структуру данных, которая постепенно становится медленнее, но становится все более и более время от времени.
Не похоже, что использование памяти является значительным (хотя после почти 2 часов работы мой сценарий все еще использует только 0,1% памяти моего компьютера, согласно htop).
Как работает мой код (фактический код см. В конце)
Я использую pysam модуль для разбора файла bam. Метод AlignmentFile.fetch дает мне итератор, предоставляющий информацию о последовательных выровненных чтениях в форме объектов AlignedSegment.
Я связываю аннотации с чтениями на основе их координат выравнивания и файла аннотации в формате gtf (сжатого с помощью bgzip и проиндексированного с помощью tabix). Я использую метод TabixFile.fetch (еще из pysam), чтобы получить эти аннотации, я отфильтровал их и вывел их резюме в виде frozenset строк (process_annotations, не показанный ниже, возвращает такой frozenset), в функции генератора, которая внутренне зацикливается на итераторе AlignedSegment.
Я кормлю сгенерированные заморозки Counter объект. Может ли счетчик отвечать за наблюдаемое поведение скорости?
Как я могу узнать, как избежать этих регулярных замедлений?
Дополнительные тесты
Следуя предложениям в комментариях, я профилировал весь свой анализ, используя cProfile и обнаружил, что больше всего времени было потрачено при доступе к данным аннотаций (pysam/ctabix.pyx:579(__cnext__)см. график вызовов позже), который, если я правильно понимаю, является некоторым кодом Cython, взаимодействующим с библиотеками s samtools. Казалось, причину наблюдаемого замедления будет трудно понять.
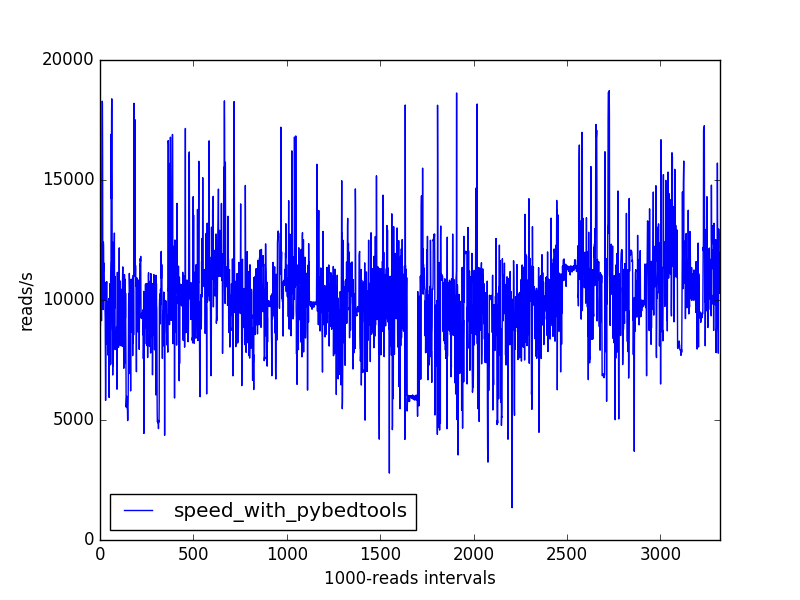
В попытке ускорить мой сценарий, я попробовал другое решение, основанное на pybedtools интерфейс python с bedtools, который также может извлекать аннотации из файлов gtf ( https://daler.github.io/pybedtools/index.html).
скорость
Улучшение скорости довольно важно. Вот фактические результаты команды и времени (два были фактически выполнены параллельно):
$ time python3 -m cProfile -o tests/total_pybedtools.prof ~/src/bioinfo_utils/small_RNA_seq_annotate.py -b results/bowtie2/mapped_C_elegans/WT_1_21-26_on_C_elegans_sorted.bam -g annotations/all_annotations.gtf -a "pybedtools" -l total_pybedtools.log > total_pybedtools.out
real 5m48.474s
user 5m48.204s
sys 0m1.336s
$ time python3 -m cProfile -o tests/total_tabix.prof ~/src/bioinfo_utils/small_RNA_seq_annotate.py -b results/bowtie2/mapped_C_elegans/WT_1_21-26_on_C_elegans_sorted.bam -g annotations/all_annotations.gtf.gz -a "tabix" -l total_tabix.log > total_tabix.out
real 195m40.990s
user 194m54.356s
sys 0m47.696s
(Следует отметить: результаты аннотации немного отличаются между двумя подходами. Может быть, я должен проверить, как я работаю с координатами.)
Профиль скорости не имеет ранее наблюдаемых длительных падений:
Моя проблема со скоростью решена, но я по-прежнему заинтересован в ретроспективе относительно того, почему подход, основанный на табиксах, имеет такие падения скорости. По этой причине я добавил теги "биоинформатика" и "samtools".
Графики звонков
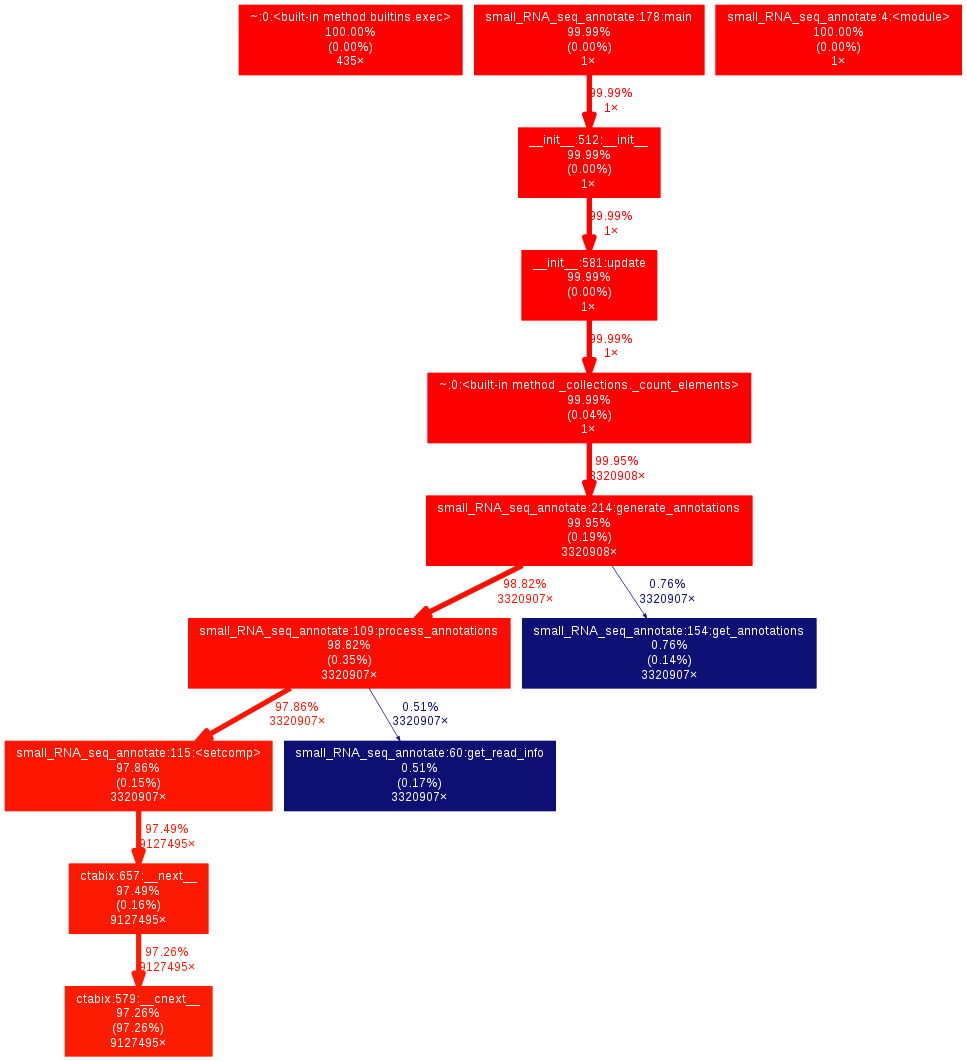
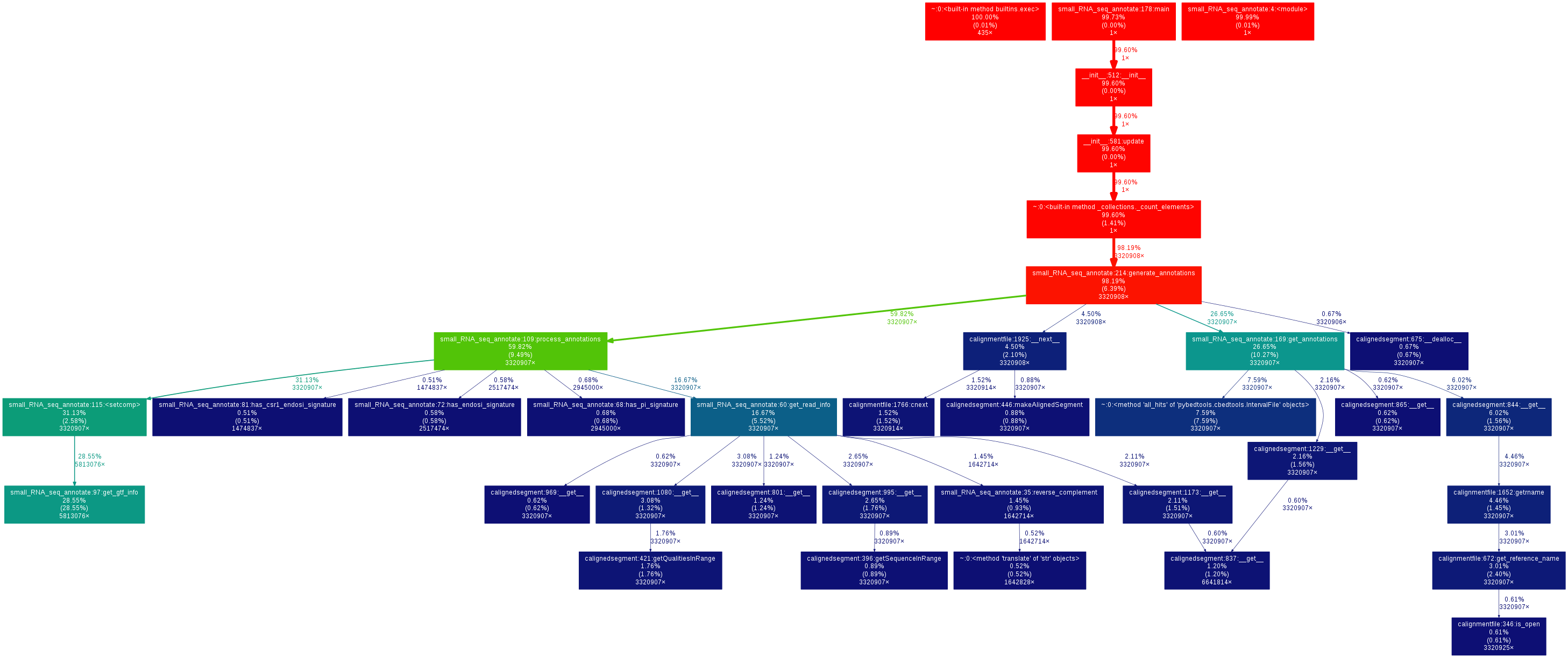
Для записи я сгенерировал графы вызовов, используя gprof2dot для результатов профилирования:
$ gprof2dot -f pstats tests/total_pybedtools.prof \
| dot -Tpng -o tests/total_pybedtools_callgraph.png
$ gprof2dot -f pstats tests/total_tabix.prof \
| dot -Tpng -o tests/total_tabix_callgraph.png
Вот график вызовов для подхода на основе табикса:
Для подхода на основе pybedtools:
Код
Вот основная часть моего текущего кода:
@contextmanager
def annotation_context(annot_file, getter_type):
"""Yields a function to get annotations for an AlignedSegment."""
if getter_type == "tabix":
gtf_parser = pysam.ctabix.asGTF()
gtf_file = pysam.TabixFile(annot_file, mode="r")
fetch_annotations = gtf_file.fetch
def get_annotations(ali):
"""Generates an annotation getter for *ali*."""
return fetch_annotations(*ALI2POS_INFO(ali), parser=gtf_parser)
elif getter_type == "pybedtools":
gtf_file = open(annot_file, "r")
# Does not work because somehow gets "consumed" after first usage
#fetch_annotations = BedTool(gtf_file).all_hits
# Much too slow
#fetch_annotations = BedTool(gtf_file.readlines()).all_hits
# https://daler.github.io/pybedtools/topical-low-level-ops.html
fetch_annotations = BedTool(gtf_file).as_intervalfile().all_hits
def get_annotations(ali):
"""Generates an annotation list for *ali*."""
return fetch_annotations(Interval(*ALI2POS_INFO(ali)))
else:
raise NotImplementedError("%s not available" % getter_type)
yield get_annotations
gtf_file.close()
def main():
"""Main function of the program."""
parser = argparse.ArgumentParser(
description=__doc__,
formatter_class=argparse.ArgumentDefaultsHelpFormatter)
parser.add_argument(
"-b", "--bamfile",
required=True,
help="Sorted and indexed bam file containing the mapped reads."
"A given read is expected to be aligned at only one location.")
parser.add_argument(
"-g", "--gtf",
required=True,
help="A sorted, bgzip-compressed gtf file."
"A corresponding .tbi tabix index should exist.")
parser.add_argument(
"-a", "--annotation_getter",
choices=["tabix", "pybedtools"],
default="tabix",
help="Method to use to access annotations from the gtf file.")
parser.add_argument(
"-l", "--logfile",
help="File in which to write logs.")
args = parser.parse_args()
if not args.logfile:
logfilename = "%s.log" % args.annotation_getter
else:
logfilename = args.logfile
logging.basicConfig(
filename=logfilename,
level=logging.DEBUG)
INFO = logging.info
DEBUG = logging.debug
WARNING = logging.warning
process_annotations = make_annotation_processor(args.annotation_getter)
with annotation_context(args.gtf, args.annotation_getter) as get_annotations:
def generate_annotations(bamfile):
"""Generates annotations for the alignments in *bamfile*."""
last_t = perf_counter()
for i, ali in enumerate(bamfile.fetch(), start=1):
if not i % 1000:
now = perf_counter()
INFO("%d alignments processed (%.0f alignments / s)" % (
i,
1000.0 / (now - last_t)))
#if not i % 50000:
# gc.collect()
last_t = perf_counter()
yield process_annotations(get_annotations(ali), ali)
with pysam.AlignmentFile(args.bamfile, "rb") as bamfile:
annot_stats = Counter(generate_annotations(bamfile))
print(*reversed(annot_stats.most_common()), sep="\n")
return 0
(Я использовал контекстный менеджер и другие функции более высокого порядка (make_annotation_processor и выполняет функции, которые он вызывает), чтобы упростить использование различных подходов извлечения аннотаций в одном и том же сценарии.)