атрибуты в функции autoplot() ничего не делают, R
У меня есть набор данных со временем, статусом (1=смерть, 0=цензура), обработка =1,2.
Я создаю свой объект выживания km_2, я хочу построить график Каплана-Мейера, используя autoplot(). Я не знаю, в чем моя ошибка, но установка атрибутов (например, legendLabs) не вносит никаких изменений в основной график КМ.
km_2 <- survfit(Surv(time, status)~treatment, data=prostate)
library(ggplot2)
library(ggfortify)
autoplot(km_2,
alpha=0.7, #transparency of CIs
shape= 10, #shape used to incdicaed censored obs
xlab= 'month', ylab='% survived',
title = 'KM- plot to compare tr1, tr 2',
legendLabs= c('tr1','tr2'),
pval=T,
plotTable= T
)
1 ответ
Здесь следует отметить несколько вещей. Во-первых,
autoplotявляется общей функцией, поэтому используемый метод и аргументы, которые он принимает, зависят от типа объекта, который вы ему передаете. В этом случае вы передаете объект и сможете увидеть правильные параметры для использования, если наберете
?autoplot.survfitв консоль.
Отсюда вы увидите, что нет
legendLabsили же
plotTableвариант, и что альфа для доверительных интервалов контролируется с помощью
conf.int.alpha = . Точно так же форма цензурирования управляется с помощью
censor.shape.
Другая проблема заключается в том, что, по-видимому, нет способа изменить метки факторов в легенде, но в этом случае достаточно просто изменить их в данных при создании таблицы.
survfitобъект.
Наконец, хорошей идеей будет создать воспроизводимый пример, если вам нужны быстрые и полезные ответы. Потребовалось некоторое время, чтобы воссоздать разумную структуру данных для проверки и демонстрации этого ответа.
library(survival)
library(ggplot2)
library(ggfortify)
km_2 <- survfit(Surv(time, status) ~ treatment,
data = within(prostate, treatment <- c("tr1", "tr2")[treatment]))
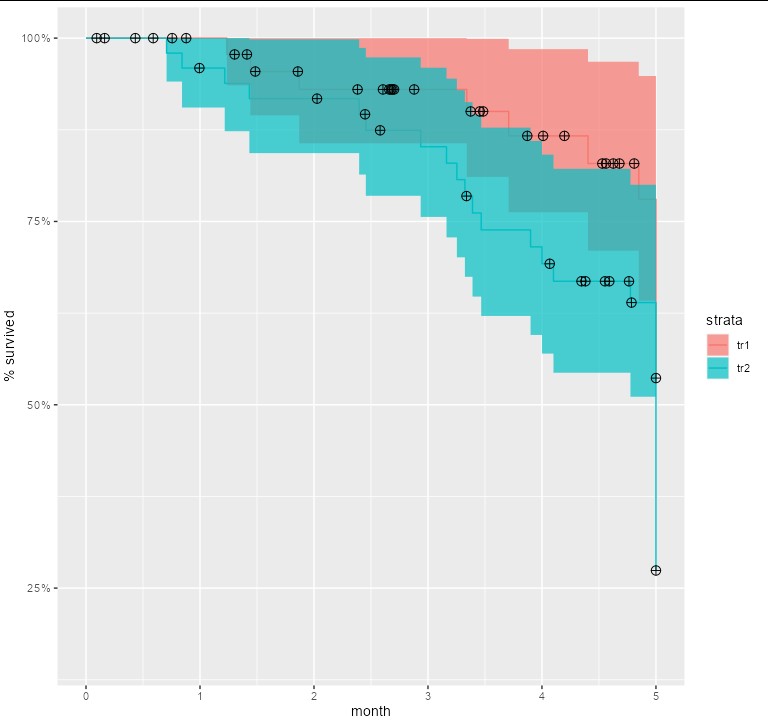
autoplot(km_2,
conf.int.alpha = 0.7,
censor.shape = 10,
xlab = 'month',
ylab = '% survived',
title = 'KM- plot to compare tr1, tr2'
)
Кроме того, вы можете получить результат ближе к вашим ожиданиям, используя
survminer:
library(survminer)
ggsurvplot(km_2, conf.int = TRUE, risk.table = TRUE)
Воспроизводимые данные
set.seed(1)
prostate <- data.frame(time = pmin(runif(100) * rep(c(7, 10), each = 50), 5),
treatment = rep(1:2, each = 50),
status = c(rbinom(50, 1, 0.3), rbinom(50, 1, 0.5)))
prostate$status[prostate$time > 5] <- 0