Неправильные буквы ANOVA+tukey с пакетом multcompView и функцией multcompLetters4
Я пытаюсь визуализировать свои статистические показатели ANOVA и апостериорного Тьюки в виде гистограммы. До сих пор это работало, но мои буквы в неправильном порядке, полоса «mit Downsampling» (2) должна отличаться от других, а не первой «ohne Sampling».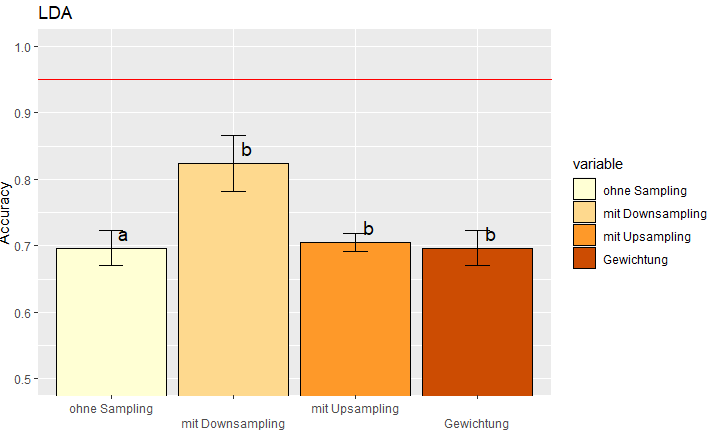
Код, который я использую:
library(ggplot2)
library(multcompView)
library(reshape2)
a.lda <- read.table(header = TRUE, text = " rowname ohne_Sampling mit_Downsampling mit_Upsampling Gewichtung
1 Fold1 0.6732673 0.8390805 0.7192982 0.6732673
2 Fold2 0.7227723 0.8181818 0.7105263 0.7227723
3 Fold3 0.7100000 0.7586207 0.6842105 0.7100000
4 Fold4 0.6633663 0.8295455 0.7105263 0.6633663
5 Fold5 0.7128713 0.8750000 0.7017544 0.7128713")
#Transformation of the dataframe to get a format ggplot2 is able to use
a.lda <- melt(a.lda, id.vars="rowname")
#data_summary Function
data_summary <- function(data, varname, groupnames){
require(plyr)
summary_func <- function(x, col){
c(mean = mean(x[[col]], na.rm=TRUE),
sd = sd(x[[col]], na.rm=TRUE),
minimum = min(x[[col]], na.rm=TRUE))
}
data_sum<-ddply(data, groupnames, .fun=summary_func,
varname)
data_sum <- rename(data_sum, c("mean" = varname))
return(data_sum)
}
a.sd.lda <- data_summary(a.lda, varname = "value", groupnames = "variable")
#ANOVA+Tuckey
a.anova <- aov(data=a.lda, value ~ variable)
tukey <- TukeyHSD(a.anova)
cld <- as.data.frame.list((multcompLetters4(a.anova,tukey))$variable)
#The wrong letters do already appear here
a.sd.lda$cld <- cld$Letters
Таким образом, проверив
a.sd.ldaтаблице уже можно увидеть неправильные буквы как a,b,b,b вместо a,b,a,a. Также, проверяя результаты tukey, НЕТ существенной разницы между ohne Sampling, mit Upsampling и Gewichtung. Так что я думаю,
multcompLetters4()функция вызывает нарушение порядка.
Буду очень благодарен за любые предложения!!!
В поисках ответа я нашел эту запись в стеке (неправильный порядок букв Tukey в пакете R multcompView ), но ни один из ответов не решил мою проблему.
Чтобы подвести итоги, это код для визуализации, хотя ошибка в моем коде должна быть выше
#Visualization
ldaplot <- ggplot(a.sd.lda, aes(variable,value,fill=variable))+
labs(title="LDA")+
scale_x_discrete(guide = guide_axis(n.dodge=2))+
coord_cartesian(ylim=c(y_min,1))+
geom_bar(stat="identity", color="black",
position=position_dodge()) +
scale_fill_brewer(palette="YlOrBr")+
geom_text(data = a.sd.lda, aes(x = variable, y = value, label = cld), size = 5, vjust=-.5, hjust=-.7)+
geom_errorbar(aes(ymin=value-sd, ymax=value+sd), width=.2,
position=position_dodge(.9))+
labs(x="", y="Accuracy")+
geom_abline(aes(intercept=Akzeptanzwert,slope=0), color="red")
Исходный код функции multcompLetter4() и связанных с ней доступен здесь: https://rdrr.io/cran/multcompView/f/
2 ответа
Хм, я вижу, вы уже решили свою проблему, но все равно вот мое решение:
a.lda <- tibble::tribble(
~Fold, ~Var, ~Val,
"Fold1", "ohne_Sampling", 0.6732673,
"Fold2", "ohne_Sampling", 0.7227723,
"Fold3", "ohne_Sampling", 0.71,
"Fold4", "ohne_Sampling", 0.6633663,
"Fold5", "ohne_Sampling", 0.7128713,
"Fold1", "mit_Downsampling", 0.8390805,
"Fold2", "mit_Downsampling", 0.8181818,
"Fold3", "mit_Downsampling", 0.7586207,
"Fold4", "mit_Downsampling", 0.8295455,
"Fold5", "mit_Downsampling", 0.875,
"Fold1", "mit_Upsampling", 0.7192982,
"Fold2", "mit_Upsampling", 0.7105263,
"Fold3", "mit_Upsampling", 0.6842105,
"Fold4", "mit_Upsampling", 0.7105263,
"Fold5", "mit_Upsampling", 0.7017544,
"Fold1", "Gewichtung", 0.6732673,
"Fold2", "Gewichtung", 0.7227723,
"Fold3", "Gewichtung", 0.71,
"Fold4", "Gewichtung", 0.6633663,
"Fold5", "Gewichtung", 0.7128713
)
library(tidyverse)
library(dlookr)
library(emmeans)
library(multcomp)
library(multcompView)
library(scales)
# data summary ------------------------------------------------------------
a.sd.lda <- a.lda %>%
group_by(Var) %>%
dlookr::describe(Val) %>%
ungroup()
a.sd.lda
#> # A tibble: 4 x 27
#> variable Var n na mean sd se_mean IQR skewness kurtosis
#> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Val Gewichtung 5 0 0.696 0.0264 0.0118 0.0396 -0.536 -2.63
#> 2 Val mit_Downs~ 5 0 0.824 0.0423 0.0189 0.0209 -0.798 1.79
#> 3 Val mit_Upsam~ 5 0 0.705 0.0133 0.00595 0.00877 -1.12 1.46
#> 4 Val ohne_Samp~ 5 0 0.696 0.0264 0.0118 0.0396 -0.536 -2.63
#> # ... with 17 more variables: p00 <dbl>, p01 <dbl>, p05 <dbl>, p10 <dbl>,
#> # p20 <dbl>, p25 <dbl>, p30 <dbl>, p40 <dbl>, p50 <dbl>, p60 <dbl>,
#> # p70 <dbl>, p75 <dbl>, p80 <dbl>, p90 <dbl>, p95 <dbl>, p99 <dbl>,
#> # p100 <dbl>
# compact letter display --------------------------------------------------
emmcld <- lm(Val ~ Var, data = a.lda) %>%
emmeans(~ Var) %>%
cld(Letters = letters, adjust = "Tukey")
Я бы сказал, что это весь код, необходимый для получения описательной статистики и компактного отображения букв из теста Тьюки.
Обратите внимание, что для графиков я использую только необработанные данные.
a.ldaи вывод результата
emmcld:
воспроизведение вашего сюжета
ggplot() +
# y-axis
scale_y_continuous(
name = "Accuracy",
limits = c(NA, 1),
breaks = pretty_breaks(),
expand = expansion(mult = c(0, 0))
) +
# x-axis
scale_x_discrete(name = NULL) +
# general layout
theme_classic() +
# colors
scale_fill_brewer(palette = "YlOrBr") +
# horizontal line
geom_hline(yintercept = 0.9, color = "red") +
# bars
geom_bar(
data = emmcld,
aes(y = emmean, x = Var, fill = Var),
color = "black",
stat = "identity"
) +
# errorbars
geom_errorbar(data = emmcld,
aes(
ymin = emmean - SE,
ymax = emmean + SE,
x = Var
),
width = 0.1) +
# letters
geom_text(
data = emmcld,
aes(
y = emmean + SE,
x = Var,
label = str_trim(.group)
),
hjust = 0.5,
vjust = -0.5
) +
# caption
labs(
caption = str_wrap(
"Bars with errorbars represent (estimated marginal) means ± standard error. Means not sharing any letter are significantly different by the Tukey-test at the 5% level of significance.",
width = 90
)
)

предложение альтернативного сюжета
..потому что некоторым людям не нравятся "динамитные заговоры", красиво описанные в этом блоге
# optional: sort factor levels of groups column according to highest mean
# ...in means table
emmcld <- emmcld %>%
mutate(Var = fct_reorder(Var, emmean))
# ...in data table
a.lda <- a.lda %>%
mutate(Var = fct_relevel(Var, levels(emmcld$group)))
# base plot setup
ggplot() +
# y-axis
scale_y_continuous(limits = c(NA, 1),
name = "Accuracy",
breaks = pretty_breaks()) +
# x-axis
scale_x_discrete(name = NULL) +
# general layout
theme_classic() +
# colors
scale_fill_brewer(palette = "YlOrBr", guide = "none") +
scale_color_brewer(palette = "YlOrBr", guide = "none") +
# horizontal line
geom_hline(yintercept = 0.9,
color = "red",
linetype = "dashed") +
# black data points
geom_point(
data = a.lda,
aes(y = Val, x = Var, fill = Var),
shape = 21,
alpha = 0.9,
position = position_nudge(x = -0.2)
) +
# black boxplot
geom_boxplot(
data = a.lda,
aes(y = Val, x = Var, fill = Var),
width = 0.05,
outlier.shape = NA,
position = position_nudge(x = -0.1)
) +
# red mean value
geom_point(data = emmcld,
aes(y = emmean, x = Var),
size = 2,
color = "red") +
# red mean errorbar
geom_errorbar(
data = emmcld,
aes(ymin = lower.CL, ymax = upper.CL, x = Var),
width = 0.05,
color = "red"
) +
# red letters
geom_text(
data = emmcld,
aes(
y = emmean,
x = Var,
label = str_trim(.group)
),
position = position_nudge(x = 0.1),
hjust = 0,
color = "red"
) +
# caption
labs(
caption = str_wrap(
"Black dots represent raw data. Red dots and error bars represent (estimated marginal) means ± 95% confidence interval per group. Means not sharing any letter are significantly different by the Tukey-test at the 5% level of significance.",
width = 90
)
)

Создано 27 января 2022 г. пакетом reprex (v2.0.1)
Хорошо, попробовав МНОГО, я нашел способ решить эту проблему с помощью другого пакета:
multcomp
Итак, вот мой код для получения правильных результатов и графика:
library(ggplot2)
library(multcomp)
library(reshape2)
a.lda <- read.table(header = TRUE, text = " rowname ohne_Sampling mit_Downsampling mit_Upsampling Gewichtung
1 Fold1 0.6732673 0.8390805 0.7192982 0.6732673
2 Fold2 0.7227723 0.8181818 0.7105263 0.7227723
3 Fold3 0.7100000 0.7586207 0.6842105 0.7100000
4 Fold4 0.6633663 0.8295455 0.7105263 0.6633663
5 Fold5 0.7128713 0.8750000 0.7017544 0.7128713")
#Transformation of the dataframe to get a format ggplot2 is able to use
a.lda <- melt(a.lda, id.vars="rowname")
#data_summary Function
data_summary <- function(data, varname, groupnames){
require(plyr)
summary_func <- function(x, col){
c(mean = mean(x[[col]], na.rm=TRUE),
sd = sd(x[[col]], na.rm=TRUE),
minimum = min(x[[col]], na.rm=TRUE))
}
data_sum<-ddply(data, groupnames, .fun=summary_func,
varname)
data_sum <- rename(data_sum, c("mean" = varname))
return(data_sum)
}
a.sd.lda <- data_summary(a.lda, varname = "value", groupnames = "variable")
#ANOVA+Tuckey **NEW**
a.anova <- aov(data=a.lda, value ~ variable)
tukey <- glht(a.anova, linfct = mcp(variable = "Tukey"))
tuk.cld <- cld(tukey)
a.sd.lda$cld <- tuk.cld$mcletters$Letters
#Visualization
ldaplot <- ggplot(a.sd.lda, aes(variable,value,fill=variable))+
labs(title="LDA")+
scale_x_discrete(guide = guide_axis(n.dodge=2))+
coord_cartesian(ylim=c(y_min,1))+
geom_bar(stat="identity", color="black",
position=position_dodge()) +
scale_fill_brewer(palette="YlOrBr")+
geom_text(data = a.sd.lda, aes(x = variable, y = value, label = cld), size = 5, vjust=-.5, hjust=-.7)+
geom_errorbar(aes(ymin=value-sd, ymax=value+sd), width=.2,
position=position_dodge(.9))+
labs(x="", y="Accuracy")+
geom_abline(aes(intercept=Akzeptanzwert,slope=0), color="red")
идея для этого решения пришла из: https://stats.stackexchange.com/questions/31547/how-to-obtain-the-results-of-a-tukey-hsd-post-hoc-test-in-a-table -показ-группа
Надеюсь, это поможет и другим пользователям R :)