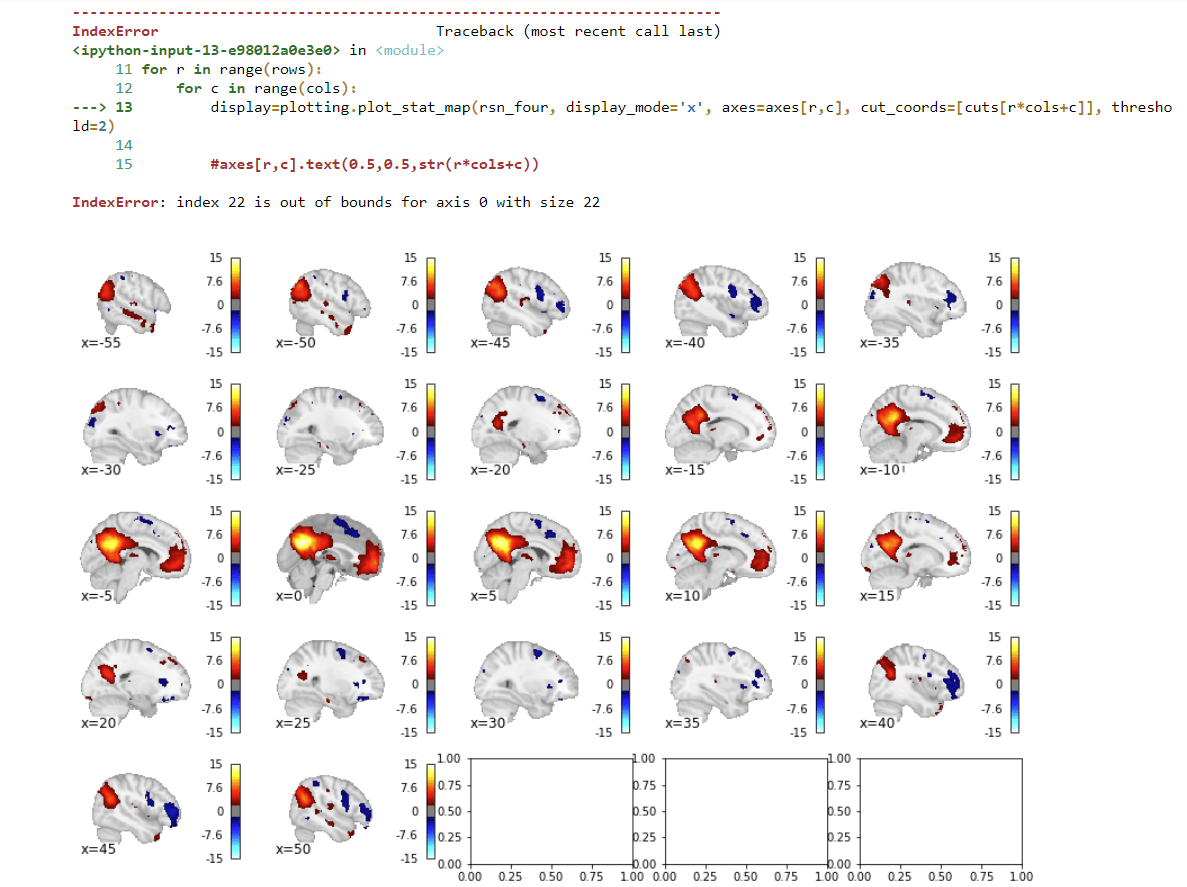
Удаление / скрытие пустых подзаголовков в matplotlib при построении гибкой сетки
Я использую nilearn, чтобы нанести ряд разрезов вдоль оси через файл трехмерного изображения мозга. Моя цель - сделать гибкую функцию, в которой я могу изменять количество строк, столбцов и диапазон координат разрезов по своему усмотрению. Причина этого в том, что я могу создать файл.png и использовать его для визуализации, например, непосредственно в документе.
Итак, в основном я использую вложенный цикл для создания сетки подзаголовков matplotlib и заполнения их изображениями мозга. Они поступают из одной строчки кода функции nilearn, так что проблема не в этом.
Мой вопрос: есть ли способ обнаружить и скрыть / удалить пустые участки? Проблема в том, что теперь пустые подзаголовки приходят с сообщением об ошибке, которое, кажется, портит файл.png, который он возвращает. Кроме того, в файле.png показаны пустые участки, чего не должно быть.
Вот мой код:
i = -55
j = 55
rows = 5
cols = 5
k = np.ceil((abs(i)+j)/(rows*cols))
cuts = np.arange(i,j,k)
rsn_img = 'rsn_img.png'
fig, axes = plt.subplots(rows, cols, figsize=(15,10))
for r in range(rows):
for c in range(cols):
display=plotting.plot_stat_map(rsn_four, display_mode='x', axes=axes[r,c], cut_coords=[cuts[r*cols+c]], threshold=2)
print(f'plotting at index [ {r} , {c}]')
display.savefig(rsn_img)
Незаполненные участки следует удалить. Заранее спасибо за помощь!
1 ответ
Я полагаю, вы знаете количество изображений
N что вы хотите построить.
Вот как я бы это написал:
i = -55
j = 55
cols = 5
rows = 5
k = np.ceil((abs(i)+j)/(rows*cols))
cuts = np.arange(i,j,k)
N = len(cuts)
fig, axs = plt.subplots(rows, cols)
for ax,cut in zip(axs.flat,cuts):
display=plotting.plot_stat_map(rsn_four, display_mode='x', axes=ax, cut_coords=cut, threshold=2)
# remove unused axes
for ax in axs.flat[N:]:
ax.remove()