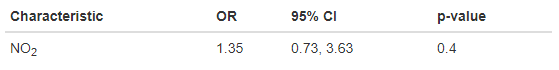
Как добавить индексы к меткам в таблицах с помощью пакета gtsummary в R?
Я хотел бы ввести нижний индекс в имени переменной для сводной таблицы регрессии, созданной с помощью tbl_regression в пакете R gtsummary. Может ли кто-нибудь дать указания, как это сделать? Код, использованный для создания таблицы, и итоговая таблица представлены ниже. Я бы хотел, чтобы метка "NO2" появилась как "NO2".
library(tidyverse)
library(gtsummary)
case <- c(0,1,0,0,0,0)
no2 <- c(17, 14, 8, 9, 9, 7)
df <- data.frame(case, no2)
mod_adj <- glm(case~no2,data=df, family="binomial")
regression_table_adj <- mod_adj %>%
tbl_regression(exponentiate = TRUE, label = list(no2~"NO2"))
regression_table_adj
Создано 2020-03-04 пакетом REPEX (v0.3.0)
2 ответа
Пакет gtsummary по умолчанию использует пакет gt для печати таблиц. Я просмотрел их документацию и не нашел способа включить индекс в тело таблицы.
Хорошей новостью является то, что gtsummary также поддерживает печать таблиц с knitr::kable() через as_kable()функция. Вы можете заключить текст между двумя тильдами, чтобы сделать его нижним индексом (например,label = list(no2 ~ "NO~2~")). Используйте приведенный ниже код в файле уценки R, и вы должны получить подстрочный индекс. Обратной стороной использования kable является то, что он не поддерживает сноски, отступы и охватывающий заголовок. Удачного кодирования!
library(gtsummary)
case <- c(0,1,0,0,0,0)
no2 <- c(17, 14, 8, 9, 9, 7)
df <- data.frame(case, no2)
mod_adj <- glm(case~no2,data=df, family="binomial")
mod_adj %>%
tbl_regression(
exponentiate = TRUE,
label = list(no2 ~ "NO~2~")
) %>%
as_kable()
Создано 2020-03-04 пакетом REPEX (v0.3.0)
Это не мое, но я нашел здесь ответ и адаптировал его. https://community.rstudio.com/t/subscripts-to-annotate-gt-table/87089
По сути, вы преобразуете его в
gtobject и взломайте метку, чтобы она печаталась как индекс.
library(tidyverse)
library(gtsummary)
library(gt)
case <- c(0,1,0,0,0,0)
no2 <- c(17, 14, 8, 9, 9, 7)
df <- data.frame(case, no2)
mod_adj <- glm(case~no2,data=df, family="binomial")
regression_table_adj <- mod_adj %>%
tbl_regression(exponentiate = TRUE, label = list(no2 = "NO@2~")) %>%
as_gt() %>%
text_transform(
locations = cells_body(),
fn = function(x) {
str_replace_all(x,
pattern = "@",
replacement = "<sub>") %>%
str_replace_all("~",
"</sub>") }
)
Да, символы можно легко заменить. Я думаю, что было бы лучше использовать что-то другое, чем
'~'так как это может легко означать что-то еще.