Добавление символов и информации в филогенетическое дерево
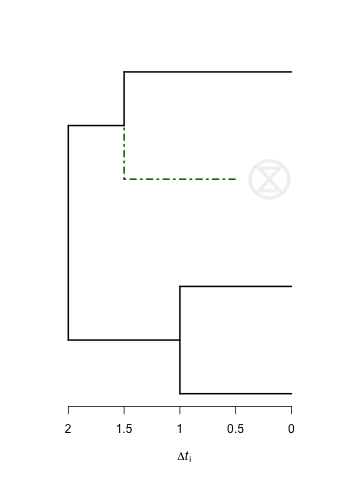
Я рисую филогенетическое дерево, и я хотел бы добавить что-то вроде "мертвого символа" (например, череп) на кончиках вымерших видов.
Я также хотел бы добавить панель осей X с латексными символами во времена ветвления (например, $\Delta t_i$ или числа), отмеченные точками.
То, что у меня есть, это дерево. Я хотел бы добавить мертвый символ сразу после зеленой пунктирной линии в этом случае.
library(ape)
rec1 = '((B:1,A:1):1,(F:1,C:1.5):0.5);'
rec1 = read.tree(text = rec1)
plot(rec1,show.tip.label = F,edge.color = c("black","black","black","black","darkgreen","black"),edge.width = 2,edge.lty = c(rep(1,4),4,1))
2 ответа
Я вижу два варианта, как отобразить "потухший" символ на кончике дерева.
- Используйте символ Unicode с соответствующим шрифтом, который может отображать его в соответствии с этим блогом.
- Добавьте растровое изображение на древовидный график.
Следующий код будет отображать символ исчезновения рядом с зеленым краем в вашем дереве. Он опирается на информацию, найденную здесь.
library(jpeg)
logo <- readJPEG("Downloads/Symbol1.jpg")
logo2 <- as.raster(logo)
r <- nrow(logo2)/ncol(logo2) # aspect ratio
s <- 0.4 # symbol size
# display plot to obtain its size
plot(rec1, edge.color = c("black","black","black","black","darkgreen","black"),
edge.width = 2, edge.lty = c(rep(1,4),4,1))
lims <- par("usr") # plot area size
file_r <- (lims[2]-lims[1]) / (lims[4]-lims[3]) # aspect ratio for the file
file_s <- 480 # file size
# save tree with added symbol
png("tree_logo.png", height=file_s, width=file_s*file_r)
plot(rec1, show.tip.label = F,
edge.color = c("black","black","black","black","darkgreen","black"),
edge.width = 2, edge.lty = c(rep(1,4),4,1))
rasterImage(logo2, 1.6, 2.8, 1.6+s/r, 2.8+s)
# add axis
axisPhylo()
mtext(expression(Delta*italic("t")["i"]), side = 1, line = 3)
dev.off()
Одна возможность - использовать ggtree. Как в: https://www.bioconductor.org/packages/devel/bioc/vignettes/ggtree/inst/doc/advanceTreeAnnotation.html
#source("https://bioconductor.org/biocLite.R")
#biocLite("BiocUpgrade") # you may need this
#biocLite("ggtree")
library(ggtree)
imgfile <- tempfile(, fileext=".png")
download.file("https://avatars1.githubusercontent.com/u/626539?v=3&u=e731426406dd3f45a73d96dd604bc45ae2e7c36f&s=140", destfile=imgfile, mode='wb')
img <- list(imgfile, imgfile)
names(img) <- c("1", "2")
p<-ggtree(rtree(20))
inset(p, img)
С филопией
#https://github.com/sckott/rphylopic
# devtools::install_github("sckott/rphylopic")
library('rphylopic')
string<-name_search(text = "Homo sapiens")
selectstr<-string[2,]
string2<-name_images(uuid = selectstr)$same[[1]]$uid
tree<-rtree(10)
ggtree(tree) %>% phylopic(string2, color="steelblue", alpha = .8, node=10, width=0.5)