Есть ли способ нарисовать график UMAP или t-SNE для таблицы данных?
У меня есть огромный файл (ниже небольшой набор данных), как показано ниже, я хотел бы нарисовать PCA, я мог бы нарисовать PCA с помощью функции PCA, но это выглядит немного беспорядочно, потому что у меня 200 столбцов, поэтому я думаю, что, возможно, t-SNE или UMAP работают лучше, но я не мог рисовать с их помощью.
Я хотел бы показать связь и кластеризацию между столбцами (имя столбца) на графике. Фактически, я собрал данные A, B и... из разных исследований, и мне нравится проверять, есть ли между ними какой-либо групповой эффект или нет.
Буду признателен, если кто-нибудь может мне помочь!
DF:
A B C D
1:540450-541070 0.12495878 0.71580434 0.65399319 1.04879290
1:546500-548198 0.41064192 0.26136554 0.11939805 0.28721360
1:566726-567392 0.00000000 0.06663644 0.45661687 0.24408844
1:569158-570283 0.34433086 0.27614141 0.54063437 0.21675053
1:603298-605500 0.07036734 0.42324126 0.23017472 0.29530045
1:667800-669700 0.20388011 0.11678913 0.00000000 0.12833913
1:713575-713660 7.29171225 12.53078648 2.38515165 3.82500941
1:724497-727160 0.40730086 0.26664585 0.45678834 0.12209005
1:729399-731900 0.74345727 0.49685579 0.72956458 0.32499580
1 ответ
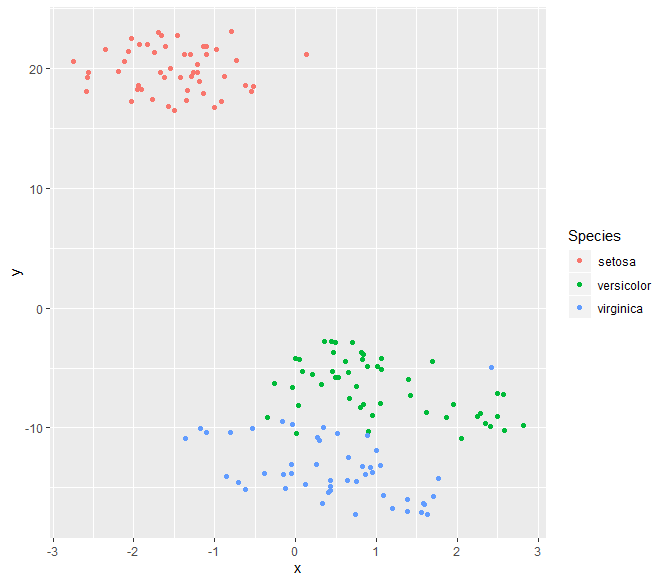
Вот несколько примеров использования набора данных радужной оболочки глаза, так как данные вашего примера слишком малы для уменьшения размерности.
Для tSNE:
library(ggplot2)
library(Rtsne)
dat <- iris
tsne <- Rtsne(dat[!duplicated(dat), -5])
df <- data.frame(x = tsne$Y[,1],
y = tsne$Y[,2],
Species = dat[!duplicated(dat), 5])
ggplot(df, aes(x, y, colour = Species)) +
geom_point()
Для UMAP:
library(umap)
umap <- umap(dat[!duplicated(dat), -5])
df <- data.frame(x = umap$layout[,1],
y = umap$layout[,2],
Species = dat[!duplicated(dat), 5])
ggplot(df, aes(x, y, colour = Species)) +
geom_point()
РЕДАКТИРОВАТЬ: Предположим, у нас есть данные, в которых каждая тема представляет собой столбец:
dat <- t(mtcars)
Единственными дополнительными шагами было бы транспонировать данные перед их загрузкой в tSNE/UMAP, а затем скопировать имена столбцов в графических данных:
tsne <- Rtsne(t(dat), perplexity = 5) # got warning perplexity is too large
df <- data.frame(x = tsne$Y[,1],
y = tsne$Y[,2],
car = colnames(dat))
ggplot(df, aes(x, y, colour = car)) +
geom_point()