R цикл для применения уравнения к каждой уникальной категории
Я ищу помощь в написании цикла R для расчета информационного содержания Шеннона (SIC) для каждого уникального гаплотипа. Данные включают в себя гаплотипы в столбце 1 и частоту гаплотипов в столбце 2. Как видно из данных примера только с 4 уникальными гаплотипами, у каждого гаплотипа есть разные числа с частотой, соответствующей каждому. Частота для всех гаплотипов H* суммируется до 1. Уравнение для SIC
Σi (πhi*log(1/(πhi)))
где πhi - частота высокого гаплотипа
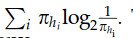
Haplotype Frequency
H1 0.8278
H1 0.02248
H1 0.1494
H2 0.8238
H2 0.02248
H2 0.1497
H3 0.1497
H3 0.02248
H3 0.8244
H4 0.628
H4 0.02248
H4 0.1483
H4 0.1637
H4 0.01081
H4 0.01798
В этом примере SIC для H1 будет
(π*0,8278*log(1/(π*0,8278)))) + (π*0,02248*log(1/(π*0,02248)))) + (π*0,1494*log(1/(π*0,1494))))
и окончательный результат должен дать 4 значения SIC, одно из которых соответствует каждому уникальному гаплотипу.
Я верю, используя lapply() это правильный способ идти вперед, но мои навыки R очень элементарны, чтобы знать, что делать дальше. Спасибо за любую помощь.
2 ответа
Ты хочешь aggregate:
result <- aggregate(df, by = list(df$Haplotype), function (h) sic(h[2]))
library(plyr)
ddply(df1,.(Haplotype),summarize,mysum=sum(pi*Frequency*log(1/(pi*Frequency))))
Haplotype mysum
1 H1 -1.9433259
2 H2 -1.9190102
3 H3 -1.9226882
4 H4 -0.1784109
library(data.table)
dt1<-data.table(df1)
dt1[,list(sum=sum(pi*Frequency*log(1/(pi*Frequency)))),by=Haplotype]
Haplotype sum
1: H1 -1.9433259
2: H2 -1.9190102
3: H3 -1.9226882
4: H4 -0.1784109