Проблемы с отображением линии регрессии LOESS и доверительного интервала
У меня возникли некоторые проблемы, пытаясь сопоставить регрессию LOESS с набором данных. Мне удалось правильно создать линию, но я не могу заставить ее правильно построить график.
Я проверил данные, как это.
animals.lo <- loess(X15p5 ~ Period, animals, weights = n.15p5)
animals.lo
summary(animals.lo)
plot(X15p5~ Period, animals)
lines(animals$X15p5, animals.lo, col="red")
В этот момент я получил ошибку
"Ошибка в xy.coords(x, y): длины" x "и" y "различаются"
Я искал вокруг и прочитал, что эта проблема может быть из-за пунктов, которые должны быть заказаны, поэтому я продолжил.
a <- order(animals$Period)
lines(animals$X15p5[a], animals.lo$Period[a], col="red", lwd=3)
В этот момент ошибок не было, но линия LOESS все еще не появлялась на графике. Точки были отображены правильно, но не линия.
Это похоже на набор данных, который я использую...
structure(list(Site = c("Cat", "Dog", "Bear", "Chicken", "Cow",
"Bird", "Tiger", "Lion", "Leopard", "Wolf", "Puppy", "Kitten",
"Emu", "Ostrich", "Elephant", "Sheep", "Goat", "Fish", "Iguana",
"Monkey", "Gorilla", "Baboon", "Lemming", "Mouse", "Rat", "Hamster",
"Eagle", "Parrot", "Crow", "Dove", "Falcon", "Hawk", "Sparrow",
"Kite", "Chimpanzee", "Giraffe", "Bear", "Donkey", "Mule", "Horse",
"Zebra", "Ox", "Snake", "Cobra", "Iguana", "Lizard", "Fly", "Mosquito",
"Llama", "Butterfly", "Moth", "Worm", "Centipede", "Unicorn",
"Pegasus", "Griffin", "Ogre", "Monster", "Demon", "Witch", "Vampire",
"Mummy", "Ghoul", "Zombie"), Region = c(6L, 4L, 4L, 5L, 7L, 6L,
2L, 4L, 6L, 7L, 7L, 4L, 6L, 4L, 4L, 4L, 4L, 3L, 4L, 4L, 4L, 4L,
4L, 4L, 4L, 4L, 3L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 8L, 4L, 6L, 6L,
4L, 2L, 7L, 4L, 2L, 2L, 7L, 3L, 4L, 7L, 4L, 4L, 4L, 7L, 7L, 4L,
4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 2L, 8L), Period = c(-2715, -3500,
-3500, -4933.333333, -2715, -2715, -2715, -3500, -2715, -4350,
-3500, -3500, -2950, -4350, -3650, -3500, -3500, -2715, -3650,
-4350, -3500, -3500, -3400, -4350, -3500, -3500, -4350, -3900,
-3808.333333, -4233.333333, -3500, -3900, -3958.333333, -3900,
-3500, -3500, -3500, -2715, -3650, -2715, -2715, -2715, -2715,
-3500, -2715, -2715, -3500, -4350, -3650, -3650, -4350, -5400,
-3500, -3958.333333, -3400, -3400, -4350, -3600, -4350, -3650,
-3500, -2715, -5400, -3500), Value = c(0.132625995, 0.163120567,
0.228840125, 0.154931973, 0.110047847, 0.054347826, 0.188679245,
0.245014245, 0.128378378, 0.021428571, 0.226277372, 0.176923077,
0.104938272, 0.17659805, 0.143798024, 0.086956522, 0.0625, 0.160714286,
0, 0.235588972, 0, 0, 0.208333333, 0.202247191, 0.364705882,
0.174757282, 0, 0.4, 0.1, 0.184027778, 0.232876712, 0.160493827,
0.74702381, 0.126984127, 0.080645161, 0.06557377, 0, 0.057692308,
0.285714286, 0.489361702, 0.108695652, 0.377777778, 0, 0.522727273,
0.024390244, 0.097560976, 0.275, 0, 0.0625, 0.255319149, 0.135135135,
0.216216216, 0.222222222, 0.296296296, 0.222222222, 0.146341463,
0.09375, 0.125, 0.041666667, 0.078947368, 0.2, 0.137931034, 0.571428571,
0.142857143), Sample_size = c(188.5, 105.75, 79.75, 70, 52.25,
46, 39.75, 39, 37, 35, 34.25, 32.5, 32.4, 30.76666667, 30.36666667,
28.75, 28, 28, 28, 26.6, 25, 25, 24, 22.25, 21.25, 20.6, 20,
20, 20, 19.2, 18.25, 18, 18, 16.8, 15.5, 15.25, 15, 13, 12.6,
11.75, 11.5, 11.25, 11, 11, 10.25, 10.25, 10, 10, 9.6, 9.4, 9.25,
9.25, 9, 9, 9, 8.2, 8, 8, 8, 7.6, 7.5, 7.25, 7, 7), Sample_sub = c(25,
17.25, 18.25, 10.8452381, 5.75, 2.5, 7.5, 9.555555556, 4.75,
0.75, 7.75, 5.75, 3.4, 5.433333333, 4.366666667, 2.5, 1.75, 4.5,
0, 6.266666667, 0, 0, 5, 4.5, 7.75, 3.6, 0, 8, 2, 3.533333333,
4.25, 2.888888889, 13.44642857, 2.133333333, 1.25, 1, 0, 0.75,
3.6, 5.75, 1.25, 4.25, 0, 5.75, 0.25, 1, 2.75, 0, 0.6, 2.4, 1.25,
2, 2, 2.666666667, 2, 1.2, 0.75, 1, 0.333333333, 0.6, 1.5, 1,
4, 1)), .Names = c("Site", "Region", "Period", "Value", "Sample_size",
"Sample_sub"), class = "data.frame", row.names = c(NA, -64L))
Я работал над этим некоторое время и пытался читать столько, сколько я могу, но я не смог сделать никакого дополнительного прогресса. Любой совет или руководство будет принята с благодарностью.
Продолжение добавления доверительного интервала к графику
Я пытался добавить доверительные интервалы, следуя другому примеру, найденному на сайте на этой странице. Как получить доверительные интервалы для подбора НИЗКОГО, используя R?,
Пример, приведенный на этой странице:
plot(cars)
plx<-predict(loess(cars$dist ~ cars$speed), se=T)
lines(cars$speed,plx$fit)
lines(cars$speed,plx$fit - qt(0.975,plx$df)*plx$se, lty=2)
lines(cars$speed,plx$fit + qt(0.975,plx$df)*plx$se, lty=2)
Я адаптировал это так:
plot(X15p5 ~ Period, animals)
animals.lo2<-predict(loess(animals$X15p5 ~ animals$Period), se=T)
a <- order(animals$Period)
lines(animals$Period[a],animals.lo2$fit, col="red", lwd=3)
lines(animals$Period[a],animals.lo2$fit - qt(0.975,animals.lo2$df)*animals.lo2$se, lty=2)
lines(animals$Period[a],animals.lo2$fit + qt(0.975,animals.lo2$df)*animals.lo2$se, lty=2)
Хотя это и обеспечивает доверительные интервалы, все линии регрессии неверны. Я не уверен, если это проблема с predict функция или другая проблема. Еще раз спасибо!
1 ответ
правильный код
Я искал вокруг и прочитал, что эта проблема может быть из-за пунктов, которые должны быть заказаны, поэтому я продолжил.
Нет нет. Проблема заказа не связана с ошибкой, которую вы видите. Чтобы преодолеть ошибку, нужно заменить
lines(animals$X15p5, animals.lo, col="red")
с
lines(animals$Period, animals.lo$fitted, col="red")
Вот причины:
loessвозвращает список объектов, а не один вектор. Увидетьstr(animals.lo)или жеnames(animals.lo),- почему вы используете
animals$X15p5как ось х? Вы подходите своей модели:X15p5 ~ Period, поэтому ось X должна бытьPeriod,
о переупорядочении
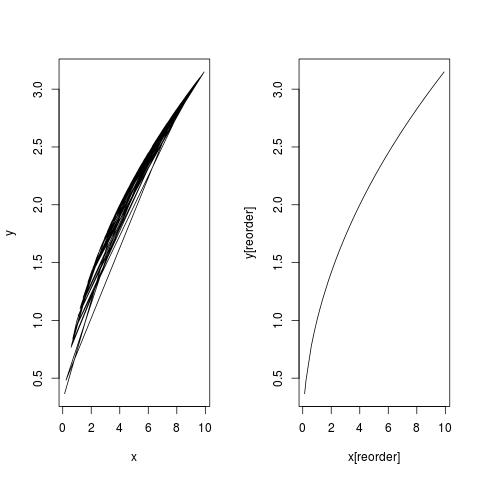
Вам нужно сделать упорядочивание, потому что по умолчанию R выстраивает точки в порядке. Возьмите это как пример:
set.seed(0); x <- runif(100, 0, 10) ## x is not in order
set.seed(1); y <- sqrt(x) ## plot curve y = sqrt(x)
par(mfrow = c(1,2))
plot(x, y, type = "l") ## this is a mess!!
reorder <- order(x)
plot(x[reorder], y[reorder], type = "l") ## this is nice
Точно так же:
a <- order(animals$Period)
lines(animals$Period[a], animals.lo$fitted[a], col="red", lwd=3)
наблюдение за доверительным интервалом
Попробуй это:
plot(X15p5 ~ Period, animals)
animals.lo <- loess(X15p5 ~ Period, animals)
pred <- predict(animals.lo, se = TRUE)
a <- order(animals$Period)
lines(animals$Period[a], pred$fit[a], col="red", lwd=3)
lines(animals$Period[a], pred$fit[a] - qt(0.975, pred$df)*pred$se[a],lty=2)
lines(animals$Period[a], pred$fit[a] - qt(0.975, pred$df)*pred$se[a],lty=2)
Вы забыли о повторном заказе. Вам нужно изменить порядок как подгоненных значений, так и стандартных ошибок.
Теперь dist ~ speed модель для cars данные не нуждаются в переупорядочении. Так как:
is.unsorted(cars$speed) ## FALSE
Да, данные там уже отсортированы.
Обратите внимание, что я сделал два других изменения в вашем коде:
- Я рассталась
loessпозвонить иpredictвызов; Возможно, вам не нужно этого делать, но обычно полезно разделять подгонку модели и прогнозирование модели и хранить копии обоих объектов. - я изменился
loess(animals$X15p5 ~ animals$Period)вloess(X15p5 ~ Period, animals), Это плохая привычка$войдите в указав формулу модели. У меня есть другой ответ на /questions/19504166/predskazanieglm-s-tremya-novyimi-kategoriyami-v-testovyih-dannyih-roshibka/19504183#19504183 показывающий недостаток такого стиля. Вы можете прочитать в разделе "Обновление" там. Я использовалglmв качестве примера, но дляlm,glm,loessвсе одинаково.