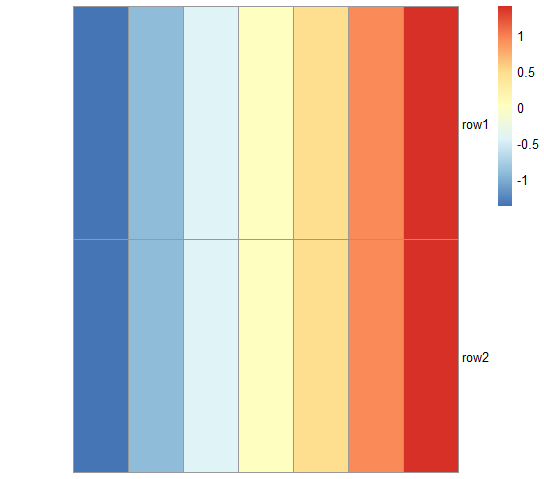Как привязать цветовую шкалу к минимуму / максимуму для каждой строки, используя pheatmap в R
В настоящее время я использую pheatmap в R, чтобы создать тепловую карту выражений генов из данных одной клетки.
Проблема, с которой я сейчас сталкиваюсь, заключается в том, что я не могу воспроизвести относительную цветовую шкалу, используемую в других генераторах тепловых карт, таких как Morpheus: https://software.broadinstitute.org/morpheus/
Для уточнения, в Morpheus цветовая шкала может быть привязана к минимальному и максимальному значениям выражения для каждой строки. Например, если предположить, что цветовая гамма является чем-то простым c('dark_blue', 'blue', 'bright_blue', 'white', 'bright_red', 'red', 'dark_red') и я предоставляю матрицу, которая выглядит как t(data.frame(row1=c(0, 1, 2, 3, 4, 5, 6), row2=c(10, 11, 12, 13, 14, 15, 16))), Морфеус закрасит 0 темно-синим и 6 темно-красными в первом ряду, в то время как 10 будет темно-синим и 16 будет темно-красным во втором ряду. Цветная полоса представляет относительные минимальные и максимальные значения для каждой строки.
Обратите внимание, что это не то же самое, что простое масштабирование данных, что я и делал в pheatmap. Проблема в том, что если у меня есть что-то вроде t(data.frame(row1=c(1, 2, 3, 4, 5, 6), row2=c(10, 0, 0, 0, 0, 0)))тогда даже масштабирование данных не позволит 6 (~1,34 после масштабирования) быть окрашенным в темно-красный цвет, поскольку цветовая шкала привязана к минимуму (~-0,41 после масштабирования) и максимуму (~2,04 после масштабирования) для всей тепловой карты,
Я не сомневаюсь, что есть простой способ достичь этого, но я не смог понять это, просматривая основную документацию. Я также понимаю, что отображение тепловой карты таким образом не позволит проводить сравнение между генами, но это не особенно важно для моих целей.
1 ответ
Вы можете масштабировать данные по строкам следующим образом:
mat <- t(data.frame(row1=c(0, 1, 2, 3, 4, 5, 6), row2=c(10, 11, 12, 13, 14, 15, 16)))
mat2 <- t(apply(mat, 1, scale))
затем pheatmap имеет аргумент cluster_cols который вы можете установить на FALSE, Сложив все это вместе, мы получим 0 и 10 темно-синий в окончательной тепловой карте.
pheatmap(mat2, cluster_cols = F)