Получите больше метаданных из исследования Нейротрансмиттер с использованием Allen SDK
Я загружаю все изображения из исследования нейротрансмиттера Аллена Мозга, используя этот скрипт:
from allensdk.api.queries.image_download_api import ImageDownloadApi
from allensdk.config.manifest import Manifest
import pandas as pd
import os
#getting transmitter study
#product id from http://api.brain-map.org/api/v2/data/query.json?criteria=model::Product
nt_datasets = image_api.get_section_data_sets_by_product([27])
#an instance of Image Api for downloading
image_api = ImageDownloadApi()
for index, row in df_nt.iterrows():
#get section dataset id
section_dataset_id= row['id']
#each section dataset id has multiple image sections
section_images = pd.DataFrame(
image_api.section_image_query(
section_data_set_id=section_dataset_id)
)
for section_image_id in section_images['id'].tolist():
file_path = os.path.join('/path/to/save/dir/',
str(section_image_id) + '.jpg' )
Manifest.safe_make_parent_dirs(file_path)
image_api.download_section_image(section_image_id,
file_path=file_path,
downsample=downsample)
Этот скрипт загружает предположительно все доступные эксперименты ISH. Тем не менее, мне интересно, что будет лучшим способом получить больше метаданных следующим образом:
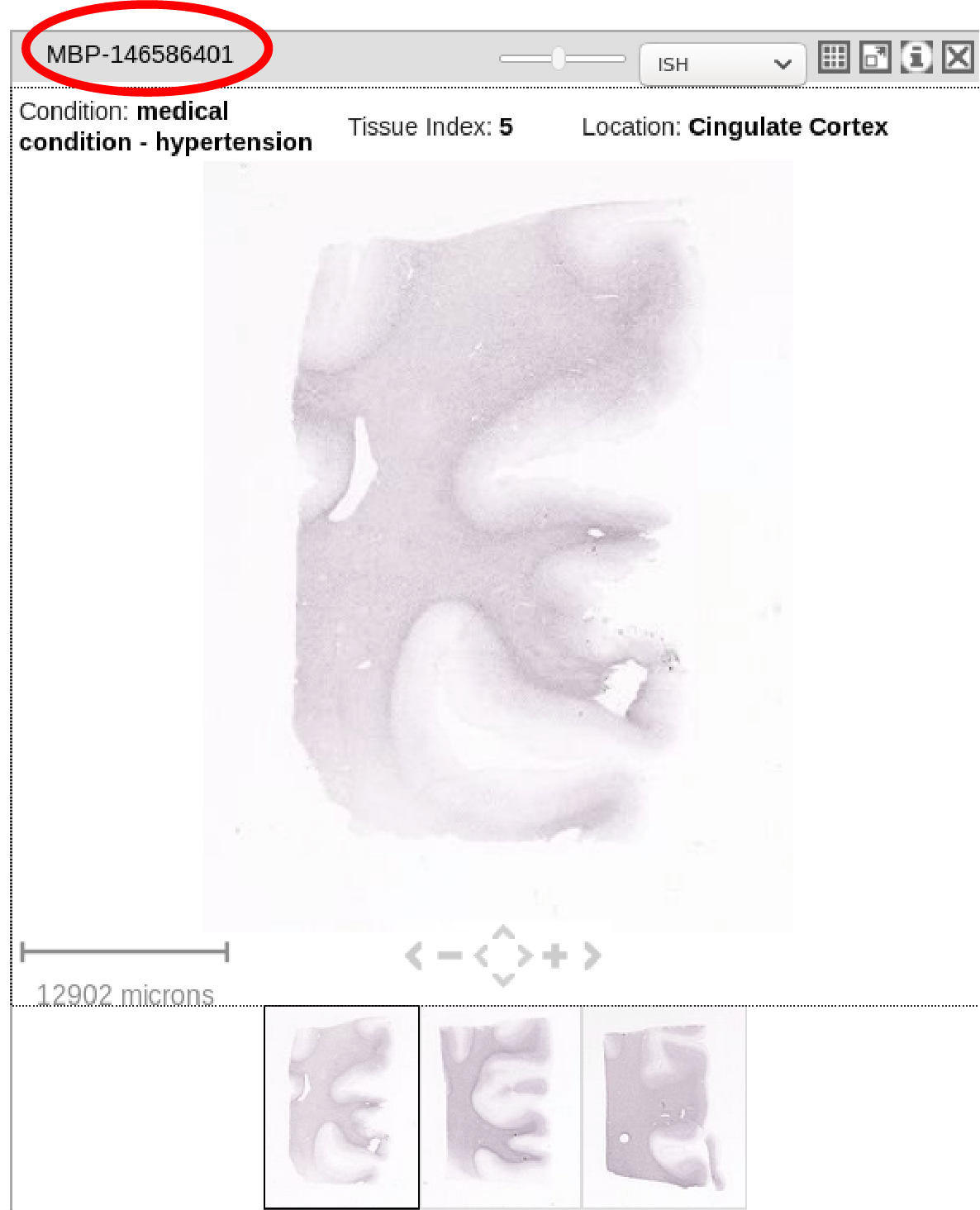
1) тип эксперимента по ISH, известный как "ген" (например, окрашен ли MBP, окрашен по Nissl или т. Д.). Показано в красном круге ниже.
2) Структура и соответствие изображению атласа (аннотации, например, чтобы увидеть, к какой части мозга принадлежит раздел). Я думаю, что это может быть приобретено с tree_search но не уверен как. Показанный в красных кругах ниже от двух разных веб-страниц на сайте Аллена.
3) Масштаб изображения, например, насколько велик один пиксель в загруженном изображении (например, 0,001x0,001 мм). Я бы потребовал это для анализа изображений в отношении МРТ, например. Показано ниже в красном круге.
Вся вышеупомянутая информация так или иначе доступна на веб-сайте, мой вопрос заключается в том, можете ли вы помочь мне сделать это программно через SDK.
РЕДАКТИРОВАТЬ:
Также было бы неплохо загружать пятна "Nissl" программно, так как они не отображаются с использованием вышеуказанной итерации цикла. Картинка показана ниже.
1 ответ
Чтобы получить доступ к этой информации, вам нужно сформулировать несколько сложный запрос API.
from allensdk.api.queries.rma_api import RmaApi
api = RmaApi()
data_set_id = 146586401
data = api.model_query('SectionDataSet',
criteria='[id$eq%d]' % data_set_id,
include='section_images,treatments,probes(gene),specimen(structure)')
print("gene symbol: %s" % data[0]['probes'][0]['gene']['acronym'])
print("treatment name: %s" % data[0]['treatments'][0]['name'])
print("specimen location: %s" % data[0]['specimen']['structure']['name'])
print("section xy resolution: %f um" % data[0]['section_images'][0]['resolution'])
gene symbol: MBP
treatment name: ISH
specimen location: Cingulate Cortex
section xy resolution: 1.008000 um
Не углубляясь в модель данных API, SectionDataSets иметь составляющую SectionImages, Treatments, Probesи источник Specimens, Probes цель Genes, а также Specimens может быть связан с Structure, Запрос загружает всю эту информацию для одного SectionDataSet во вложенный словарь.
Я не помню, как найти протяженность блока образца. Я обновлю ответ, если найду его.