Как получить порядок узлов филогенетического дерева в R?
Я использовал пакет ggtree R для построения филогенетического дерева, и мне нужно знать порядок советов, чтобы я мог комбинировать его с конкретной информацией для каждого наконечника. Конечно, я могу вручную записать заказ, но у меня много таких деревьев. Более того, tr $ tip.label дает мне другой порядок узлов по сравнению с порядком отображения на древовидном графике.
Мне нужны некоторые R-скрипты, которые могут получить правильный порядок подсказок от объекта филогенетического дерева, который должен быть того же порядка, когда R строит дерево.
Дерево: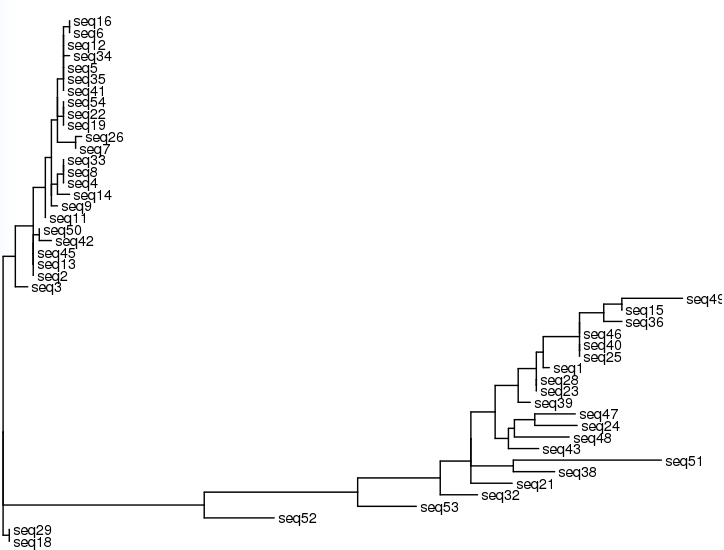
tr$tip.label дает:
"seq6" "seq16" "seq12" "seq34" "seq5" "seq35" "seq41" "seq19" "seq22" "seq54" "seq7" "seq26" "seq9" "seq14" "seq4" "seq8" "" seq11 "" seq2 "" seq13 "" seq45 "" seq42 "" seq50 "" seq3 "" seq18 "" seq29 "" seq52 "" seq53 "" seq32 "" seq21 "" seq38 "" seq51 "" seq24 "" seq47 "" seq48 "" seq43 "" seq39 "" seq23 "" seq28 "" seq1 "" seq25 "" seq40 "" seq46 "" seq36 "" seq15 "" seq49 "
Что я хочу: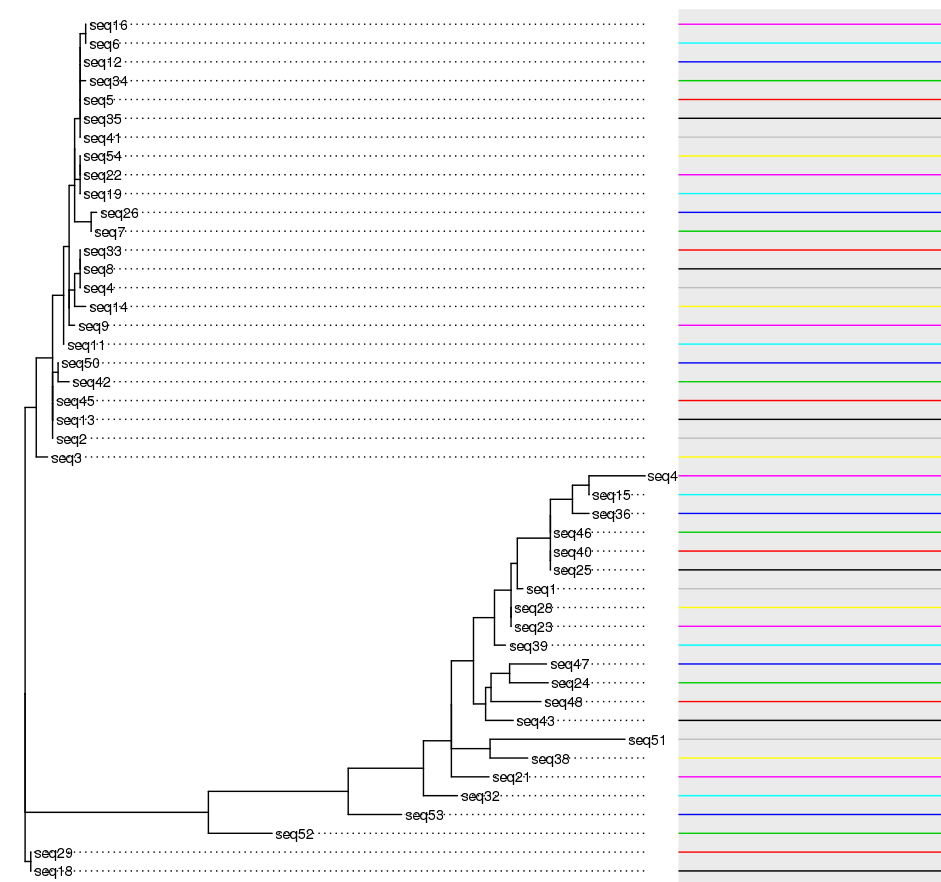
Каждая вершина дерева выравнивается по линии определенного цвета.
1 ответ
> library(ggtree)
> set.seed(123)
> tr = rtree(15)
> d=fortify(tr)
> dd = subset(d, isTip)
> dd$label[order(dd$y, decreasing=TRUE)]
[1] "t5" "t8" "t4" "t10" "t12" "t1" "t2" "t3" "t11" "t13" "t9" "t6"
[13] "t14" "t7" "t15"
Вот пример получения меток наконечников, соответствующих их позициям.